Genoma

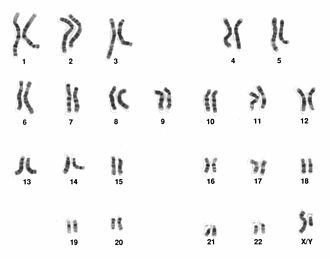
Em biologia, o genoma é toda a informação hereditária de um organismo que está codificada em seu DNA (ou, no caso de alguns vírus, no RNA). Isso inclui tanto genes como regiões intergénicas, DNA mitocondrial e DNA plastídico. Os genes consistem tanto na sua região codificante (exons) quanto a região não codificante, conhecida como íntrons ou junk DNA,[Nota 1] termo criado, em 1920, por Hans Winkler, professor de botânica na Universidade de Hamburgo, e que não é mais usado, porque atualmente se sabe que essas sequências não codificadoras são muito importantes para a regulação gênica, dentre outras funções.[1] Mais precisamente, o genoma de um organismo é uma sequência de DNA completa de um conjunto de cromossomos; por exemplo, um dos dois conjuntos que um indivíduo diploide contém em cada uma das suas células somáticas. Quando se diz que o genoma de uma espécie que se reproduz sexualmente foi "sequenciado", normalmente está a referir-se à determinação das sequências de um conjunto de autossomos e de um de cada tipo de cromossomo sexual, que determinam o sexo. Mesmo em espécies cujos indivíduos são todos do mesmo sexo, o que é descrito como "uma sequência genómica" pode ser um composto de cromossomos de vários indivíduos.
Em português corrente, a expressão constituição genética pode ser usada para designar o genoma de um dado indivíduo ou organismo. O estudo das propriedades globais dos genomas de organismos relacionados chama-se geralmente genómica,[2] termo que distingue essa disciplina da anatomia, que em geral se preocupa com o estudo das propriedades de genes únicos ou de grupos de genes.
- 1) É o conjunto simples de cromossomos de uma célula (cariótipo). É o conjunto formado por apenas um cromossomo de cada tipo, na espécie estudada. No ser humano o genoma é constituído por 23 pares de cromossomas.
- 2) Projeto genoma, denominação dada a tarefa de decodificação do DNA humano aceita por diversas nações associadas.[3]
Visão geral
[editar | editar código-fonte]Alguns organismos possuem múltiplas cópias de cromossomos: diploides, triploides, tetraploides e assim por diante. Na genética clássica, em um organismo reproduzindo sexualmente (tipicamente eukarya), o gameta tem metade do número de cromossomos da célula somática e o genoma é um conjunto completo de cromossomos em uma célula diploide. A redução à metade do material genético em gametas é realizada pela segregação de cromossomos homólogos durante a meiose. Em organismos haploides, incluindo células de bactérias, arqueas e organelas, incluindo mitocôndrias e cloroplastos, ou vírus, que contêm genes semelhantes, o único ou conjunto de cadeias circulares ou lineares de DNA (ou RNA para alguns vírus), também constituem o genoma . O termo genoma pode ser aplicado especificamente para significar o que é armazenado em um conjunto completo de DNA nuclear (ou seja, o "genoma nuclear"), mas também pode ser aplicado ao que é armazenado dentro de organelas que contêm seu próprio DNA, como com o "mitocondrial Genoma "ou o" genoma do cloroplasto ". Além disso, o genoma pode compreender elementos genéticos não cromossômicos, como vírus, plasmídeos e elementos transponíveis.
Normalmente, quando se diz que o genoma de uma espécie reproduzindo sexualmente foi "sequenciado", ele se refere a uma determinação das seqüências de um conjunto de autossomos e um de cada tipo de cromossomo sexual, que juntos representam ambos os sexos possíveis . Mesmo em espécies que existem em apenas um sexo, o que é descrito como uma "sequência do genoma" pode ser uma leitura composta dos cromossomos de vários indivíduos. De forma coloquial, a frase "maquiagem genética" às vezes é usada para significar o genoma de um determinado indivíduo ou organismo. O estudo das propriedades globais dos genomas de organismos relacionados geralmente é referido como genômica, o que a distingue da genética que Geralmente estuda as propriedades de genes ou grupos de genes únicos.[4]
Mapeamento e Sequência
[editar | editar código-fonte]Em 1976, Walter Fiers na Universidade de Ghent (Bélgica) foi o primeiro a estabelecer a sequência de nucleotídeos completa de um genoma de ARN viral (Bacteriophage MS2). No ano seguinte, Fred Sanger completou a primeira sequência de DNA-genoma: Phage
O desenvolvimento de novas tecnologias tornou-se dramaticamente mais fácil e mais barato fazer sequenciação, e o número de sequências completas do genoma está crescendo rapidamente. Os Institutos Nacionais de Saúde dos EUA mantêm uma das várias bases de dados abrangentes de informações genômicas.[6] Entre os milhares de projetos completos de sequenciamento do genoma, incluem-se o arroz, o mouse, a planta Arabidopsis thaliana, o peixe-soprador e a bactéria E. coli. Em dezembro de 2013, os cientistas primeiro sequenciaram todo o genoma de um Neanderthal, uma espécie extinta de humanos. O genoma foi extraído do osso dos pés de um Neanderthal de 130 mil anos encontrado em uma caverna da Sibéria.[7][8]

As novas tecnologias de sequenciamento, como aquencial paralela maciça, também abriram a perspectiva de sequenciamento pessoal do genoma como ferramenta de diagnóstico, como pioneira pela Manteia Predictive Medicine. Um passo importante em direção a esse objetivo foi a conclusão em 2007 do genoma completo de James D. Watson, um dos codescobridores da estrutura do DNA.[9]
Enquanto uma sequência do genoma lista a ordem de cada base de DNA em um genoma, um mapa do genoma identifica os pontos de referência. Um mapa de genoma é menos detalhado do que uma sequência de genoma e ajuda a navegar pelo genoma. O Projeto Genoma Humano foi organizado para mapear e sequenciar o genoma humano. Um passo fundamental no projeto foi o lançamento de um mapa genômico detalhado de Jean Weissenbach e sua equipe no Genoscope em Paris.[10][11]
As sequências e mapas do genoma de referência continuam a ser atualizados, removendo erros e esclarecendo regiões de alta complexidade alelica.[12] O custo decrescente do mapeamento genômico permitiu que os sites genealógicos a ofereçam como serviço,[13] na medida em que se pode submeter seu genoma a engajar pesquisas científicas, tais como o DNA. land no New York Genome Center, um exemplo tanto de
Tamanhos de genomas
[editar | editar código-fonte]Tabela comparativa de tamanhos de genomas (em pares de bases):
| Tipo | Organismo | Tamanho
(pares de base) |
Total de genes | |
|---|---|---|---|---|
| Vírus | Porcine circovirus type 1 | 1.759 | 1,8 kB | |
| Vírus | Bacteriophage MS2 [14] | 3.569 | 3,5 kB | |
| Vírus | SV40 | 5.224 | 5,2 kB | |
| Vírus | Phage |
5.386 | 5,4 kB | |
| Vírus | HIV | 9.749 | 9,7 kB | |
| Vírus | Phage |
48.502 | 48,5 kB | |
| Vírus | Megavirus | 1.259.197 | 1,3 MB | |
| Vírus | Pandoravirus salinus | 2.470.000 | 2,47 MB | |
| Organela Eucariota | Human mitochondrion | 16.569 | 16,6 kB | |
| Bactéria | Nasuia deltocephalinicola (strain NAS-ALF) | 112.091 | 112 kB | 137 |
| Bactéria | Carsonella ruddii | 159.662 | 160 kB | |
| Bactéria | Buchnera aphidicola | 600.000 | 600 kB | |
| Bactéria | Wigglesworthia glossinidia | 700.000 | 700Kb | |
| Bactéria – Cianobactéria | Prochlorococcus spp. (1.7 Mb) | 1.700.000 | 1,7 MB | 1.884 |
| Bactéria | Haemophilus influenzae | 1.830.000 | 1,8 MB | |
| Bactéria | Escherichia coli [16] | 4.600.000 | 4,6 MB | 4.288 |
| Bactéria – Cianobactéria | Nostoc punctiforme | 9.000.000 | 9 MB | 7.432 |
| Bactéria | Solibacter usitatus (strain Ellin 6076) | 9.970.000 | 10 MB | |
| Planta | Genlisea tuberosa | 61.000.000 | 61 MB | |
| Planta | Arabidopsis thaliana | 135.000.000 | 135 MB | 27.655 |
| Planta | Populus trichocarpa | 480.000.000 | 480 MB | 73.013 |
| Planta | Fritillaria assyriaca | 130.000.000.000 | 130 GB | |
| Planta | Paris japonica [17] | 150.000.000.000 | 150 GB | |
| Planta | Physcomitrella patens | 480.000.000 | 480 MB | |
| Fungo - levedura | Saccharomyces cerevisiae | 12.100.000 | 12,1 MB | 6.294 |
| Fungo | Aspergillus nidulans | 30.000.000 | 30 MB | 9.541 |
| Nematode | Pratylenchus coffeae | 20.000.000 | 20 MB | |
| Nematode | Caenorhabditis elegans | 100.300.000 | 100 MB | 19.000 |
| Inseto | Drosophila melanogaster [18] | 175.000.000 | 175 MB | 13.600 |
| Inseto | Apis mellifera (honey bee) | 236.000.000 | 236 MB | 10.157 |
| Inseto | Bombyx mori (silk moth) | 432.000.000 | 432 MB | 14.623 |
| Inseto | Solenopsis invicta (fire ant) | 480.000.000 | 480 MB | 16.569 |
| Mamífero | Mus musculus [19] | 2.700.000.000 | 2,7 GB | 20.210 |
| Mamífero | Pan paniscus | 3.286.640.000 | 3,3 GB | 20.000 |
| Mamífero | Homo sapiens [20] | 3.117.000.000 | 3,1 GB | 20.000 |
| Pássaro | Gallus gallus | 1.043.000.000 | 1,0 GB | 20.000 |
| Peixe | Tetraodon nigroviridis | 385.000.000 | 390 MB | |
| Peixe | Protopterus aethiopicus | 130.000.000.000 | 130 GB | |
Ver também
[editar | editar código-fonte]Notas
- ↑ Embora as regiões não codificantes tenham sido chamadas de Junk DNA, uma vez que não são expressas, pesquisadores descobriram que muitas delas regulam a expressão de outras seções do DNA. Ver Cohen, Jon (2010). Almost Chimpanzee. Searching for What Makes us Human, in Rainforests, Labs, Sanctuaries, and Zoos (em inglês). New York: Times Books. p. 24–26. 369 páginas. ISBN 978-0-8050-8307-1
Referências
- ↑ Kevin Davies, Decifrando o Genoma: a corrida para desvendar o DNA Humano ; Companhia das Letras, 2001, ISBN 8-535-90106-X
- ↑ Gibson, Greg; Muse, Spencer V (2004). A Primer of Genome Science (em inglês) 2ª ed. Sunderland, Massachusetts: Sinauer. p. 1. 378 páginas. ISBN 0-87893-232-1
- ↑ Valdomiro José de Souza, Projeto genoma humano; Edicoes Loyola, 2004, ISBN 8-515-02877-8
- ↑ «Genome». Wikipedia (em inglês). 25 de junho de 2017
- ↑ «All about genes». www.beowulf.org.uk (em inglês). Consultado em 17 de agosto de 2017
- ↑ «Home - Genome - NCBI». www.ncbi.nlm.nih.gov. Consultado em 17 de agosto de 2017
- ↑ Zimmer, Carl (18 de dezembro de 2013). «Toe Fossil Provides Complete Neanderthal Genome». The New York Times (em inglês). ISSN 0362-4331
- ↑ Prüfer, Kay; Racimo, Fernando; Patterson, Nick; Jay, Flora; Sankararaman, Sriram; Sawyer, Susanna; Heinze, Anja; Renaud, Gabriel; Sudmant, Peter H. «The complete genome sequence of a Neanderthal from the Altai Mountains». Nature. 505 (7481): 43–49. doi:10.1038/nature12886
- ↑ Wade, Nicholas (31 de maio de 2007). «Genome of DNA Pioneer Is Deciphered». The New York Times (em inglês). ISSN 0362-4331
- ↑ «What's a Genome?». www.genomenewsnetwork.org. Consultado em 17 de agosto de 2017
- ↑ «Mapping Factsheet». 19 de julho de 2010. Consultado em 17 de agosto de 2017
- ↑ «Help - Genome Reference Consortium». www.ncbi.nlm.nih.gov. Consultado em 17 de agosto de 2017
- ↑ https://www.facebook.com/sarah.kaplan.31. «How do your 20,000 genes determine so many wildly different traits? They multitask.». Washington Post. Consultado em 17 de agosto de 2017
- ↑ Fiers, W.; Contreras, R.; Duerinck, F.; Haegeman, G.; Iserentant, D.; Merregaert, J.; Min Jou, W.; Molemans, F.; Raeymaekers, A. (8 de abril de 1976). «Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene». Nature (em inglês) (5551): 500–507. ISSN 0028-0836. doi:10.1038/260500a0. Consultado em 14 de setembro de 2022
- ↑ Sanger, F.; Air, G. M.; Barrell, B. G.; Brown, N. L.; Coulson, A. R.; Fiddes, J. C.; Hutchison, C. A.; Slocombe, P. M.; Smith, M. (fevereiro de 1977). «Nucleotide sequence of bacteriophage
φ X174 DNA». Nature (em inglês) (5596): 687–695. ISSN 0028-0836. doi:10.1038/265687a0. Consultado em 14 de setembro de 2022 - ↑ Blattner, Frederick R.; Plunkett, Guy; Bloch, Craig A.; Perna, Nicole T.; Burland, Valerie; Riley, Monica; Collado-Vides, Julio; Glasner, Jeremy D.; Rode, Christopher K. (5 de setembro de 1997). «The Complete Genome Sequence of Escherichia coli K-12». Science (em inglês) (5331): 1453–1462. ISSN 0036-8075. doi:10.1126/science.277.5331.1453. Consultado em 14 de setembro de 2022
- ↑ Pellicer, Jaume; Fay, Michael F.; Leitch, Ilia J. (15 de setembro de 2010). «The largest eukaryotic genome of them all?: THE LARGEST EUKARYOTIC GENOME?». Botanical Journal of the Linnean Society (em inglês) (1): 10–15. doi:10.1111/j.1095-8339.2010.01072.x. Consultado em 14 de setembro de 2022
- ↑ Ellis, Lisa L.; Huang, Wen; Quinn, Andrew M.; Ahuja, Astha; Alfrejd, Ben; Gomez, Francisco E.; Hjelmen, Carl E.; Moore, Kristi L.; Mackay, Trudy F. C. (24 de julho de 2014). Copenhaver, Gregory P., ed. «Intrapopulation Genome Size Variation in D. melanogaster Reflects Life History Variation and Plasticity». PLoS Genetics (em inglês) (7): e1004522. ISSN 1553-7404. doi:10.1371/journal.pgen.1004522. Consultado em 14 de setembro de 2022
- ↑ Church, Deanna M.; Goodstadt, Leo; Hillier, LaDeana W.; Zody, Michael C.; Goldstein, Steve; She, Xinwe; Bult, Carol J.; Agarwala, Richa; Cherry, Joshua L. (26 de maio de 2009). Roberts, Richard J., ed. «Lineage-Specific Biology Revealed by a Finished Genome Assembly of the Mouse». PLoS Biology (em inglês) (5): e1000112. ISSN 1545-7885. PMC PMC2680341
 Verifique
Verifique |pmc=(ajuda). PMID 19468303. doi:10.1371/journal.pbio.1000112. Consultado em 14 de setembro de 2022 - ↑ Venter, J. Craig; Adams, Mark D.; Myers, Eugene W.; Li, Peter W.; Mural, Richard J.; Sutton, Granger G.; Smith, Hamilton O.; Yandell, Mark; Evans, Cheryl A. (16 de fevereiro de 2001). «The Sequence of the Human Genome». Science (em inglês) (5507): 1304–1351. ISSN 0036-8075. doi:10.1126/science.1058040. Consultado em 14 de setembro de 2022
