アイソフォーム
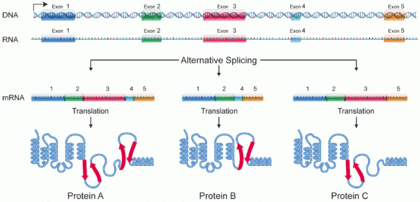
さまざまなアイソフォーム
概説
α サブユニットは触媒 サブユニットで、2つのアイソフォームが存在 する。α 1とα 2はそれぞれPRKAA1、PRKAA2遺伝子 にコードされている。β サブユニットは調節 サブユニットで、2つのアイソフォームが存在 する。β 1とβ 2はそれぞれPRKAB1、PRKAB2遺伝子 にコードされている。γ サブユニットは調節 サブユニットで、3つのアイソフォームが存在 する。γ 1、γ 2、γ 3はそれぞれPRKAG1、PRKAG2、PRKAG3遺伝子 にコードされている。
ヒトの
機構
スプライシングは
特徴
関連 する概念
グリコフォーム
グリコフォームもしくはグライコフォーム(glycoform)は、
例
- アクチン:
保存 されたタンパク質 であるにもかかわらず、さまざまな(哺乳類 では少 なくとも6種類 の)アイソフォームが存在 する。 - クレアチンキナーゼ:
血 中 に存在 し、心筋梗塞 の診断 に利用 される。3つのアイソフォームが存在 する。 - ヒアルロン
酸 合成 酵素 : ヒアルロン酸 の合成 を担 う。哺乳類 細胞 では3つのアイソフォームが存在 する。 - グルクロン
酸 転移 酵素 :多 くの薬剤 や環境 汚染 物質 、有毒 な内在 化合 物 の解毒 を担 う酵素 のスーパーファミリー。ヒトゲノムは16種類 のアイソフォームがコードされていることが知 られている[16]。 - モノアミン
酸化 酵素 : モノアミンの酸化 を触媒 する酵素 のファミリーで、MAO-AとMAO-Bの2つのアイソフォームが存在 する。
出典
- ^ “Alternative splicing and genome complexity”. Nature Genetics 30 (1): 29–30. (January 2002). doi:10.1038/ng803. PMID 11743582.
- ^ Kozlowski, L.; Orlowski, J.; Bujnicki, J. M. (2012). “Structure Prediction for Alternatively Spliced Proteins”. Alternative pre-mRNA Splicing. pp. 582. doi:10.1002/9783527636778.ch54. ISBN 9783527636778
- ^ a b “Generation of protein isoform diversity by alternative splicing: mechanistic and biological implications”. Annual Review of Cell Biology 3 (1): 207–42. (1987-01-01). doi:10.1146/annurev.cb.03.110187.001231. PMID 2891362.
- ^ a b c “Alternative splicing: a ubiquitous mechanism for the generation of multiple protein isoforms from single genes”. Annual Review of Biochemistry 56 (1): 467–95. (1987-01-01). doi:10.1146/annurev.bi.56.070187.002343. PMID 3304142.
- ^ “On the Dependency of Cellular Protein Levels on mRNA Abundance”. Cell 165 (3): 535–50. (April 2016). doi:10.1016/j.cell.2016.03.014. PMID 27104977.
- ^ a b “Evolving Lessons on the Complex Role of AMPK in Normal Physiology and Cancer” (English). Trends in Pharmacological Sciences 37 (3): 192–206. (March 2016). doi:10.1016/j.tips.2015.11.007. PMC 4764394. PMID 26711141.
- ^ a b “Alternative splicing: a pivotal step between eukaryotic transcription and translation” (
英語 ). Nature Reviews. Molecular Cell Biology 14 (3): 153–65. (March 2013). doi:10.1038/nrm3525. PMID 23385723. - ^ “Mechanisms and Regulation of Alternative Pre-mRNA Splicing”. Annual Review of Biochemistry 84 (1): 291–323. (2015-01-01). doi:10.1146/annurev-biochem-060614-034316. PMC 4526142. PMID 25784052.
- ^ a b “Alternative Splicing May Not Be the Key to Proteome Complexity”. Trends in Biochemical Sciences 42 (2): 98–110. (February 2017). doi:10.1016/j.tibs.2016.08.008. PMID 27712956.
- ^ “Genomic variation. Impact of regulatory variation from RNA to protein”. Science 347 (6222): 664–7. (February 2015). doi:10.1126/science.1260793. PMC 4507520. PMID 25657249.
- ^ “Noisy splicing drives mRNA isoform diversity in human cells”. PLoS Genetics 6 (12): e1001236. (December 2010). doi:10.1371/journal.pgen.1001236. PMC 3000347. PMID 21151575.
- ^ “Proteoform: a single term describing protein complexity” (
英語 ). Nature Methods 10 (3): 186–7. (March 2013). doi:10.1038/nmeth.2369. PMC 4114032. PMID 23443629. - ^ “Revisiting the identification of canonical splice isoforms through integration of functional genomics and proteomics evidence”. Proteomics 14 (23-24): 2709–18. (December 2014). doi:10.1002/pmic.201400170. PMC 4372202. PMID 25265570.
- ^ “Cell death or survival promoted by alternative isoforms of ErbB4”. Molecular Biology of the Cell 21 (23): 4275–86. (December 2010). doi:10.1091/mbc.E10-04-0332. PMC 2993754. PMID 20943952.
- ^ “Widespread Expansion of Protein Interaction Capabilities by Alternative Splicing”. Cell 164 (4): 805–17. (February 2016). doi:10.1016/j.cell.2016.01.029. PMC 4882190. PMID 26871637.
- ^ “Substrate specificity of the human UDP-glucuronosyltransferase UGT2B4 and UGT2B7. Identification of a critical aromatic amino acid residue at position 33”. The FEBS Journal 274 (5): 1256–64. (March 2007). doi:10.1111/j.1742-4658.2007.05670.x. PMID 17263731.