ERCC4 (excision repair cross-complementation group 4)もしくはXPF (xeroderma pigmentosum complementation group F)は、ヒトではERCC4 遺伝子 いでんし タンパク質 たんぱくしつ ERCC1 とともにERCC1-XPF複 ふく 合体 がったい 形成 けいせい DNA修復 しゅうふく やDNA組 ぐみ 換 か に関与 かんよ [ 5] [ 6]
ERCC1-XPFはDNAの特定 とくてい 構造 こうぞう 切断 せつだん ヌクレアーゼ である。ERCC1とERCC4はDNA修復 しゅうふく 時 じ 因子 いんし 多 おお 面 めん 関 かん 共 とも 記載 きさい 複 ふく 合体 がったい ヌクレオチド除去 じょきょ 修復 しゅうふく 経路 けいろ 必須 ひっす ヌクレアーゼ である。ERCC1-XPFヌクレアーゼはDNA二 に 本 ほん 鎖 くさり 切断 せつだん 修復 しゅうふく 本 ほん 鎖 くさり 連結 れんけつ 有害 ゆうがい 架橋 かきょう 損傷 そんしょう 修復 しゅうふく 経路 けいろ 機能 きのう
ERCC4 に不 ふ 活 かつ 化 か 変異 へんい 有 ゆう 細胞 さいぼう 紫外線 しがいせん 照射 しょうしゃ 鎖 くさり 間 あいだ 架橋 かきょう 行 おこな 化学 かがく 物質 ぶっしつ 特定 とくてい 損傷 そんしょう 因子 いんし 対 たい 感受性 かんじゅせい 正常 せいじょう 細胞 さいぼう 高 たか Ercc4 に不 ふ 活 かつ 化 か 変異 へんい 有 ゆう 遺伝子 いでんし 改変 かいへん 修復 しゅうふく 欠陥 けっかん 代謝 たいしゃ 誘発 ゆうはつ 性 せい 生理 せいり 的 てき 変化 へんか 伴 ともな 早老 そうろう 引 ひ 起 お [ 7] Ercc4 を完全 かんぜん ホモ接合 せつごう 型 がた で)欠 かけ 失 しっ 生存 せいぞん ERCC4 のホモ接合 せつごう 型 がた 欠 かけ 失 しつ 見 み 集団 しゅうだん 稀 まれ 機能 きのう 不全 ふぜん 遺伝 いでん 的 てき 変異 へんい 有 ゆう 個体 こたい 正常 せいじょう 遺伝子 いでんし 存在 そんざい 場合 ばあい 変異 へんい 色 いろ 素性 すじょう 乾 いぬい 皮 かわ 症 しょう コケイン症候群 しょうこうぐん やファンコニ貧血 ひんけつ などの疾患 しっかん 原因 げんいん 場合 ばあい
ERCC4 遺伝子 いでんし 導入 どうにゅう CHO細胞 さいぼう 由来 ゆらい 特定 とくてい 紫外線 しがいせん 感受性 かんじゅせい 変異 へんい 株 かぶ 修復 しゅうふく 欠陥 けっかん 修正 しゅうせい [ 8] 細胞 さいぼう 複数 ふくすう 独立 どくりつ 相補 そうほ 群 ぐん 単 たん 離 はな [ 9] 遺伝子 いでんし 細胞 さいぼう 紫外線 しがいせん 耐 たい 性 せい 回復 かいふく 種 たね 間 あいだ 遺伝 いでん 的 てき 相補 そうほ 性 せい 反映 はんえい 遺伝子 いでんし 命名 めいめい [ 10]
ヒトのERCC4 遺伝子 いでんし アミノ酸 あみのさん 約 やく タンパク質 たんぱくしつ [ 11]
ERCC4 と同等 どうとう 機能 きのう 有 ゆう 類似 るいじ 遺伝子 いでんし 他 た 真 ま 核 かく 生物 せいぶつ 存在 そんざい 最 もっと 研究 けんきゅう 遺伝子 いでんし 出芽 しゅつが 酵母 こうぼ Saccharomyces cerevisiae のRAD1 遺伝子 いでんし 分裂 ぶんれつ 酵母 こうぼ Schizosaccharomyces pombe のrad16+ 遺伝子 いでんし [ 11]
XPFの模 も 式 しき 図 ず 不 ふ 活性 かっせい 示 しめ 1分子 ぶんし 分子 ぶんし 結合 けつごう 活性 かっせい 型 がた 二 に 量 りょう 体 たい 形成 けいせい 二 に 量 りょう 体 たい タンパク質 たんぱくしつ 間 あいだ 相互 そうご 作用 さよう タンパク質 たんぱくしつ タンパク質 たんぱくしつ 間 あいだ 相互 そうご 作用 さよう 媒介 ばいかい エンドヌクレアーゼ 活性 かっせい 部位 ぶい 持 も 結合 けつごう タンパク質 たんぱくしつ タンパク質 たんぱくしつ 間 あいだ 相互 そうご 作用 さよう 関与 かんよ [ 8]
ERCC4/XPFには2つの保存 ほぞん 存在 そんざい 間 あいだ 低 てい 保存 ほぞん 性 せい 領域 りょういき 隔 へだ N末端 まったん 領域 りょういき DNAヘリカーゼ スーパーファミリーIIの保存 ほぞん 相 あい 同性 どうせい [ 12] C末端 まったん 領域 りょういき 活性 かっせい 活性 かっせい 部位 ぶい 残 ざん 基 もと 位置 いち [ 13] 大 だい 部分 ぶぶん 末端 まったん 領域 りょういき 配列 はいれつ 関連 かんれん [ 14] 残 ざん 基 もと 存在 そんざい 末端 まったん 結合 けつごう 存在 そんざい
ERCC1-XPFヌクレアーゼの基質 きしつ 構造 こうぞう ERCC1-XPF複 ふく 合体 がったい 構造 こうぞう 特異 とくい 的 てき 一本 いっぽん 鎖 くさり 二 に 本 ほん 鎖 くさり 対 たい 切断 せつだん 活性 かっせい 持 も 二 に 本 ほん 鎖 くさり 一本 いっぽん 鎖 くさり 接合 せつごう 部 ぶ 骨格 こっかく 特異 とくい 的 てき 切断 せつだん 切断 せつだん 導入 どうにゅう 接合 せつごう 部 ぶ 側 がわ 約 やく 離 はな 位置 いち 二 に 本 ほん 鎖 くさり [ 15] 構造 こうぞう 特異 とくい 性 せい 酵母 こうぼ 初 はじ 示 しめ [ 16]
ERCC1とXPFのC末端 まったん 領域 りょういき 位置 いち 疎水 そすい 的 てき 互 たが 相互 そうご 作用 さよう 両者 りょうしゃ 二 に 量 りょう 体 からだ 化 か 促進 そくしん [ 17] [ 18] 二 に 量 りょう 体 からだ 化 か 場合 ばあい 触媒 しょくばい 活性 かっせい 示 しめ 触媒 しょくばい 内 ない 存在 そんざい 自体 じたい 触媒 しょくばい 活性 かっせい 持 も 複 ふく 合体 がったい 活性 かっせい 必要 ひつよう 不可欠 ふかけつ
ERCC1-XPFのDNAへの結合 けつごう 関 かん 関連 かんれん タンパク質 たんぱくしつ 断片 だんぺん 原子 げんし 分解能 ぶんかいのう 構造 こうぞう 基 もと 提唱 ていしょう [ 17] 結合 けつごう 媒介 ばいかい 二 に 量 りょう 体 たい 接合 せつごう 部 ぶ 配置 はいち 考 かんが
ヌクレオチド除去 じょきょ 修復 しゅうふく 時 じ タンパク質 たんぱくしつ 複 ふく 合体 がったい 協調 きょうちょう 的 てき 損傷 そんしょう 認識 にんしき 損傷 そんしょう 部位 ぶい 両側 りょうがわ 短 みじか 範囲 はんい 局所 きょくしょ 的 てき 開 ひら 損傷 そんしょう 鎖 くさり 損傷 そんしょう 部位 ぶい 側 がわ 切 き 込 こ 入 い [ 15] 過程 かてい XPA (英語 えいご 版 ばん 相互 そうご 作用 さよう タンパク質 たんぱくしつ 協調 きょうちょう 的 てき 相互 そうご 作用 さよう 行 おこな
ERCC1-XPFに変異 へんい 有 ゆう 哺乳類 ほにゅうるい 細胞 さいぼう 正常 せいじょう 細胞 さいぼう 比較 ひかく 二 に 本 ほん 鎖 くさり 切断 せつだん 引 ひ 起 お 因子 いんし 電離 でんり 放射線 ほうしゃせん 対 たい 感受性 かんじゅせい 程度 ていど 高 たか [ 19] [ 20] 相 あい 同 どう 組 くみ 換 か 修復 しゅうふく 非 ひ 相 あい 同 どう 末端 まったん 結合 けつごう 双方 そうほう 機能 きのう 依存 いぞん [ 21] [ 22] 二 に 本 ほん 鎖 くさり 切断 せつだん 修復 しゅうふく 経路 けいろ 活性 かっせい 再 さい 結合 けつごう 前 まえ 末 まつ 端 はし 非 ひ 相 あい 同 どう 一本 いっぽん 鎖 くさり 除去 じょきょ 関係 かんけい 相 あい 同 どう 組 くみ 換 か 活性 かっせい 一本 いっぽん 鎖 くさり 経路 けいろ 必要 ひつよう 末端 まったん 一本 いっぽん 鎖 くさり 非 ひ 相 あい 同 どう 末端 まったん 結合 けつごう 経路 けいろ 必要 ひつよう 経路 けいろ Kuタンパク質 たんぱくしつ の活性 かっせい 依存 いぞん [ 23] [ 24] 相 あい 同 どう 組 くみ 換 か 利用 りよう 組 く 込 こ 遺伝子 いでんし 操作 そうさ 重要 じゅうよう 技術 ぎじゅつ 細胞 さいぼう 機能 きのう 依存 いぞん [ 25]
ERCC1もしくはXPFに変異 へんい 有 ゆう 哺乳類 ほにゅうるい 細胞 さいぼう 鎖 くさり 間 あいだ 架橋 かきょう 引 ひ 起 お 因子 いんし 対 たい 感受性 かんじゅせい 特 とく 高 たか [ 26] 鎖 くさり 間 あいだ 架橋 かきょう 複製 ふくせい 進行 しんこう 遮断 しゃだん 遮断 しゃだん 複製 ふくせい 部分 ぶぶん 構造 こうぞう 切断 せつだん 基質 きしつ [ 27] [ 28] 一方 いっぽう 鎖 くさり 架橋 かきょう 両側 りょうがわ 切 き 込 こ 入 い 架橋 かきょう 外 はず 修復 しゅうふく 開始 かいし 可能 かのう 性 せい 他 た 鎖 くさり 間 あいだ 架橋 かきょう 付近 ふきん 二 に 本 ほん 鎖 くさり 切断 せつだん 導入 どうにゅう 後 ご 相 あい 同 どう 組 くみ 換 か 修復 しゅうふく 関与 かんよ 可能 かのう 性 せい 鎖 くさり 間 あいだ 架橋 かきょう 修復 しゅうふく 関与 かんよ 唯一 ゆいいつ 細胞 さいぼう 周期 しゅうき 段階 だんかい 鎖 くさり 間 あいだ 架橋 かきょう 修復 しゅうふく 必要 ひつよう [ 29] [ 30]
希少 きしょう 遺伝 いでん 疾患 しっかん 色 いろ 素性 すじょう 乾 いぬい 皮 かわ 症 しょう 患者 かんじゃ 一部 いちぶ ERCC4 に変異 へんい 抱 かか 患者 かんじゃ 分類 ぶんるい 診断 しんだん 基準 きじゅん 特徴 とくちょう 乾燥 かんそう 鱗状 りんじょう 皮膚 ひふ 露光 ろこう 部 ぶ 皮膚 ひふ 異常 いじょう 色素 しきそ 沈着 ちんちゃく 重度 じゅうど 光 ひかり 感受性 かんじゅせい 紫外線 しがいせん 皮膚 ひふ 発症 はっしょう 倍 ばい 以上 いじょう 高 たか [ 5]
XP-Fの患者 かんじゃ 大 だい 部分 ぶぶん 症状 しょうじょう 中等 ちゅうとう 度 ど 中 なか 割合 わりあい コケイン症候群 しょうこうぐん (CS)の症状 しょうじょう [ 31] 症候群 しょうこうぐん 患者 かんじゃ 光 ひかり 感受性 かんじゅせい 示 しめ 発生 はっせい 欠陥 けっかん 神経症 しんけいしょう 状 じょう 示 しめ [ 5] [ 7]
ERCC4 遺伝子 いでんし 変異 へんい 非常 ひじょう 稀 まれ 早老 そうろう 症候群 しょうこうぐん 引 ひ 起 お 場合 ばあい [ 32] 患者 かんじゃ 特徴 とくちょう 加 くわ 神経 しんけい 肝胆 かんたん 道 どう 筋骨 きんこつ 格 かく 造血 ぞうけつ 系 けい 症状 しょうじょう
ファンコニ貧血 ひんけつ (FA)の症状 しょうじょう 示 しめ 患者 かんじゃ 中 なか ERCC4 遺伝子 いでんし 原因 げんいん 変異 へんい 存在 そんざい 場合 ばあい 貧血 ひんけつ 主 おも 造血 ぞうけつ 系 けい 症状 しょうじょう 伴 ともな 複 ふく 合 あい 的 てき 疾患 しっかん 特徴 とくちょう 鎖 くさり 間 あいだ 架橋 かきょう 引 ひ 起 お 因子 いんし 対 たい 過敏 かびん 性 せい ERCC4 遺伝子 いでんし 変異 へんい 有 ゆう 患者 かんじゃ 分類 ぶんるい [ 31] [ 33]
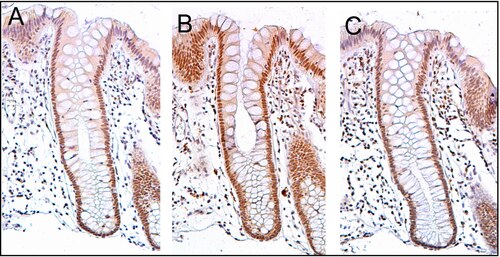
免疫 めんえき 染色 せんしょく 行 おこな 結腸 けっちょう 陰 かげ 連続 れんぞく 断面 だんめん 修復 しゅうふく タンパク質 たんぱくしつ 正常 せいじょう 高 こう 発現 はつげん 示 しめ 陰 かげ 結腸 けっちょう 新 しん 生物 せいぶつ 発生 はっせい 歳 さい 男性 だんせい 生 なま 検 けん 試料 しりょう 陰 かげ 大 だい 部分 ぶぶん 吸収 きゅうしゅう 細胞 さいぼう 核 かく 内 ない タンパク質 たんぱくしつ 高 こう 発現 はつげん 陰 かげ 頂 いただき 上部 じょうぶ 陰 かげ 内 うち 面 めん 細胞 さいぼう 核 かく 発現 はつげん 低下 ていか [ 34] 通常 つうじょう 結腸 けっちょう 内壁 ないへき 細胞 さいぼう 細胞 さいぼう 核 かく 高 こう 発現 はつげん 結腸 けっちょう 内壁 ないへき 陥 おちい 入 いれ 伴 ともな 単 たん 層 そう 円柱 えんちゅう 上皮 じょうひ 覆 おお 陥 おちい 入部 にゅうぶ 腸 ちょう 腺 せん 腸 ちょう 陰 かげ 呼 よ 結腸 けっちょう 陰 かげ 厚 あつ 壁 かべ 試験管 しけんかん 微視的 びしてき 形状 けいじょう 管 かん 長 なが 方向 ほうこう 中心 ちゅうしん 穴 あな 開 ひら 陰 かげ 深 ふか 細胞 さいぼう 個 こ 個 こ 分 ぶん 程度 ていど 正常 せいじょう 非 ひ 腫瘍 しゅよう 性 せい 結腸 けっちょう 上皮 じょうひ 修復 しゅうふく 非常 ひじょう 活発 かっぱつ PMS2 、ERCC1の高 こう 発現 はつげん 伴 ともな
細胞 さいぼう 陰 かげ 底部 ていぶ 生 う 出 だ 陰 かげ 軸 じく 沿 そ 上方 かみがた 移動 いどう 数日 すうじつ 後 ご 結腸 けっちょう 管 かん 脱落 だつらく [ 35] 陰 かげ 底部 ていぶ 個 こ 個 こ 幹 みき 細胞 さいぼう 存在 そんざい 底部 ていぶ 幹 みき 細胞 さいぼう 発現 はつげん 場合 ばあい 一般 いっぱん 的 てき 陰 かげ 数 すう 千 せん 細胞 さいぼう 全 すべ 発現 はつげん 節 ふし 図 ず 陰 かげ 全 すべ 腸 ちょう 細胞 さいぼう 対 たい 免疫 めんえき 染色 せんしょく 褐色 かっしょく 染色 せんしょく 示 しめ 正常 せいじょう 結腸 けっちょう 上皮 じょうひ 各 かく 陰 かげ 数 すう 千 せん 腸 ちょう 細胞 さいぼう 同様 どうよう 発現 はつげん 図 ず ヘマトキシリン によるDNAの対比 たいひ 染色 せんしょく 行 おこな 核 かく 青 あお 灰色 はいいろ 示 しめ 粘膜 ねんまく 固有 こゆう 層 そう 細胞 さいぼう 陰 かげ 下部 かぶ 周囲 しゅうい 細胞 さいぼう 核 かく 大 だい 部分 ぶぶん 青 あお 灰色 はいいろ 呈 てい 発現 はつげん 陰 かげ 頂 いただき 上部 じょうぶ 位置 いち 細胞 さいぼう 発現 はつげん 低下 ていか 同様 どうよう 青 あお 灰色 はいいろ 呈 てい [ 34]
結腸 けっちょう 隣接 りんせつ 領域 りょういき 欠損 けっそん [ 編集 へんしゅう ] 大腸 だいちょう 近傍 きんぼう 結腸 けっちょう 上皮 じょうひ 連続 れんぞく 断面 だんめん 結腸 けっちょう 陰 かげ 発現 はつげん 低下 ていか 消失 しょうしつ 示 しめ 組織 そしき 断片 だんぺん 状 じょう 結腸 けっちょう 腺腫 せんしゅ 発生 はっせい 男性 だんせい 患者 かんじゃ 切除 せつじょ 結腸 けっちょう 中 なか 組織 そしき 学 がく 的 てき 正常 せいじょう 領域 りょういき 由来 ゆらい 関 かん 陰 かげ 本体 ほんたい 入口 いりくち 部分 ぶぶん 隣接 りんせつ 管 かん 面 めん 全 すべ 上皮 じょうひ 細胞 さいぼう 細胞 さいぼう 核 かく 発現 はつげん 関 かん 陰 かげ 細胞 さいぼう 核 かく 大 だい 部分 ぶぶん 発現 はつげん 低下 ていか 入口 いりくち 部分 ぶぶん 隣接 りんせつ 管 かん 面 めん 高 こう 発現 はつげん 関 かん 陰 かげ 管 かん 面 めん 細胞 さいぼう 核 かく 大 だい 部分 ぶぶん 発現 はつげん 一部 いちぶ 陰 かげ 入口 いりくち 部分 ぶぶん 発現 はつげん 検出 けんしゅつ 組織 そしき 修復 しゅうふく 遺伝子 いでんし 発現 はつげん 低下 ていか 喪失 そうしつ 抑制 よくせい [ 34] 大腸 だいちょう 約 やく 周囲 しゅうい 以内 いない 発 はつ 可能 かのう 性 せい 高 たか 発 はつ 素地 そじ 位置 いち 結腸 けっちょう 陰 かげ 約 やく 発現 はつげん 欠損 けっそん [ 34] 発 はつ 素地 そじ 減少 げんしょう 場合 ばあい 修復 しゅうふく 酵素 こうそ 発現 はつげん 同様 どうよう 低下 ていか 多 おお 結腸 けっちょう 上皮 じょうひ 欠乏 けつぼう エピジェネティック な抑制 よくせい [ 34] 欠乏 けつぼう 損傷 そんしょう 修復 しゅうふく 減少 げんしょう 考 かんが 多 おお 種類 しゅるい 根底 こんてい 損傷 そんしょう 対 たい 適切 てきせつ 応答 おうとう 修復 しゅうふく 行 おこな 能力 のうりょく 欠如 けつじょ [ 36] 周囲 しゅうい 発 はつ 素地 そじ 抑制 よくせい 抑制 よくせい 変化 へんか 多 おお 結腸 けっちょう 進行 しんこう 中心 ちゅうしん 的 てき 役割 やくわり 果 は 可能 かのう 性 せい 示 しめ
ERCC4の発現 はつげん 低下 ていか 結腸 けっちょう 高 こう 頻度 ひんど 変異 へんい 生 しょう 稀 まれ [ 37] 変異 へんい 皮膚 ひふ 高 たか [ 37] ERCC4 の遺伝 いでん 的 てき 多 た 型 かた 乳 にゅう 重要 じゅうよう [ 38] 稀 まれ 変異 へんい 欠乏 けつぼう 進行 しんこう 関与 かんよ 可能 かのう 性 せい 高 たか 強調 きょうちょう
^ a b c GRCh38: Ensembl release 89: ENSG00000175595 - Ensembl , May 2017
^ a b c GRCm38: Ensembl release 89: ENSMUSG00000022545 - Ensembl , May 2017
^ Human PubMed Reference: ^ Mouse PubMed Reference: ^ a b c Friedberg, EC; Walker, GC; Siede, W; Wood, RD; Schultz, RA; Ellenberger, T (2006). DNA Repair and Mutagenesis . ASM Press. ISBN 978-1555813192
^ “Entrez Gene: ERCC4 excision repair cross-complementing rodent repair deficiency, complementation group 4 ”. 2023年 ねん 月 がつ 日 にち 閲覧 えつらん ^ a b “Physiological consequences of defects in ERCC1-XPF DNA repair endonuclease” . DNA Repair 10 (7): 781–91. (Jul 2011). doi :10.1016/j.dnarep.2011.04.026 . PMC 3139823 . PMID 21612988 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3139823/ .
^ a b “Molecular cloning of a human DNA repair gene”. Nature 310 (5976): 425–9. (1984). Bibcode : 1984Natur.310..425W . doi :10.1038/310425a0 . PMID 6462228 .
^ “Summary of complementation groups of UV-sensitive CHO cell mutants isolated by large-scale screening”. Mutagenesis 4 (5): 349–54. (Sep 1989). doi :10.1093/mutage/4.5.349 . PMID 2687628 . ^ “ERCC4 (XPF) encodes a human nucleotide excision repair protein with eukaryotic recombination homologs” . Molecular and Cellular Biology 16 (11): 6553–62. (Nov 1996). doi :10.1128/mcb.16.11.6553 . PMC 231657 . PMID 8887684 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC231657/ . ^ a b Manandhar, Mandira; Boulware, Karen S.; Wood, Richard D. (2015-09-15). “The ERCC1 and ERCC4 (XPF) genes and gene products” . Gene 569 (2): 153–161. doi :10.1016/j.gene.2015.06.026 . ISSN 1879-0038 . PMC 4536074 . PMID 26074087 . https://pubmed.ncbi.nlm.nih.gov/26074087 .
^ “A relationship between a DNA-repair/recombination nuclease family and archaeal helicases”. Trends in Biochemical Sciences 24 (3): 95–7. (Mar 1999). doi :10.1016/s0968-0004(99)01355-9 . PMID 10203755 . ^ “The active site of the DNA repair endonuclease XPF-ERCC1 forms a highly conserved nuclease motif” . The EMBO Journal 21 (8): 2045–53. (Apr 2002). doi :10.1093/emboj/21.8.2045 . PMC 125967 . PMID 11953324 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC125967/ . ^ “Activity of individual ERCC1 and XPF subunits in DNA nucleotide excision repair” . Nucleic Acids Research 29 (4): 872–9. (Feb 2001). doi :10.1093/nar/29.4.872 . PMC 29621 . PMID 11160918 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC29621/ . ^ a b “Xeroderma pigmentosum group F caused by a defect in a structure-specific DNA repair endonuclease”. Cell 86 (5): 811–22. (Sep 1996). doi :10.1016/s0092-8674(00)80155-5 . hdl :1765/3110 PMID 8797827 .
^ “Specific cleavage of model recombination and repair intermediates by the yeast Rad1-Rad10 DNA endonuclease”. Science 265 (5181): 2082–5. (Sep 1994). Bibcode : 1994Sci...265.2082B . doi :10.1126/science.8091230 . PMID 8091230 . ^ a b “The structure of the human ERCC1/XPF interaction domains reveals a complementary role for the two proteins in nucleotide excision repair.”. Structure 13 (12): 1849–1858. (December 2005). doi :10.1016/j.str.2005.08.014 . hdl :1874/14818 ISSN 1633-8413 . PMID 16338413 .
^ “The Cerebro-oculo-facio-skeletal Syndrome Point Mutation F231L in the ERCC1 DNA Repair Protein Causes Dissociation of the ERCC1-XPF Complex.” . J Biol Chem 290 (33): 20541–20555. (August 2015). doi :10.1074/jbc.M114.635169 . ISSN 1083-351X . PMC 4536458 . PMID 26085086 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4536458/ . ^ “Radiation-induced lethality and mutation in a repair-deficient CHO cell line”. International Journal of Radiation Biology and Related Studies in Physics, Chemistry, and Medicine 43 (2): 207–13. (Feb 1983). doi :10.1080/09553008314550241 . PMID 6600735 . ^ “ERCC1-XPF endonuclease facilitates DNA double-strand break repair” . Molecular and Cellular Biology 28 (16): 5082–92. (Aug 2008). doi :10.1128/MCB.00293-08 . PMC 2519706 . PMID 18541667 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2519706/ . ^ “Recombination-dependent deletion formation in mammalian cells deficient in the nucleotide excision repair gene ERCC1” . Proceedings of the National Academy of Sciences of the United States of America 94 (24): 13122–7. (Nov 1997). Bibcode : 1997PNAS...9413122S . doi :10.1073/pnas.94.24.13122 . PMC 24273 . PMID 9371810 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC24273/ . ^ “The ERCC1/XPF endonuclease is required for efficient single-strand annealing and gene conversion in mammalian cells” . Nucleic Acids Research 36 (1): 1–9. (Jan 2008). doi :10.1093/nar/gkm888 . PMC 2248766 . PMID 17962301 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2248766/ . ^ “Alternative-NHEJ is a mechanistically distinct pathway of mammalian chromosome break repair” . PLOS Genetics 4 (6): e1000110. (Jun 2008). doi :10.1371/journal.pgen.1000110 . PMC 2430616 . PMID 18584027 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2430616/ . ^ “ERCC1-XPF endonuclease facilitates DNA double-strand break repair” . Molecular and Cellular Biology 28 (16): 5082–92. (Aug 2008). doi :10.1128/MCB.00293-08 . PMC 2519706 . PMID 18541667 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2519706/ . ^ “The structure-specific endonuclease Ercc1-Xpf is required for targeted gene replacement in embryonic stem cells” . The EMBO Journal 20 (22): 6540–9. (Nov 2001). doi :10.1093/emboj/20.22.6540 . PMC 125716 . PMID 11707424 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC125716/ . ^ “Mammalian nucleotide excision repair proteins and interstrand crosslink repair” . Environmental and Molecular Mutagenesis 51 (6): 520–6. (Jul 2010). doi :10.1002/em.20569 . PMC 3017513 . PMID 20658645 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3017513/ . ^ “Recombination-dependent deletion formation in mammalian cells deficient in the nucleotide excision repair gene ERCC1” . Proceedings of the National Academy of Sciences of the United States of America 94 (24): 13122–7. (Nov 1997). Bibcode : 1997PNAS...9413122S . doi :10.1073/pnas.94.24.13122 . PMC 24273 . PMID 9371810 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC24273/ . ^ “XPF-ERCC1 acts in Unhooking DNA interstrand crosslinks in cooperation with FANCD2 and FANCP/SLX4” . Molecular Cell 54 (3): 460–71. (May 2014). doi :10.1016/j.molcel.2014.03.015 . PMC 5067070 . PMID 24726325 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5067070/ . ^ “Multiple roles of ERCC1-XPF in mammalian interstrand crosslink repair”. Environmental and Molecular Mutagenesis 51 (6): 567–81. (Jul 2010). doi :10.1002/em.20583 . PMID 20658648 . ^ “Advances in understanding the complex mechanisms of DNA interstrand cross-link repair” . Cold Spring Harbor Perspectives in Biology 5 (10): a012732. (Oct 2013). doi :10.1101/cshperspect.a012732 . PMC 4123742 . PMID 24086043 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4123742/ . ^ a b “Malfunction of nuclease ERCC1-XPF results in diverse clinical manifestations and causes Cockayne syndrome, xeroderma pigmentosum, and Fanconi anemia” . American Journal of Human Genetics 92 (5): 807–19. (May 2013). doi :10.1016/j.ajhg.2013.04.007 . PMC 3644632 . PMID 23623389 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3644632/ .
^ “A new progeroid syndrome reveals that genotoxic stress suppresses the somatotroph axis”. Nature 444 (7122): 1038–43. (Dec 2006). Bibcode : 2006Natur.444.1038N . doi :10.1038/nature05456 . PMID 17183314 . ^ “Mutations in ERCC4, encoding the DNA-repair endonuclease XPF, cause Fanconi anemia” . American Journal of Human Genetics 92 (5): 800–6. (May 2013). doi :10.1016/j.ajhg.2013.04.002 . PMC 3644630 . PMID 23623386 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3644630/ . ^ a b c d e “Deficient expression of DNA repair enzymes in early progression to sporadic colon cancer” . Genome Integr 3 (1): 3. (2012). doi :10.1186/2041-9414-3-3 . PMC 3351028 . PMID 22494821 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3351028/ .
^ “Quantification of crypt and stem cell evolution in the normal and neoplastic human colon” . Cell Rep 8 (4): 940–7. (2014). doi :10.1016/j.celrep.2014.07.019 . PMC 4471679 . PMID 25127143 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4471679/ . ^ “The DNA damage response: ten years after”. Mol. Cell 28 (5): 739–45. (2007). doi :10.1016/j.molcel.2007.11.015 . PMID 18082599 . ^ a b “Physiological consequences of defects in ERCC1-XPF DNA repair endonuclease” . DNA Repair (Amst.) 10 (7): 781–91. (2011). doi :10.1016/j.dnarep.2011.04.026 . PMC 3139823 . PMID 21612988 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3139823/ .
^ “Combined genetic and nutritional risk models of triple negative breast cancer”. Nutr Cancer 66 (6): 955–63. (2014). doi :10.1080/01635581.2014.932397 . PMID 25023197 .