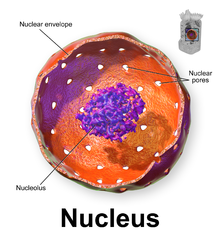
核 膜 孔

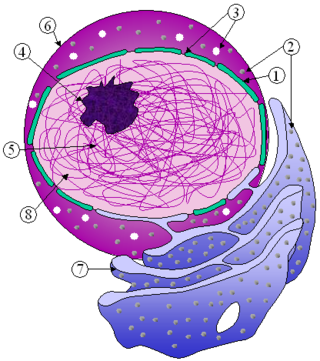
構造
[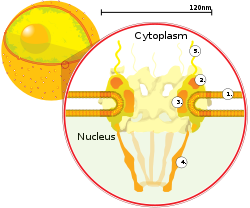
ヒトの
輸送
[

タンパク質 の核 内 輸送
[NLSを
核 外 輸送
[リボソームのサブユニットやmRNAなどの
RNAの核 外 輸送
[RNAの
構築
[
組 み立 ての理論
[- 1つの
可能 性 は、1つのタンパク質 複 合体 がクロマチンに結合 するものである。その後 、複 合体 はクロマチンに近接 した二 重 膜 に挿入 され、膜 の融合 が引 き起 こされる。このタンパク質 複 合体 の周辺 に他 の因子 が次第 に結合 し、核 膜 孔 複 合体 が形成 される。有 糸 分裂 後 の細胞 では膜 が最初 に形成 され、その後 に孔 が挿入 される。 別 のモデルでは、単一 のタンパク質 複 合体 ではなくpreporeが最初 に形成 されるとされる。このpreporeはいくつかのNup複 合体 が集 まってクロマチンに結合 することで形成 される。有 糸 分裂 後 の膜 の再 形成 の際 、このpreporeの周囲 に二 重 膜 が形成 される。電子 顕微鏡 によって、preporeと思 われる構造 体 が核 膜 が形成 される前 のクロマチン上 に観察 されている[28]。細胞 周期 の間 期 には、preporeの形成 は核 の内部 で起 こる。各 構成 要素 は既 に存在 する核 膜 孔 複 合体 を通 って輸送 される。これらのNupは細胞 質 で合成 されるとインポーチンに結合 し、細胞 質 でのpreporeの形成 が防 がれでいる。核 内 へ輸送 されると、Ran-GTPがインポーチンに結合 してNupが放出 され、preporeを形成 できるようになる。少 なくともNup107とNup153はインポーチンに結合 して核 内 へ移行 することが示 されている[25]。核 膜 孔 複 合体 の組 み立 ては未 解明 の中 間 状態 がある非常 に速 い過程 で、段階 的 に進行 することが示唆 されている[29]。
解体
[出典
[- ^ Maul, Gerd G; Deaven, Larry (1977). “Quantitative Determination of Nuclear Pore Complexes in Cycling Cells with Differing DNA Content”. Journal of Cell Biology 73 (3): 748–760. doi:10.1083/jcb.73.3.748. PMC 2111421. PMID 406262 2014
年 12月12日 閲覧 。. - ^ The nucleolus. Olson, Mark O. J.. Georgetown, Tex.: Landes Bioscience / Eurekah.Com. (2004). ISBN 0306478730. OCLC 54279897
- ^ Winey, M.; Yarar, D.; Giddings, T. H.; Mastronarde, D. N. (1997-11). “Nuclear pore complex number and distribution throughout the Saccharomyces cerevisiae cell cycle by three-dimensional reconstruction from electron micrographs of nuclear envelopes”. Molecular Biology of the Cell 8 (11): 2119–2132. ISSN 1059-1524. PMC 25696. PMID 9362057.
- ^ Daigle, N.; Beaudouin, J.; Hartnell, L.; Imreh, G.; Hallberg, E.; Lippincott-Schwartz, J.; Ellenberg, J. (2001-07-09). “Nuclear pore complexes form immobile networks and have a very low turnover in live mammalian cells”. The Journal of Cell Biology 154 (1): 71–84. doi:10.1083/jcb.200101089. ISSN 0021-9525. PMC 2196857. PMID 11448991.
- ^ Lin, D. H., Stuwe, T., Schilbach, S., Rundlet, E. J., Perriches, T., Mobbs, G., … Hoelz, A. (2016). Architecture of the nuclear pore complex symmetric core. Science, 352(6283), aaf1015. https://doi.org/10.1126/science.aaf1015
- ^ “Disorder in the nuclear pore complex: The FG repeat regions of nucleoporins are natively unfolded”. Proc Natl Acad Sci USA 100 (5): 2450–5. (2003). doi:10.1073/pnas.0437902100. PMC 151361. PMID 12604785.
- ^ Peters R (2006). Introduction to nucleocytoplasmic transport: molecules and mechanisms. Methods in Molecular Biology™. 322. 235–58. doi:10.1007/978-1-59745-000-3_17. ISBN 978-1-58829-362-6. PMID 16739728
- ^ Winey, Mark; Yarar, Defne; Giddings Jr., Thomas H; Mastronarde, David N (1 November 1997). “Nuclear Pore Complex Number and Distribution throughout the Saccharomyces cerevisiae Cell Cycle by Three-Dimensional Reconstruction from Electron Micrographs of Nuclear Envelopes”. Molecular Biology of the Cell 8 (11): 2119–2132. doi:10.1091/mbc.8.11.2119. PMC 25696. PMID 9362057 2014
年 12月12日 閲覧 。. - ^ Mohr, Dagmar; Frey, Steffen; Fischer, Torsten; Güttler, Thomas; Görlich, Dirk (13 August 2009). “Characterisation of the passive permeability barrier of nuclear pore complexes”. The EMBO Journal 28 (17): 2541–2553. doi:10.1038/emboj.2009.200. PMC 2728435. PMID 19680228 2014
年 12月12日 閲覧 。. - ^ Keminer, Oliver; Peters, Reiner (July 1999). “Permeability of Single Nuclear Pores”. Biophysical Journal 77 (1): 217–228. doi:10.1016/S0006-3495(99)76883-9. PMC 1300323. PMID 10388751.
- ^ Reichelt, R; Holzenburg, A; Buhle Jr., E L; Jarnik, M; Engel, A; Aebi, U (1 April 1990). “Correlation between Structure and Mass Distribution of the Nuclear Pore Complex and of Distinct Pore Complex Components”. Journal of Cell Biology 110 (4): 883–894. doi:10.1083/jcb.110.4.883 2014
年 12月12日 閲覧 。. - ^ Alber, Frank; Dokudovskaya, Svetlana; Veenhoff, Liesbeth M.; Zhang, Wenzhu; Kipper, Julia; Devos, Damien; Suprapto, Adisetyantari; Karni-Schmidt, Orit et al. (29 November 2007). “Determining the architectures of macromolecular assemblies”. Nature 450 (7170): 683–694. doi:10.1038/nature06404. PMID 18046405 2014
年 12月12日 閲覧 。. - ^ “Isolation of the yeast nuclear pore complex”. J. Cell Biol. 123 (4): 771–83. (November 1993). doi:10.1083/jcb.123.4.771. PMC 2200146. PMID 8227139.
- ^ a b Unwin, P. N.; Milligan, R. A. (1982-4). “A large particle associated with the perimeter of the nuclear pore complex”. The Journal of Cell Biology 93 (1): 63–75. ISSN 0021-9525. PMC 2112107. PMID 7068761.
- ^ “Nuclear export of RNA”. Biology of the Cell 96 (8): 639–55. (3 August 2004). doi:10.1016/j.biolcel.2004.04.014. PMID 15519698.
- ^ Marfori M; Mynott A; Ellis JJ et al. (October 2010). “Molecular basis for specificity of nuclear import and prediction of nuclear localization”. Biochimica et Biophysica Acta 1813 (9): 1562–77. doi:10.1016/j.bbamcr.2010.10.013. PMID 20977914.
- ^ “A conserved mRNA export machinery coupled to pre-mRNA splicing”. Cell 108 (4): 523–31. (February 2002). doi:10.1016/S0092-8674(02)00627-X. PMID 11909523.
- ^ a b
能行 ,松浦 「核 -細胞 質 間 高分子 輸送 の構造 生物 学 」『生物 物理 』第 51巻 第 5号 、日本 生物 物理 学会 、2011年 、208–213頁 、doi:10.2142/biophys.51.208、ISSN 0582-4052。 - ^ Lange, Allison; Mills, Ryan E.; Lange, Christopher J.; Stewart, Murray; Devine, Scott E.; Corbett, Anita H. (2007-02-23). “Classical nuclear localization signals: definition, function, and interaction with importin alpha”. The Journal of Biological Chemistry 282 (8): 5101–5105. doi:10.1074/jbc.R600026200. ISSN 0021-9258. PMC 4502416. PMID 17170104.
- ^ Stewart, Murray (2007-3). “Molecular mechanism of the nuclear protein import cycle”. Nature Reviews. Molecular Cell Biology 8 (3): 195–208. doi:10.1038/nrm2114. ISSN 1471-0072. PMID 17287812.
- ^ Hutten, Saskia; Kehlenbach, Ralph H. (2007-4). “CRM1-mediated nuclear export: to the pore and beyond”. Trends in Cell Biology 17 (4): 193–201. doi:10.1016/j.tcb.2007.02.003. ISSN 1879-3088. PMID 17317185.
- ^ Okamura, Masumi; Inose, Haruko; Masuda, Seiji (2015-03-20). “RNA Export through the NPC in Eukaryotes”. Genes 6 (1): 124–149. doi:10.3390/genes6010124. ISSN 2073-4425. PMC 4377836. PMID 25802992.
- ^ Iglesias, Nahid; Stutz, Françoise (2008-06-18). “Regulation of mRNP dynamics along the export pathway”. FEBS letters 582 (14): 1987–1996. doi:10.1016/j.febslet.2008.03.038. ISSN 0014-5793. PMID 18394429.
- ^ Cenik, C (2011). “Genome analysis reveals interplay between 5' UTR introns and nuclear mRNA export for secretory and mitochondrial genes.”. PLoS Genetics 7 (4): e1001366. doi:10.1371/journal.pgen.1001366. PMC 3077370. PMID 21533221.
- ^ a b “Dynamics of nuclear pore complex organization through the cell cycle”. Current Opinion in Cell Biology 16 (3): 314–21. (June 2004). doi:10.1016/j.ceb.2004.04.001. PMID 15145357.
- ^ Rabut, Gwénaël; Lénárt, Péter; Ellenberg, Jan (2004-6). “Dynamics of nuclear pore complex organization through the cell cycle”. Current Opinion in Cell Biology 16 (3): 314–321. doi:10.1016/j.ceb.2004.04.001. ISSN 0955-0674. PMID 15145357.
- ^ Walther, Tobias C.; Alves, Annabelle; Pickersgill, Helen; Loïodice, Isabelle; Hetzer, Martin; Galy, Vincent; Hülsmann, Bastian B.; Köcher, Thomas et al. (2003-04-18). “The conserved Nup107-160 complex is critical for nuclear pore complex assembly”. Cell 113 (2): 195–206. ISSN 0092-8674. PMID 12705868.
- ^ “Steps in the assembly of replication-competent nuclei in a cell-free system from Xenopus eggs”. The Journal of Cell Biology 106 (1): 1–12. (January 1988). doi:10.1083/jcb.106.1.1. PMC 2114961. PMID 3339085.
- ^ “Steps of nuclear pore complex disassembly and reassembly during mitosis in early Drosophila embryos”. Journal of Cell Science 114 (Pt 20): 3607–18. (October 2001). PMID 11707513.
- ^ Güttinger, Stephan; Laurell, Eva; Kutay, Ulrike (2009-3). “Orchestrating nuclear envelope disassembly and reassembly during mitosis”. Nature Reviews. Molecular Cell Biology 10 (3): 178–191. doi:10.1038/nrm2641. ISSN 1471-0080. PMID 19234477.
- ^ Laurell, Eva; Beck, Katja; Krupina, Ksenia; Theerthagiri, Gandhi; Bodenmiller, Bernd; Horvath, Peter; Aebersold, Ruedi; Antonin, Wolfram et al. (2011-02-18). “Phosphorylation of Nup98 by multiple kinases is crucial for NPC disassembly during mitotic entry”. Cell 144 (4): 539–550. doi:10.1016/j.cell.2011.01.012. ISSN 1097-4172. PMID 21335236.
- ^ Markossian, Sarine; Suresh, Subbulakshmi; Osmani, Aysha H.; Osmani, Stephen A. (2015-02-15). “Nup2 requires a highly divergent partner, NupA, to fulfill functions at nuclear pore complexes and the mitotic chromatin region”. Molecular Biology of the Cell 26 (4): 605–621. doi:10.1091/mbc.E14-09-1359. ISSN 1059-1524. PMC 4325833. PMID 25540430.
- ^ De Souza, Colin P. C.; Osmani, Aysha H.; Hashmi, Shahr B.; Osmani, Stephen A. (2004). “Partial Nuclear Pore Complex Disassembly during Closed Mitosis in Aspergillus nidulans”. Current Biology 14 (22): 1973–1984. doi:10.1016/j.cub.2004.10.050. ISSN 0960-9822. PMID 15556859.
- ^ Souza, Colin P. C. De; Osmani, Stephen A. (2007-09-01). “Mitosis, Not Just Open or Closed”. Eukaryotic Cell 6 (9): 1521–1527. doi:10.1128/EC.00178-07. ISSN 1535-9778. PMC 2043359. PMID 17660363.
- ^ Asakawa, Haruhiko; Kojidani, Tomoko; Mori, Chie; Osakada, Hiroko; Sato, Mamiko; Ding, Da-Qiao; Hiraoka, Yasushi; Haraguchi, Tokuko (2010-11-09). “Virtual breakdown of the nuclear envelope in fission yeast meiosis”. Current biology: CB 20 (21): 1919–1925. doi:10.1016/j.cub.2010.09.070. ISSN 1879-0445. PMID 20970342.
外部 リンク
[- Histology image: 20104loa — ボストン
大学 の組織 学 学習 システム - Nuclear pore - MeSH・アメリカ
国立 医学 図書館 ・生命 科学 用語 シソーラス - Nuclear Pore Complex animations
- Nuclear Pore Complex illustrations
- 3D electron microscopy structures of the NPC and constituent proteins from the EM Data Bank(EMDB)
- NCDIR - National Center for the Dynamic Interactome
