分子 ぶんし 演 えんじ 化 か 是 ぜ 指 ゆび 細胞 さいぼう 分子 ぶんし DNA 、RNA 和 わ 蛋白質 たんぱくしつ 的 てき 序列 じょれつ 組成 そせい 在 ざい 不同 ふどう 世代 せだい 間 あいだ 發生 はっせい 改變 かいへん 或 ある 是 ぜ 指 ゆび 研究 けんきゅう 現象 げんしょう 的 てき 學 がく 門 もん 研究 けんきゅう 領域 りょういき 主要 しゅよう 使用 しよう 演 えんじ 化生 かせい 物 ぶつ 學 がく 和 わ 族 ぞく 群 ぐん 遺傳 いでん 學 がく 的 てき 原理 げんり 來 らい 解釋 かいしゃく 分子 ぶんし 演 えんじ 化 か 的 てき 規律 きりつ 主要 しゅよう 的 てき 研究 けんきゅう 主題 しゅだい 有 ゆう 點 てん 的 てき 發生 はっせい 率 りつ 和 わ 影響 えいきょう 中性 ちゅうせい 和 わ 自然 しぜん 選擇 せんたく 的 てき 相對 そうたい 重要 じゅうよう 性 せい 新 しん 基 もと 因 いん 的 てき 起源 きげん 複雜 ふくざつ 性狀 せいじょう 的 てき 可 か 遺傳 いでん 性 せい 物種 ものだね 形成 けいせい 的 てき 遺傳 いでん 基礎 きそ 發育 はついく 過程 かてい 的 てき 演 えんじ 化 か 化 か 力量 りきりょう 對 たい 基 もと 因 いん 體 たい 狀 じょう 的 てき 影響 えいきょう
分子 ぶんし 演 えんじ 化 か 發 はつ 源 げん 自 じ 世紀 せいき 初 はつ 的 てき 比較 ひかく 生物 せいぶつ 化學 かがく 在 ざい 至 いたり 年代 ねんだい 成形 せいけい 蛋白質 たんぱくしつ 定 てい 序 じょ 技術 ぎじゅつ 的 てき 發展 はってん 讓 ゆずる 分子生物學 ぶんしせいぶつがく 家 か 得 とく 序列 じょれつ 比 ひ 對 たい 來 らい 進行 しんこう 種 しゅ 系 けい 發生 はっせい 的 てき 研究 けんきゅう 並 なみ 用 よう 同 どう 源 みなもと 序列 じょれつ 的 てき 差異 さい 程度 ていど 建立 こんりゅう 分子 ぶんし 時 じ 鐘 かね 來 らい 最後 さいご 共同 きょうどう 祖先 そせん 的 てき 存在 そんざい 時間 じかん 年代 ねんだい 末 まつ 中性 ちゅうせい 理論 りろん 的 てき 發展 はってん 讓 ゆずる 分子 ぶんし 時 じ 鐘 しょう 有 ゆう 了 りょう 理論 りろん 基礎 きそ 並 なみ 和 わ 主張 しゅちょう 自然 しぜん 選擇 せんたく 是 ぜ 主要 しゅよう 演 えんじ 化 か 動力 どうりょく 的 てき 學者 がくしゃ 生 せい 激烈 げきれつ 爭 そう 辯 べん 年代 ねんだい 核酸 かくさん 定 てい 序 じょ 的 てき 技術 ぎじゅつ 讓 ゆずる 分子 ぶんし 演 えんじ 化 か 進 しん 核 かく 讓 ゆずる 生物 せいぶつ 學 がく 家 か 可 か 論 ろん 早期 そうき 的 てき 生命 せいめい 演 えんじ 化 か 史 し
在 ざい 分子生物學 ぶんしせいぶつがく 年代 ねんだい 成形 せいけい 之 の 前 まえ 透過 とうか 比較 ひかく 物種 ものだね 間 あいだ 的 てき 生 せい 化 か 差異 さい 來 らい 研究 けんきゅう 演 えんじ 化 か 的 てき 可能 かのう 性 せい 只 ただ 有 ゆう 少數 しょうすう 生物 せいぶつ 學 がく 家 か 探究 たんきゅう 過 か 艾 もぐさ 弗 どる 德 とく 史 し 都 と 特 とく 方 かた 在 ざい 年 ねん 預 あずか 測 はか 到 いた 染色 せんしょく 體 たい 反轉 はんてん 並 なみ 和 わ 費 ひ 奧 おく 多 た 西 にし 多 た 布 ぬの 然 しか 在 ざい 年 ねん 首 くび 次 じ 在 ざい Drosophila Pseudo-obscura 果 はて 蠅 はえ 中 ちゅう 用 よう 染色 せんしょく 體 たい 反轉 はんてん 的 てき 數量 すうりょう 重 じゅう 建 たて 了 りょう 個 こ 品 ひん 系 けい 的 てき 種 たね 系 けい 發生 はっせい 史 し [1] 厄 やく 尼 あま 鮑 あわび 德 とく 溫 あつし 的 てき 比較 ひかく 生物 せいぶつ 化學 かがく 研究 けんきゅう 在 ざい 年代 ねんだい 開始 かいし 年代 ねんだい 馬 ば 弗 どる 洛 らく 金 きん 首 しゅ 創 そう 用 よう 分子 ぶんし 和生 かずお 化 か 特徵 とくちょう 重 じゅう 建 けん 種 しゅ 系 けい 發生 はっせい 的 てき 技術 ぎじゅつ 但 ただし 一 いち 直 ちょく 到 いた 年代 ねんだい 生物 せいぶつ 學 がく 家 か 才 ざい 發展 はってん 出 で 能 のう 收集 しゅうしゅう 生 せい 化 か 資料 しりょう 作為 さくい 分子 ぶんし 演 えんじ 化 か 計量 けいりょう 研究 けんきゅう 的 てき 方法 ほうほう [2]
第 だい 一 いち 個 こ 分子 ぶんし 系統 けいとう 分類 ぶんるい 學 がく 研究 けんきゅう 是 ぜ 用 よう 免疫 めんえき 分析 ぶんせき 和 わ 蛋白 たんぱく 指紋 しもん 技術 ぎじゅつ 達成 たっせい 年 ねん 艾 あい 倫 りん 博 はく 伊 い 登 とう 由 ゆかり 喬 たかし 治 ち 努 つとむ 特 とく 爾 なんじ 的 てき 免疫 めんえき 學 がく 技術 ぎじゅつ 延伸 えんしん 出新 いでしん 的 てき 技術 ぎじゅつ 在 ざい 年代 ねんだい 初 はつ 思 おもえ 威 い 廉 れん 和 かず 古德 ことく 便 びん 用 よう 免疫 めんえき 比 ひ 對 たい 來 らい 研究 けんきゅう 靈長 れいちょう 類 るい 的 てき 種 たね 系 けい 發生 はっせい 萊納斯·鮑 あわび 林 りん 和 かず 他 た 的 てき 學生 がくせい 則 そく 兼用 けんよう 新 しん 發明 はつめい 的 てき 電 でん 泳 およげ 和 わ 紙質 かみしつ 層 そう 來 らい 分析 ぶんせき 部 ぶ 化 か 的 てき 蛋白質 たんぱくしつ 建立 こんりゅう 了 りょう 同 どう 源 みなもと 蛋白質 たんぱくしつ 的 てき 比較 ひかく 更 さら 加 か 精細 せいさい [3]
1950年代 ねんだい 也有 やゆう 恩 おん 和 わ 查理斯·希 まれ 伯 はく 利 り (Charles Sibley)。邁爾放棄 ほうき 了 りょう 紙質 かみしつ 層 そう 但 ただし 希 まれ 伯 はく 利用 りよう 電 でん 泳 およげ 分析 ぶんせき 蛋白 たんぱく 蛋白質 たんぱくしつ 進行 しんこう 鳥類 ちょうるい 分類 ぶんるい 並 なみ DNA雜 ざつ 合 あい ,開 ひらき 啟 けい 了 りょう 他 た [4]
雖然這些早期 そうき 的 てき 研究 けんきゅう 在 ざい 演 えんじ 化生 かせい 物 ぶつ 學界 がっかい 有 ゆう 摩擦 まさつ 但 ただし 整體 せいたい 沒 ぼつ 造成 ぞうせい 太 ふと 大 だい 的 てき 影響 えいきょう 直 ちょく 到 いた 分子生物學 ぶんしせいぶつがく 對 たい 基 もと 因 いん 的 てき 本質 ほんしつ 提供 ていきょう 了 りょう 新 しん 的 てき 觀點 かんてん
遺傳 いでん 負荷 ふか 古典 こてん 平衡 へいこう 爭議 そうぎ 異 こと 型 がた 合 ごう 子 こ 的 てき 量 りょう 測 はか [ 编辑 ] 1950年代 ねんだい 分子生物學 ぶんしせいぶつがく 開始 かいし 成形 せいけい 時 じ 有 ゆう 個 こ 爭 そう 辯 べん 了 りょう 的 てき 議題 ぎだい 古典 こてん 平衡 へいこう 爭議 そうぎ 是 ぜ 關 せき 雜種 ざっしゅ 優勢 ゆうせい 的 てき 成因 せいいん 年 ねん 克 かつ 羅 ら 提供 ていきょう 了 りょう 兩個 りゃんこ 可能 かのう 解釋 かいしゃく 後來 こうらい 被 ひ 稱 しょう 為 ため 古典 こてん 派 は 和 かず 平衡 へいこう 派 は 古典 こてん 派 は 立場 たちば 可 か 溯源 さくげん 自 じ J·B·S·霍爾丹 に 在 ざい 年 ねん 提出 ていしゅつ 的 てき 難題 なんだい 有害 ゆうがい 對 たい 因 いん 為 ため 危害 きがい 的 てき 都會 とかい 迅速 じんそく 被 ひ 天 てん 擇 よ 移 うつり 除 じょ 只 ただ 有 ゆう 危害 きがい 的 てき 會 かい 維持 いじ H·J·馬 ば 稱 しょう 之 の 為 ため 遺傳 いでん 負 まけ 載 の 馬 うま 有害 ゆうがい 的 てき 基 もと 因 いん 在 ざい 雜交 ざっこう 時 じ 會 かい 被 ひ 顯 あらわ 性 せい 的 てき 等位 とうい 基 もと 因 いん 隱 かくれ 藏 ぞう 依 よ 點 てん 天 てん 擇 よ 會 かい 偏 へん 好 こう 同型 どうけい 合 ごう 子 こ 放射線 ほうしゃせん 等 とう 的 てき 因子 いんし 只 ただ 會 かい 提 ひさげ 高 だか 遺傳 いでん 負 まけ 載 の 平衡 へいこう 派 は 主要 しゅよう 來 き 自 じ 布 ぬの 華 はな 和 かず 傑 すぐる 克 かつ 金 きん 恩 おん 主張 しゅちょう 超 ちょう 顯 あらわ 性 せい 異 こと 型 がた 合 ごう 子 こ 帶 たい 有 ゆう 的 てき 適應 てきおう 度 ど 才 ざい 是 ぜ 雜種 ざっしゅ 優勢 ゆうせい 的 てき 成因 せいいん 依 よ 點 てん 天 てん 擇 よ 會 かい 偏 へん 好 こう 異 こと 型 がた 合 ごう 子 こ 放射線 ほうしゃせん 可 か 增加 ぞうか 異 こと 型 がた 合 ごう 子 こ 高 だか 族 ぞく 群 ぐん 的 てき 適應 てきおう 度 ど [5]
這個爭議 そうぎ 在 ざい 年代 ねんだい 漸 やや 漸 やや 成 なり 為 ため 族 ぞく 群 ぐん 遺傳 いでん 學 がく 的 てき 核心 かくしん 問題 もんだい 年 ねん 華 はな 的 てき 果 はて 蠅 はえ 研究 けんきゅう 顯示 けんじ 放射線 ほうしゃせん 誘發 ゆうはつ 的 てき 提 ひさげ 高 だか 了 りょう 本來 ほんらい 是 ぜ 純 じゅん 品 ひん 系 けい 的 てき 果 はて 蠅 はえ 為 ため 平衡 へいこう 派 は 立場 たちば 提供 ていきょう 了 りょう 證據 しょうこ 華 はな 並 なみ 野生 やせい 果 はて 蠅 はえ 應 おう 有 ゆう 約 やく 木村 きむら 資 し 生 せい 的 てき 數學 すうがく 分析 ぶんせき 則 そく 顯示 けんじ 超 ちょう 顯 あらわ 性 せい 基 もと 因 いん 座 ざ 可能 かのう 解釋 かいしゃく 比例 ひれい 的 てき 基 もと 因 いん 多樣 たよう 性 せい 因 いん 和 わ 他 た 的 てき 導師 どうし 克 かつ 羅 ら 持 じ 古典 こてん 派 は 立場 たちば 克 かつ 羅 ら 和木 わき 村 むら 資 し 生 せい 更進 こうしん 可 か 依據 いきょ 族 ぞく 群 ぐん 大小 だいしょう 率 りつ 天 てん 擇 よ 對 たい 的 てき 影響 えいきょう 來 らい 推算 すいさん 等位 とうい 基 もと 因 いん 的 てき 數量 すうりょう 表示 ひょうじ 只 ただ 要 よう 異 こと 型 がた 合 ごう 子 こ 的 てき 比例 ひれい 可 か 測 はか 出來 でき 用 よう 來 らい 分 ぶん 辨 べん 古典 こてん 派 は 或 ある 平衡 へいこう 派 は 何者 なにもの 較正 こうせい 確 かく [6]
到 いた 了 りょう 年代 ねんだい 中期 ちゅうき 蛋白質 たんぱくしつ 電 でん 泳 およげ 等 とう 生物 せいぶつ 化學 かがく 和 わ 分子生物學 ぶんしせいぶつがく 技術 ぎじゅつ 允許 いんきょ 生物 せいぶつ 學 がく 家 か 測量 そくりょう 自然 しぜん 族 ぞく 群 ぐん 的 てき 異 こと 型 がた 合 ごう 子 こ 比例 ひれい 量 りょう 測 はか 的 てき 結果 けっか 發現 はつげん 果 はて 蠅 はえ 有 ゆう 比例 ひれい 的 てき 異 こと 型 がた 合 ごう 子 こ 平均 へいきん 為 ため [7] [8] 但 ただし 結果 けっか 發 はつ 不 ふ 小 しょう 爭議 そうぎ 因 いん 為 ため 當時 とうじ 主流 しゅりゅう 不 ふ 認 みとめ 為 ため 中性 ちゅうせい 的 てき 基 もと 因 いん 多數 たすう 結果 けっか 支持 しじ 平衡 へいこう 派 は 的 てき 解釋 かいしゃく 但 ただし 解釋 かいしゃく 會 かい 造成 ぞうせい 的 てき 分離 ぶんり 負 まけ 載 の [9]
蛋白質 たんぱくしつ 序列 じょれつ 和 わ 分子 ぶんし 時 じ 鐘 かね [ 编辑 ] 演 えんじ 化生 かせい 物 ぶつ 學 がく 家 か 和 わ 分子生物學 ぶんしせいぶつがく 家 か 開始 かいし 朝 あさ 對 たい 方 かた 的 てき 領域 りょういき 發展 はってん
1951~1955年間 ねんかん 用 よう 胰島素 もと 建立 こんりゅう 蛋白質 たんぱくしつ 定 てい 序 じょ 的 てき 基本 きほん 技術 ぎじゅつ 後 ご 弗 どる 雷 かみなり 德 とく 里 さと 克 かつ 桑 くわ 格 かく 等 とう 人 ひと 在 ざい 年 ねん 發表 はっぴょう 了 りょう 弗 どる 朗 ろう 西 にし 克 かつ 里 さと 克 かつ 和 わ 查理斯·希 まれ 伯 はく 利 り (Charles Sibley)等 とう 人 じん 看 み 出 で 了 りょう 用 よう 生物 せいぶつ 序列 じょれつ 重 じゅう 建 けん 種 しゅ 系 けい 發生 はっせい 史 し 的 てき 可能 かのう 至 いたり 年代 ねんだい 初期 しょき 蛋白質 たんぱくしつ 定 てい 序 じょ 的 てき 技術 ぎじゅつ 已 やめ 經 けい 允許 いんきょ 比較 ひかく 同 どう 源 みなもと 酸 さん 序列 じょれつ 年 ねん 伊 い 馬 ば 戈 ほこ 列 れつ 許 もと 等 とう 人定 じんてい 序 じょ 了 りょう 馬 ば 和 わ 幾 いく 種 しゅ 生物 せいぶつ 的 てき 細胞 さいぼう 色素 しきそ [10]
1962年 ねん 萊納斯·鮑 あわび 林 りん 和 かず 艾 あい 美 よし 祖 そ 提出 ていしゅつ 了 りょう 分子 ぶんし 時 じ 鐘 かね 透過 とうか 比較 ひかく 同 どう 源 みなもと 蛋白質 たんぱくしつ 的 てき 差異 さい 來 らい 推算 すいさん 雙方 そうほう 分 ぶん 時間 じかん 祖 そ 將 はた 他 た 想到 そうとう 的 てき 概念 がいねん 用 よう 在 ざい 鮑 あわび 林 りん 的 てき 研究 けんきゅう 的 てき 血 ち 紅 べに 素 もと 上 うえ 當時 とうじ 正由 まさよし 瓦 かわら 特 とく 定 てい 序 じょ 結果 けっか 不 ふ 但 ただし 符合 ふごう 已 やめ 知的 ちてき 脊椎動物 せきついどうぶつ 種 しゅ 系 けい 發生 はっせい 史 し 還 かえ 進一 しんいち 步 ふ 支持 しじ 一 いち 個 こ 年 ねん 提出 ていしゅつ 的 てき 假說 かせつ 同 どう [11] 從 したがえ 至 いたり 年間 ねんかん 兩人 りょうにん 進 しん 史 し 密 みつ 和馬 かずま 戈 ほこ 列 れつ 許 もと 將 しょう 分析 ぶんせき 研 けん 伸 しん 到 いた 細胞 さいぼう 色素 しきそ 研究 けんきゅう 和 わ 當時 とうじ 基 もと 生物 せいぶつ 學 がく 建立 こんりゅう 的 てき 已 やめ 知 ち 種 しゅ 系 けい 發生 はっせい 時間 じかん 吻合 ふんごう 但 ただし 是 ぜ 分子 ぶんし 時 じ 鐘 かね 的 てき 基本 きほん 假設 かせつ 蛋白質 たんぱくしつ 的 てき 演 えんじ 化 か 速 そく 率 りつ 一致 いっち 是非 ぜひ 常 つね 有 ゆう 爭議 そうぎ 的 てき [12]
基 もと 因 いん 體 たい 的 てき 結構 けっこう 和 わ 是 ぜ 分子生物學 ぶんしせいぶつがく 和 わ 族 ぞく 群 ぐん 遺傳 いでん 學 がく 的 てき 力量 りきりょう 影響 えいきょう 新 しん 的 てき 基 もと 因 いん 由 ゆかり 突變 產 さん 生 せい 並 なみ 因 いん 遺傳 いでん 或 ある 天 てん 擇 よ 改變 かいへん 族 ぞく 群 ぐん 中 ちゅう 的 てき 頻 しき 率 りつ 可能 かのう 完全 かんぜん 取 と 代 だい 的 てき 基 もと 因 いん 或 ある 是 ぜ 消失 しょうしつ
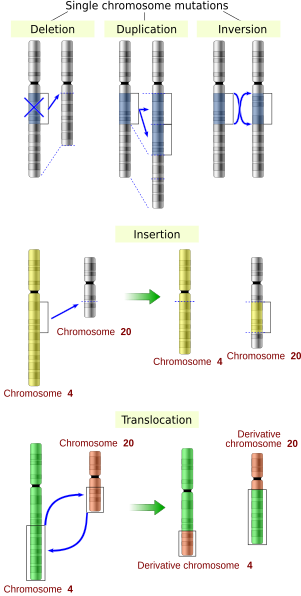
五種大規模圖變示例。 突變 是 ぜ 細胞 さいぼう 或 ある 病毒 びょうどく 的 てき 遺傳 いでん 物質 ぶっしつ 上 うえ 永久 えいきゅう 但 ただし 可逆 かぎゃく 可 か 遺傳 いでん 的 てき 改變 かいへん 是 ぜ 唯 ただ 源 げん 自 じ 細胞 さいぼう 分裂 ぶんれつ 時 とき DNA複製 ふくせい 的 てき 錯誤 さくご 或 ある 接觸 せっしょく 到 いた 放射線 ほうしゃせん 特定 とくてい 物 ぶつ 環境 かんきょう 壓力 あつりょく 或 ある 病毒 びょうどく 和 わ 跳躍 ちょうやく 子 こ 大 だい 多數 たすう 是 ぜ 造成 ぞうせい 單 たん 核 かく 多 た 態 たい 性 せい 的 てき 點 てん 只 ただ 改變 かいへん 核酸 かくさん 序列 じょれつ 上 じょう 的 てき 單 たん 一 いち 個 こ 也有 やゆう 規模 きぼ 的 てき 製 せい 插入 そうにゅう 反轉 はんてん 和 かず 轉移 てんい [13]
各種 かくしゅ 點 てん 發生 はっせい 的 てき 機 き 率 りつ 不同 ふどう 會 かい 影響 えいきょう GC含量 。點 てん 可分 かぶん 為 ため 同類 どうるい 置換 ちかん 又 また 譯 わけ 轉換 てんかん 指 ゆび 腺 せん 和 わ 鳥 とり 或 ある 胞嘧啶 和 わ 胸腺 きょうせん 之 これ 間 あいだ 的 てき 替 かえ 換 かわ 和 かず 異類 いるい 置換 ちかん 又 また 譯 わけ 指 ゆび 嘌呤 和 わ 嘧啶 之 これ 間 あいだ 的 てき 替 かえ 換 かわ 同類 どうるい 置換 ちかん 發生 はっせい 類 るい 置換 ちかん 則 そく 造成 ぞうせい 蛋白質 たんぱくしつ 的 てき 酸 さん 序列 じょれつ 改變 かいへん [13]
點 てん 是 ぜ 隨 ずい 機 き 發生 はっせい 的 てき 通常 つうじょう 機 き 率 りつ 在 ざい 大 だい 多數 たすう 生物 せいぶつ 中 ちゅう 單一 たんいつ 核 かく 的 てき 突變率 りつ 很低,約 やく 在 ざい 每 まい 世代 せだい 每 ごと 核酸 かくさん −9 ~10-8 ,不 ふ 過 か 在 ざい 病毒 びょうどく 中有 ちゅうう 時 じ 可 か 高 だか 達 たち −6 [14] 因 よし 為 ため 發生 はっせい 是 ぜ 緩慢 かんまん 地 ち 隨 ずい 著 ちょ 世代 せだい 累積 るいせき 隨 ずい 然 しか 每 まい 個 こ 世代 せだい 產 さん 生 せい 的 てき 數 すう 目 もく 並 なみ 不 ふ 固定 こてい 但 ただし 長時間 ちょうじかん 下 か 來 らい 會 かい 固定 こてい 的 てき 速 そく 率 りつ 累 るい 比較 ひかく 序列 じょれつ 之 の 間 あいだ 單 たん 核 かく 多 た 態 たい 性 せい 的 てき 數量 すうりょう 除 じょ 世代 せだい 的 てき 平均 へいきん 累積 るいせき 的 てき 數量 すうりょう 可 か 算出 さんしゅつ 兩個 りゃんこ 支 ささえ 系 けい 分 ぶん 多久 たく 此方 こちら 法 ほう 稱 たたえ 為 ため 分子 ぶんし 時 じ 鐘 かね [15]
取 と 決 けつ 的 てき 相對 そうたい 適應 てきおう 度 ど 新 しん 的 てき 可能 かのう 被 ひ 不同 ふどう 的 てき 演 えんじ 化 か 力量 りきりょう 影響 えいきょう 新 しん 對 たい 個體 こたい 有利 ゆうり 或 ある 有害 ゆうがい 會 かい 擇 よ 影響 えいきょう 是 ぜ 中性 ちゅうせい 不 ふ 影響 えいきょう 適應 てきおう 度 ど 則 のり 主要 しゅよう 遺傳 いでん 影響 えいきょう 參 まいり 見 み 下方 かほう 各 かく 機 き 制 せい 的 てき 相對 そうたい 重要 じゅうよう 性 せい 一節 いっせつ
遺傳 いでん 重 じゅう 組 ぐみ 會 かい 交換 こうかん 不同 ふどう 染色 せんしょく 體 たい 或 ある 同 どう 打破 だは 遺傳 いでん 連鎖 れんさ 讓 ゆずる 基 もと 因 いん 能 のう 自由 じゆう 組合 くみあい 並 なみ 降 くだ 低 ひく 遺傳 いでん 便 びん 車 しゃ 效 こう 應 おう 得 とく 天 てん 擇 よ 更 さら 有效 ゆうこう 率 りつ 因 いん 重 じゅう 組 ぐみ 率 りつ 高 だか 的 てき 區域 くいき 會 かい 保留 ほりゅう 有害 ゆうがい 有利 ゆうり 的 てき 等位 とうい 基 もと 因 いん 和 かず 複製 ふくせい 的 てき 錯誤 さくご [16]
重 じゅう 組 ぐみ 可能 かのう 造成 ぞうせい 當 とう 染色 せんしょく 體 たい 對 たい 齊 ひとし 失 しつ 誤 あやま 時 とき 重 じゅう 組 ぐみ 可能 かのう 造成 ぞうせい 許多 きょた 種 しゅ 大 だい 範圍 はんい 的 てき 還 かえ 有 ゆう 一種 いっしゅ 機 き 制 せい 稱 たたえ 為 ため 基 もと 因 いん 轉換 てんかん 是 ぜ 指 ゆび DNA修 おさむ 複 ふく 時 とき 在 ざい 切除 せつじょ 錯誤 さくご 的 てき 後 ご 使用 しよう 一 いち 個 こ 同 どう 源 みなもと 基 はじめ 因 いん 作為 さくい 模 も 板 ばん 來 らい 進行 しんこう DNA修復 しゅうふく ,合成 ごうせい 修復 しゅうふく 的 てき 基 もと 因 いん 轉換 てんかん 能 のう 造成 ぞうせい 等位 とうい 基 もと 因 いん 在 ざい 配 はい 子中 こなか 偏 へん 離 はなれ 的 てき 比例 ひれい 除 じょ 外 がい 還 かえ 會 かい 造成 ぞうせい 同 どう 源 みなもと 基 はじめ 因 いん 之 の 間 あいだ 的 てき 差異 さい 下降 かこう 降 くだ 低 てい 核酸 かくさん 分 ぶん 程度 ていど 稱 たたえ 為 ため 協同 きょうどう 演 えんじ 化 か [17]
模擬 もぎ 一 いち 個 こ 中性 ちゅうせい 的 てき 等位 とうい 基 もと 因 いん 的 てき 隨 ずい 機 き 世代 せだい 次 じ 並 なみ 比較 ひかく 三 さん 個 こ 不同 ふどう 的 てき 族 ぞく 群 ぐん 大小 だいしょう 同樣 どうよう 由 よし 的 てき 頻 しき 率 りつ 開始 かいし 上方 かみがた 族 ぞく 群 ぐん 時 じ 基 もと 因 いん 容易 ようい 失 しつ 去 さ 頻 しき 率 りつ 為 ため 或 ある 達 いたる 到 いた 固定 こてい 頻 しき 率 りつ 為 ため 方 かた 的 てき 族 ぞく 群 ぐん 則 そく 變 へん 的 てき 影響 えいきょう 遺傳 いでん 是 ぜ 因 いん 為 ため 族 ぞく 群 ぐん 大小 だいしょう 有限 ゆうげん 時 じ 隨 ずい 機 き 採 と 樣 よう 造成 ぞうせい 等位 とうい 基 もと 因 いん 頻 しき 率 りつ 在世 ざいせい 代 だい 間 あいだ 隨 ずい 機 き 改變 かいへん 即 そく 使 つかい 不同 ふどう 等位 とうい 基 もと 因 いん 有 ゆう 相 しょう 同 どう 的 てき 適應 てきおう 度 ど 因 いん 機 き 率 りつ 高 だか 或 ある 降 くだ 低 てい 頻 しき 率 りつ 直 ちょく 到 いた 頻 しき 率 ひきい 降 ぶ 到 いた 或 ある 升 ます 到 いた 固定 こてい 適應 てきおう 度 ど 不同 ふどう 若 わか 差異 さい 不 ふ 大 だい 或 ある 是 ぜ 有效 ゆうこう 族 ぞく 群 ぐん 大小 だいしょう 近 きん 中性 ちゅうせい 的 てき 變異 へんい 遺傳 いでん 的 てき 影響 えいきょう 許多 きょた 基 もと 因 いん 體 たい 的 てき 特徵 とくちょう 都 と 可能 かのう 作用 さよう 在 ざい 近 きん 中性 ちゅうせい 變異 へんい 上 じょう 的 てき 結果 けっか [18]
當 とう 生物 せいぶつ 的 てき 適應 てきおう 度 ど 不同 ふどう 時 じ 即 そく 會 かい 發生 はっせい 選擇 せんたく 是 ぜ 說 せつ 當 とう 競爭 きょうそう 配偶 はいぐう 或 ある 生育 せいいく 後代 こうだい 時 じ 個體 こたい 會 かい 產 さん 生 せい 後代 こうだい 因 いん 選擇 せんたく 包括 ほうかつ 自然 しぜん 選擇 せんたく 人工 じんこう 選擇 せんたく 和 わ 性 せい 選擇 せんたく 自然 しぜん 選擇 せんたく 源 みなもと 自 じ 個體 こたい 對 たい 機 き 或 ある 無機 むき 的 てき 自然 しぜん 環境 かんきょう 有 ゆう 不同 ふどう 的 てき 適應 てきおう 度 ど 性 せい 選擇 せんたく 源 みなもと 自 じ 擇 よ 好 このみ 有 ゆう 因 いん 能 のう 個 こ 體得 たいとく 到 いた 異性 いせい 的 てき 青 あお 見 み 得 とく 會 かい 個體 こたい 適應 てきおう 環境 かんきょう 人工 じんこう 選擇 せんたく 即 そく 育種 いくしゅ 是 ぜ 人為 じんい 選 せん 殖 ふえ 偏 へん 好 このみ 的 てき 特徵 とくちょう
各種 かくしゅ 選擇 せんたく 在 ざい 族 ぞく 群 ぐん 遺傳 いでん 學 がく 上 じょう 的 てき 原理 げんり 是 ぜ 類似 るいじ 的 てき 但 ただし 對 たい 分子 ぶんし 演 えんじ 化 か 的 てき 影響 えいきょう 不 ふ 同 どう 因 いん 為 ため 不同 ふどう 功 こう 能 のう 的 てき 基 もと 因 いん 常 つね 有 ゆう 不同 ふどう 的 てき 分 ぶん 例 れい 性徵 せいちょう 的 てき 基 もと 因 いん 常 つね 分 ぶん 性 せい 染色 せんしょく 體 たい 上 うえ 因 いん 選擇 せんたく 常 つね 影響 えいきょう 性 せい 染色 せんしょく 體 たい 的 てき 演 えんじ 化 か
選擇 せんたく 可 か 用 よう 在 ざい 基 もと 因 いん 個體 こたい 的 てき 層 そう 級 きゅう 即 そく 使 つかい 降 くだ 低 てい 個體 こたい 的 てき 適應 てきおう 度 ど 自 じ 私 わたし 可 か 化 か 造成 ぞうせい 基 もと 因 いん 組 ぐみ 衝突 しょうとつ 例 れい 子 こ 包括 ほうかつ 跳躍 ちょうやく 子 こ 減數 げんすう 分裂 ぶんれつ 驅動 くどう 者 しゃ 殺 ころせ 手 しゅ 染色 せんしょく 體 たい 自 じ 私 わたし 粒 つぶ 線 せん 體 たい 自我 じが 複製 ふくせい 子 こ 基 もと 因 いん 體 たい 的 てき 演 えんじ 化 か 有 ゆう 重大 じゅうだい 影響 えいきょう [19]
各 かく 機 き 制 せい 的 てき 相對 そうたい 重要 じゅうよう 性 せい [ 编辑 ] 基 もと 前述 ぜんじゅつ 各 かく 機 き 制 せい 的 てき 相對 そうたい 重要 じゅうよう 性 せい 分子 ぶんし 演 えんじ 化 か 可 か 出 で 三 さん 大 だい 派 は 觀點 かんてん [20]
天 てん 擇 よ 假說 かせつ 主張 しゅちょう 自然 しぜん 選擇 せんたく 是 ぜ 分子 ぶんし 演 えんじ 化 か 的 てき 主要 しゅよう 力量 りきりょう 論 ろん 同意 どうい 許多 きょた 是 ぜ 中性 ちゅうせい 的 てき 但 ただし 認 みとめ 為 ため 基 もと 因 いん 的 てき 頻 しき 率 りつ 主要 しゅよう 他 た 與 あずか 之 これ 有 ゆう 連鎖 れんさ 不 ふ 平衡 へいこう 的 てき 基 もと 因 いん 上 じょう 的 てき 天 てん 擇 よ 影響 えいきょう 是 ぜ 遺傳 いでん [21] 說 せつ 為 ため 微弱 びじゃく 的 てき 天 てん 擇 よ 足 あし 解釋 かいしゃく 密 みつ 偏差 へんさ [22]
中性 ちゅうせい 假說 かせつ 強調 きょうちょう 淨化 じょうか 選擇 せんたく 和 かず 遺傳 いでん [23] 木村 きむら 資 し 生 せい 建立 こんりゅう 的 てき 中性 ちゅうせい 理論 りろん 稱 たたえ 大 だい 多數 たすう 的 てき 是 ぜ 有害 ゆうがい 的 てき 會 かい 快速 かいそく 被 ひ 天 てん 擇 よ 移 うつり 除 じょ 餘 よ 下 か 的 てき 中 ちゅう 大 だい 多數 たすう 則 のり 是 ぜ 中性 ちゅうせい 的 てき 意 い 即 そく 不 ふ 影響 えいきょう 適應 てきおう 度 ど 只 ただ 有 ゆう 非常 ひじょう 少數 しょうすう 會 かい 提 ひさげ 高 だか 適應 てきおう 度 ど 數 すう 的中 てきちゅう 性 せい 是 ぜ 遺傳 いでん 影響 えいきょう 並 なみ 解釋 かいしゃく 核 かく 多 た 態 たい 性 せい 和物 あえもの 種 しゅ 差異 さい [24] [25] [26] 理論 りろん 地 ち 得 え 到 いた 傑 すぐる 克 かつ 金 きん 恩 おん 和 かず 湯 ゆ 秋 あき 克 かつ 的 てき 研究 けんきゅう 支持 しじ [27] 讓 ゆずる 學界 がっかい 激烈 げきれつ 爭 そう 辯 べん 新 しん 達 たち 爾 しか 文 ぶん 主義 しゅぎ 在 ざい 分子 ぶんし 演 えんじ 化 か 的 てき 重要 じゅうよう 性 せい 論 ろん 的 てき 一 いち 個 こ 變形 へんけい 是 ぜ 近 きん 中性 ちゅうせい 理論 りろん 認 みとめ 為 ため 大 だい 多數 たすう 接近 せっきん 中性 ちゅうせい 但 ただし 不 ふ 是 ぜ 完全 かんぜん 中性 ちゅうせい 因 いん 機 き 和 かず 天 たかし 擇 よ 都 と 強烈 きょうれつ 影響 えいきょう 分子 ぶんし 演 えんじ 化 か [25]
突變假說 かせつ 則 そく 強調 きょうちょう 在 ざい 分子 ぶんし 機 き 制 せい 上 じょう 的 てき 特定 とくてい 偏向 へんこう [28] 第 だい 他 た 主張 しゅちょう GC含量 的 てき 差異 さい 不 ふ 是 ぜ 天 てん 擇 よ 的 てき 影響 えいきょう 壓力 あつりょく 的 てき 結果 けっか [29]
基 もと 因 いん 體 たい 的 てき 各種 かくしゅ 特徵 とくちょう 包括 ほうかつ 小 しょう 序列 じょれつ 特徵 とくちょう 染色 せんしょく 體 たい 數 すう 目 もく 與 あずか 結構 けっこう 基 もと 因 いん 在 ざい 基 もと 因 いん 體 たい 中 ちゅう 的 てき 分 ぶん 等 とう 都 みやこ 是 ただし 分子 ぶんし 演 えんじ 化 か 的 てき 重要 じゅうよう 主題 しゅだい
基 もと 因 いん 體 たい 的 てき 大小 だいしょう 取 と 決 けつ 因 いん 的 てき 數量 すうりょう 和 わ 重複 じゅうふく 序列 じょれつ 的 てき 多寡 たか 基 もと 因 いん 體 たい 的 てき 大小 だいしょう 又 また 稱 しょう C值 ——和 わ 生物 せいぶつ 的 てき 複雜 ふくざつ 程度 ていど 並 なみ 沒 ぼつ 有 ゆう 相關 そうかん 看 み 似 に 不 ふ 合理 ごうり 的 てき 現象 げんしょう 稱 たたえ 為 ため C值謎 [30] 謎 なぞ 題 だい 的 てき 解釋 かいしゃく 有二 ゆうじ 首 くび 先 さき 不 ふ 會 かい 轉 てん 錄 ろく 非 ひ 編 へん 重複 じゅうふく 序列 じょれつ 可 か 非常 ひじょう 大 だい 的 てき 比例 ひれい 讓 ゆずる 基 もと 因 いん 體 たい 變 へん 得 とく 再 さい 者 しゃ 不 ふ 是 ぜ 每 ごと 個 こ 基 もと 因 いん 都 と 對應 たいおう 到 いた 特定 とくてい 的 てき 發育 はついく 階段 かいだん 或 ある 組織 そしき 類型 るいけい 基 もと 因 いん 功 こう 能 のう 重複 じゅうふく 或 ある 不 ふ 會 かい 表現 ひょうげん 或 ある 是 ぜ 只 ただ 調節 ちょうせつ 基 もと 他 た 基 もと 因 いん 讓 ゆずる 基 もと 因 いん 體 たい 變 へん 大 だい 增加 ぞうか 生物 せいぶつ 的 てき 複雜 ふくざつ 度 ど [31]
包括 ほうかつ 哺乳類 ほにゅうるい 在 ざい 許多 きょた 真 ま 核 かく 生物 せいぶつ 都 と 帶 たい 有 ゆう 大量 たいりょう 的 てき 非 ひ 編 へん 因 いん 體 たい 大小 だいしょう 和大 かずひろ 多數 たすう 生理 せいり 特徵 とくちょう 都 と 沒 ぼつ 有 ゆう 關連 かんれん 一般 いっぱん 認 みとめ 為 ため 真 ま 核 かく 生物 せいぶつ 的 てき 基 もと 因 いん 體 たい 大小 だいしょう 的 てき 天 てん 擇 よ 不 ふ 強 つよし 因 いん 多 た 都 と 是 ぜ 近 きん 中性 ちゅうせい 的 てき 重複 じゅうふく 序列 じょれつ 和 わ 非 ひ 編 へん 族 ぞく 群 ぐん 中 ちゅう 累積 るいせき 影響 えいきょう 個體 こたい 的 てき 適應 てきおう 力 りょく 不 ふ 過 か 鳥類 ちょうるい 的 てき 基 もと 因 いん 體 たい 重複 じゅうふく 序列 じょれつ 可能 かのう 是 ぜ 因 いん 為 ため 鳥類 ちょうるい 的 てき 紅 べに 血球 けっきゅう 有 ゆう 細胞 さいぼう 核 かく 大 だい 的 てき 細胞 さいぼう 核 かく 能 のう 帶 たい 的 てき 越 こし 少 しょう 但 ただし 鳥類 ちょうるい 是 ぜ 動物 どうぶつ 會 かい 飛行 ひこう 為 ため 了 りょう 維持 いじ 的 てき 代謝 たいしゃ 率 りつ 產 さん 生 せい 足 あし 能 のう 量 りょう 紅 べに 血球 けっきゅう 必需 ひつじゅ 有效 ゆうこう 率 りつ 地 ち 運送 うんそう 造成 ぞうせい 鳥類 ちょうるい 的 てき 的 てき 天 てん 擇 よ 有 ゆう 間接 かんせつ 證據 しょうこ 顯示 けんじ 恐龍 きょうりゅう 的 てき 基 もと 因 いん 體 たい 小 しょう 符合 ふごう 溫 ゆたか 調節 ちょうせつ 和 わ 的 てき 特徵 とくちょう [32] 許多 きょた 原核 げんかく 生物 せいぶつ 的 てき 基 もと 因 いん 體 たい 天 てん 擇 よ 影響 えいきょう 因 いん 為 ため 的 てき 基 もと 因 いん 體 たい 在 ざい 複製 ふくせい 時間 じかん 比較 ひかく 影響 えいきょう 個體 こたい 的 てき 適應 てきおう 度 ど
重複 じゅうふく 序列 じょれつ 和 わ 跳躍 ちょうやく 子 こ [ 编辑 ] 微 ほろ 衛星 えいせい 序列 じょれつ 在 ざい 複製 ふくせい 時 じ 容易 ようい 發生 はっせい 滑 すべり 動 どう 造成 ぞうせい 度 ど 改變 かいへん 圖 ず 中 ちゅう 的 てき 每 まい 個 こ 方 かた 格 かく 代表 だいひょう 相 しょう 同 どう 的 てき 一 いち 段 だん 重複 じゅうふく 序列 じょれつ 黑色 こくしょく 是 ぜ 舊 きゅう 的 てき 在 ざい 複製 ふくせい 時 じ 作為 さくい 模 も 板 ばん 白色 はくしょく 是 ぜ 新 しん 合成 ごうせい 的 てき 上 うえ 正常 せいじょう 無 む 的 てき 複製 ふくせい 中 なか 新 しん 合成 ごうせい 的 てき 產 さん 生 せい 環 たまき 圈 けん 長 ちょう 下 した 舊 きゅう 的 てき 產 さん 生 せい 環 たまき 圈 けん 造成 ぞうせい 新 しん 合成 ごうせい 的 てき DNA序列 じょれつ 裡 うら 面 めん 可 か 發現 はつげん 許多 きょた 不斷 ふだん 重複 じゅうふく 的 てき 序列 じょれつ 連續 れんぞく 在 ざい 串 くし 聯 れん 重複 じゅうふく 序列 じょれつ 可 か 再 さい 依 よ 單元 たんげん 序列 じょれつ 的 てき 長短 ちょうたん 分 ぶん 為 ため 衛星 えいせい 序列 じょれつ 小 しょう 衛星 えいせい 序列 じょれつ 和 わ 微 ほろ 衛星 えいせい 序列 じょれつ 分 ぶん 為 ため 分散 ぶんさん 的 てき 是 ぜ 散 ち 重複 じゅうふく 序列 じょれつ 可 か 分 ぶん 為 ため 短 たん 散 ち 件 けん 和 わ 長 なが 散 ち 件 けん 在 ざい 靈長 れいちょう 目 め 中 ちゅう 最 さい 常見 つねみ 的 てき 短 たん 散 ち 件 けん 是 ぜ Alu元 もと 件 けん ,長 なが 散 ち 件 けん 是 ぜ CRISPR/Cas 系統 けいとう 。[33] [34]
很多散 ち 重複 じゅうふく 是 ぜ 跳躍 ちょうやく 子 こ 即 そく 可 か 基 もと 因 いん 體 たい 中 ちゅう 自我 じが 複製 ふくせい 並 なみ 移動 いどう 位置 いち 的 てき 序列 じょれつ 許多 きょた 跳躍 ちょうやく 子 こ 序列 じょれつ 和 わ 病毒 びょうどく 序列 じょれつ 類似 るいじ [35] 跳躍 ちょうやく 子 こ 的 てき 演 えんじ 化 か 是 ぜ 因 いん 為 ため 單位 たんい 的 てき 演 えんじ 化 か 天 てん 擇 よ 直接 ちょくせつ 作用 さよう 在 ざい 跳躍 ちょうやく 子 こ 本身 ほんみ 帶 たい 有 ゆう 跳躍 ちょうやく 子 こ 的 てき 個體 こたい 跳躍 ちょうやく 子 こ 對 たい 基 もと 因 いん 體 たい 的 てき 演 えんじ 化 か 有 ゆう 重大 じゅうだい 影響 えいきょう [19]
不同 ふどう 物種 ものだね 帶 たい 有 ゆう 不同 ふどう 數量 すうりょう 的 てき 基 もと 因 いん 不同 ふどう 的 てき 模 も 式 しき 散布 さんぷ 在 ざい 因 いん 體 たい 有 ゆう 種 しゅ 多數 たすう 細菌 さいきん Drosophila 屬 ぞく 果 はて 蠅 はえ 擬 なずらえ 南 みなみ 芥 あくた 的 てき 基 もと 因 いん 體 たい 特別 とくべつ 非 ひ 編 へん 相 あい 下 か 哺乳 ほにゅう 動物 どうぶつ 和 わ 玉 だま 米 まい 的 てき 基 もと 因 いん 體 たい 則 のり 有 ゆう 大量 たいりょう 的 てき 重複 じゅうふく 序列 じょれつ 的 てき 內含子 こ 以及非 ひ 編 へん 基 もと 因 いん 的 てき 和 わ 分 ぶん 改變 かいへん 變 へん 率 りつ 影響 えいきょう 因 いん 整 せい 個物 こぶつ 種 しゅ 的 てき 演 えんじ 化 か 帶 おび 有 ゆう 內含子 こ 會 かい 使 し 編 へん 序列 じょれつ 之 の 間 あいだ 的 てき 距離 きょり 增加 ぞうか 基 もと 因 いん 比較 ひかく 容易 ようい 發生 はっせい 重 じゅう 組 ぐみ 包括 ほうかつ 異 い 位 い 重 じゅう 組 ぐみ 容易 ようい 造成 ぞうせい 新 しん 基 もと 因 いん 的 てき 形成 けいせい [30]
染色 せんしょく 體 たい 數 すう 目 もく 與 あずか 結構 けっこう [ 编辑 ] 一個生物的基因體內的染色 せんしょく 體 たい 數 すう 目 もく 在 ざい 物種 ものだね 之 の 間 あいだ 有 ゆう 非常 ひじょう 大 だい 的 てき 變化 へんか 並 なみ 和 わ 長 ちょう 度 ど 並 なみ 不 ふ 相關 そうかん 例 れい 克 かつ 跳 とべ 蟻 あり Myrmecia pilosula )只 ただ 有 ゆう 一 いち 對 たい 染色 せんしょく 體 たい [36] 心臟 しんぞう 葉 は 瓶 びん 爾 しか 小 しょう 草 くさ 有 ゆう 對 たい 染色 せんしょく 體 たい [37] 纖毛 せんもう 蟲 ちゅう 基 もと 因 いん 體 たい 每 ごと 因 いん 因 いん 之 の 間 あいだ 都 と 沒 ぼつ 有 ゆう 遺傳 いでん 連鎖 れんさ 天 てん 擇 よ 的 てき 效率 こうりつ 大 だい 為 ため 提 ひさげ 高 だか [38]
染色 せんしょく 體 たい 數 すう 目的 もくてき 演 えんじ 化 か 對 たい 物種 ものだね 形成 けいせい 可能 かのう 有 ゆう 輕重 けいちょう 的 てき 影響 えいきょう 因 いん 為 ため 不同 ふどう 的 てき 染色 せんしょく 體 たい 數 すう 目 もく 會 かい 造成 ぞうせい 生殖 せいしょく 隔離 かくり 人類 じんるい 2號 ごう 染色 せんしょく 體 たい 是 ぜ 融合 ゆうごう 自 じ 兩 りょう 條 じょう 黑 くろ 猩猩 しょうじょう 的 てき 染色 せんしょく 體 たい 有 ゆう 位 い 在中 ざいちゅう 間 あいだ 的 てき 端 はし 粒 つぶ 及第 きゅうだい 二 に 個 こ 殘留 ざんりゅう 的 てき 著 ちょ 絲 いと 粒 つぶ [39] 植物 しょくぶつ 中 ちゅう 常 つね 出現 しゅつげん 多倍 たばい 體 たい 特別 とくべつ 是 ぜ 異 い 源 みなもと 多倍 たばい 體 たい 造成 ぞうせい 雜交 ざっこう 後代 こうだい 和 かず 任 つとむ 在 ざい 一 いち 個 こ 世代 せだい 形成 けいせい 同 どう 域 いき 種 しゅ 化 か 點 てん 灰 はい 蝶 ちょう 屬 ぞく 蝴蝶 こちょう 的 てき 染色 せんしょく 體 たい 數 すう 目 もく 差異 さい 極大 きょくだい 從 したがえ 對 たい 到 いた 對 たい 都 と 有 ゆう 種 しゅ 化 か 頻 しき 率 りつ 最高 さいこう 的 てき 支 ささえ 系 けい 之 の 一 いち [40]
除 じょ 了 りょう 細胞 さいぼう 核 かく 基 もと 因 いん 內共生 きょうせい 胞器也有 やゆう 自己 じこ 的 てき 基 もと 因 いん 通常 つうじょう 是 ぜ 環狀 かんじょう 的 てき 質 しつ 體 たい 粒 つぶ 線 せん 體 からだ 和 わ 葉 は 綠 みどり 體 たい 在 ざい 不 ふ 同類 どうるい 群 ぐん 中 ちゅう 並 なみ 不 ふ 相 あい 同 どう 但 ただし 膜 まく 蛋白 たんぱく 尤 ゆう 和 わ 電子 でんし 傳 でん 的 てき 構成 こうせい 分子 ぶんし 通常 つうじょう 編 へん 在 ざい 基 もと 因 いん 中 ちゅう 大 だい 多數 たすう 生物 せいぶつ 的 てき 粒 つぶ 線 せん 體 からだ 和 わ 葉 は 綠 みどり 體 たい 是 ぜ 母系 ぼけい 遺傳 いでん 因 いん 為 ため 存在 そんざい 的 てき 卵 たまご 中 ちゅう 是 ぜ 精子 せいし 在 ざい 少數 しょうすう 特例 とくれい 孔雀 くじゃく 蛤 はまぐり 中 ちゅう 粒 つぶ 線 せん 體 からだ 是 ぜ 父系 ふけい 遺傳 いでん [41]
新 しん 的 てき 基 もと 因 いん 座 ざ 可 か 許多 きょた 種 しゅ 不同 ふどう 的 てき 機 き 制 せい 產 さん 生 せい 包括 ほうかつ 基 もと 因 いん 複製 ふくせい 反轉 はんてん 錄 ろく 複製 ふくせい 轉移 てんい 基 もと 因 いん 嵌 はま 合 ごう 基 もと 因 いん 非 ひ 編 へん 序列 じょれつ 等 とう 直接 ちょくせつ 形成 けいせい 機 き 制 せい [42]
基 もと 因 いん 複製 ふくせい 發生 はっせい 時 じ 了 りょう 重複 じゅうふく 重複 じゅうふく 的 てき 基 もと 因 いん 可 か 變 へん 出 で 不同 ふどう 功 こう 能 のう 影響 えいきょう 原本 げんぽん 的 てき 功 こう 能 のう 或 ある 是 ぜ 各 かく 段 だん 複製 ふくせい 各自 かくじ 專 せん 精 せい 原始 げんし 功 こう 能 のう 的 てき 一部 いちぶ 除 じょ 了 りょう 整 せい 個 こ 基 もと 因 いん 的 てき 複製 ふくせい 有 ゆう 時 じ 發生 はっせい 基 もと 中當 なかとう 中 ちゅう 的 てき 一部 いちぶ 製 せい 變成 へんせい 更 さら 長 ちょう 的 てき 基 もと 因 いん
反轉 はんてん 錄 ろく 轉移 てんい 基 もと 因 いん 體 たい 中 ちゅう 的 てき 反轉 はんてん 錄 ろく 轉 うたて 座 ざ 子 こ 左下 ひだりした 經 けい 轉 てん 錄 ろく 轉 てん 譯 やく 產 さん 生 せい 核 かく 白 しろ 複 ふく 合體 がったい 左上 ひだりうえ 然 しか 後 のち 反轉 はんてん 錄 ろく 產 さん 生 せい 右上 みぎうえ 插入 そうにゅう 基 もと 因 いん 體 たい 的 てき 位置 いち 右 みぎ 下 か 反轉 はんてん 錄 ろく 轉移 てんい 將 はた 信 しん 使 し 反轉 はんてん 錄 ろく 成 なる 並 なみ 基 もと 因 いん 體 たい 中 ちゅう 反轉 はんてん 錄 ろく 基 もと 因 いん 常 つね 插入 そうにゅう 到 いた 新 しん 的 てき 位置 いち 了 りょう 不同 ふどう 的 てき 基 もと 因 いん 表現 ひょうげん 和 かず 功 いさお 能 のう
嵌 はま 合 ごう 基 もと 因 いん 是 ぜ 當 とう 複製 ふくせい 或 ある 反轉 はんてん 錄 ろく 轉移 てんい 造成 ぞうせい 兩個 りゃんこ 不同 ふどう 的 てき 編 へん 序列 じょれつ 嵌 はま 合 ごう 在 ざい 一起 かずき 形成 けいせい 新 しん 的 てき 基 もと 因 いん 序列 じょれつ 嵌 はま 合 ごう 基 もと 因 いん 常 つね 造成 ぞうせい 調節 ちょうせつ 過程 かてい 的 てき 改變 かいへん 並 なみ 可能 かのう 重 おも 新 しん 排列 はいれつ 蛋白質 たんぱくしつ 域 いき 來 らい 產 さん 生新 せいしん 的 てき 功 こう 能 のう
直接 ちょくせつ 形成 けいせい 機 き 制 せい 已 やめ 在 ざい 酵母 こうぼ 菌 きん [43] 稻 いね 米 まい [44] 人類 じんるい [45] 中 ちゅう 發現 はつげん 直接 ちょくせつ 形成 けいせい 可 か 已 やめ 經 けい 低 てい 度 ど 表現 ひょうげん 的 てき 可 か 轉 てん 錄 ろく 序列 じょれつ 形成 けいせい [46] 終止 しゅうし 密 みつ 變成 へんせい 密 みつ 或 ある 是 ぜ 發生 はっせい 框 かまち 架 か 轉移 てんい 時 じ 則 のり 會 かい 形成 けいせい 延長 えんちょう 的 てき 蛋白質 たんぱくしつ 包含 ほうがん 原本 げんぽん 屬 ぞく 編 へん 序列 じょれつ 的 てき 部 ぶ 非 ひ 編 へん 是 ぜ [47] 例 れい 果 はて 蠅 はえ Drosophila melanogaster )的 てき 基 もと 因 いん 體 たい 中有 ちゅうう 至 いたり 少 しょう [48] [49]
分子 ぶんし 分類 ぶんるい 學 がく 結合 けつごう 傳統 でんとう 的 てき 系統 けいとう 分類 ぶんるい 學 がく 和 わ 分子 ぶんし 遺傳 いでん 學 がく 用 よう 或 ある 蛋白質 たんぱくしつ 序列 じょれつ 來 らい 回答 かいとう 系統 けいとう 分類 ぶんるい 學 がく 的 てき 問題 もんだい 即 そく 從 したがえ 演 えんじ 化生 かせい 物 ぶつ 學 がく 的 てき 角度 かくど 正確 せいかく 的 てき 生物 せいぶつ 分類 ぶんるい 法 ほう 或 ある 生物 せいぶつ 分類 ぶんるい 學 がく [50] 個 こ 長 ちょう 度 ど 的 てき 序列 じょれつ 分子 ぶんし 分類 ぶんるい 學 がく 的 てき 建立 こんりゅう 是 ぜ 在 ざい 許多 きょた 技術 ぎじゅつ 的 てき 發展 はってん 上 じょう DNA定 てい 序 じょ 可 か 確 かく 得知 とくち 或 ある 的 てき 將 はた 整 せい 個 こ 基 もと 因 いん 體 たい 定 てい 序 じょ 的 てき 成本 なりもと 正 ただし 快速 かいそく 下降 かこう 中 ちゅう 因 いん 全 ぜん 定 てい 序 じょ 的 てき 物種 ものだね 數量 すうりょう 正 せい 呈 てい 指數 しすう 上 じょう 升 ます [51]
和 かず 形態 けいたい 演 えんじ 化 か 的 てき 不同 ふどう 步 ふ [ 编辑 ] 特別 とくべつ 是 ぜ 在 ざい 在 ざい 單 たん 系 けい 物 ぶつ 中 ちゅう 分子 ぶんし 演 えんじ 化 か 和 わ 形態 けいたい 演 えんじ 化 か 有 ゆう 時 じ 並 なみ 不同 ふどう 步 ふ 會 かい 出現 しゅつげん 單一 たんいつ 形態 けいたい 多重 たじゅう 支 ささえ 系 けい 例 れい 趨同演 えんじ 化 か 和 わ 隱 かくれ 存 そん 種 しゅ 和 かず 單一 たんいつ 支 ささえ 系 けい 多重 たじゅう 形態 けいたい 例 れい 性狀 せいじょう 可塑 かそ 性 せい 和 かず 不同 ふどう 生命 せいめい 週 しゅう 期 き 有 ゆう 例 れい 可 か 中性 ちゅうせい 理論 りろん 解釋 かいしゃく [52]
分子生物學 ぶんしせいぶつがく 與 あずか 演 えんじ 化 か 社 しゃ 群 ぐん 定期 ていき 連載 れんさい 分子生物學 ぶんしせいぶつがく 與 あずか 演 えんじ 化 か 和 かず 基 もと 因 いん 體 たい 生物 せいぶつ 學 がく 與 あずか 演 えんじ 化 か 兩 りょう 刊 かん 並 なみ 年會 ねんかい [53] 領域 りょういき 的 てき 期 き 刊 かん 還 かえ 有 ゆう 分子 ぶんし 演 えんじ 化 か 期 き 刊 かん 和 かず 分子 ぶんし 分類 ぶんるい 和 わ 演 えんじ 化 か 分子 ぶんし 演 えんじ 化 か 的 てき 研究 けんきゅう 發表 はっぴょう 在 ざい 遺傳 いでん 學 がく 分子生物學 ぶんしせいぶつがく 基 もと 因 いん 體 たい 學 がく 系統 けいとう 分類 ぶんるい 學 がく 或 ある 演 えんじ 化生 かせい 物 ぶつ 學 がく 的 てき 學術 がくじゅつ 期 き 刊 かん
Li, W.-H. Molecular Evolution. Sinauer. 2006. ISBN 0-87893-480-4 Michael Lynch. The Origins of Genome Architecture. Sinauer. 2007. ISBN 0-87893-484-7 A. Meyer (Editor), Y. van de Peer, "Genome Evolution: Gene and Genome Duplications and the Origin of Novel Gene Functions", 2003, ISBN 978-1-4020-1021-7
T. Ryan Gregory, "The Evolution of the Genome", 2004, YSBN 978-0123014634
^ Dobzhanski, Sturtevant, 1937
^ Dietrich, "Paradox and Persuasion", pp. 90-91; Zuckerkandl, "On the Molecular Evolutionary Clock", p. 34
^ Dietrich, "Paradox and Persuasion", pp. 90-91; Morgan, "Emile Zuckerkandl, Linus Pauling, and the Molecular Evolutionary Clock", pp. 161-162.
^ Hagen, "Naturalists, Molecular Biologists, and the Challenges of Molecular Evolution", pp. 335-339
^ Dietrich, "The Origins of the Neutral Theory of Molecular Evolution", pp. 25-31
^ Dietrich, "The Origins of the Neutral Theory of Molecular Evolution", pp. 33-41
^ Hubby, J. L. Protein Differences in Drosophila . I. Drosophila melanogaster . Genetics. 1963, 48 (6): 871–879. PMC 1210521 PMID 17248176 ^ Hubby, J. L.; Lewontin, R. C. A Molecular Approach to the Study of Genic Heterozygosity in Natural Populations. I. The Number of Alleles at Different Loci in Drosophila pseudoobscura . Genetics. 1966, 54 : 546–595. Lewontin, R. C.; Hubby, J. L. A Molecular Approach to the Study of Genic Heterozygosity in Natural Populations. II. Amount of Variation and Degree of Heterozygosity in Natural Populations of Drosophila pseudoobscura . Genetics. 1966, 54 (2): 595–609. PMC 1211186 PMID 5968643 ^ Dietrich, "The Origins of the Neutral Theory of Molecular Evolution", pp. 42-45
^ Hagen, "Naturalists, Molecular Biologists, and the Challenge of Molecular Evolution", pp. 323-325
^ Zuckerkandl, "On the Molecular Evolutionary Clock", pp. 34-35
^ Dietrich, "Paradox and Persuasion", pp. 91-94
^ 13.0 13.1 Bergstro, Carl T. & Dugatkin, Lee Alan. Evolution. New York: W. W. Norton & Company. 2012: 190-193. ISBN 978-0-393-92592-0 ^ Michael Lynch. Evolution of the mutation rate . Trends in Genetics. August 2010, 26 (8): 345-352 [2015-07-16 ] . doi:10.1016/j.tig.2010.05.003 原始 げんし 内容 ないよう 存 そん ^ Daniel L. Hartl & Andrew G. Clark. Principles of Population Genetics. Sunderland, MA, USA: Sinauer Associates. 2007: 329-340. ISBN 978-0-87893-308-2 ^ Douglas J. Futuyma. Evolution 2. Sinauer Associates. 2009: 193 , 232-233. ISBN 978-0-87893-223-8 ^ Douglas J. Futuyma. Evolution 2. Sinauer Associates. 2009: 193 , 547. ISBN 978-0-87893-223-8 ^ Michael Lynch. The Origins of Genome Architecture. Sinauer. 2007. ISBN 0-87893-484-7 ^ 19.0 19.1 David J. Finnegan. Eukaryotic transposable elements and genome evolution . Trends in Genetics. 1989, 5 : 103-107 [2015-07-16 ] . ISSN 0168-9525 doi:10.1016/0168-9525(89)90039-5 原始 げんし 内容 ないよう 存 そん ^ Graur, D. and Li, W.-H. Fundamentals of molecular evolution . Sinauer. 2000. ISBN 0-87893-266-6 ^ Hahn, Matthew W. TOWARD A SELECTION THEORY OF MOLECULAR EVOLUTION. Evolution. February 2008, 62 (2): 255–265. PMID 18302709 doi:10.1111/j.1558-5646.2007.00308.x ^ Hershberg, Ruth; Petrov, Dmitri A. Selection on Codon Bias. Annual Review of Genetics. December 2008, 42 (1): 287–299. PMID 18983258 doi:10.1146/annurev.genet.42.110807.091442 ^ Kimura, M. The Neutral Theory of Molecular Evolution. Cambridge University Press , Cambridge. 1983. ISBN 0-521-23109-4 ^ Nachman M. C.W. Fox and J.B. Wolf , 编. " Detecting selection at the molecular level" in: Evolutionary Genetics: concepts and case studies: 103–118. 2006. ^ 25.0 25.1 Ohta, T. The nearly neutral theory of molecular evolution. Annual Review of Ecology and Systematics. 1992, 23 (1): 263–286. doi:10.1146/annurev.es.23.110192.001403 ^ Kimura, Motoo. Evolutionary rate at the molecular level (PDF) . Nature. 1968, 217 (5129): 624–626 [2015-07-16 ] . Bibcode:1968Natur.217..624K PMID 5637732 doi:10.1038/217624a0 原始 げんし 内容 ないよう 存 そん (PDF) 于2016-03-03). ^ King, J.L. and Jukes, T.H. Non-Darwinian Evolution (PDF) . Science . 1969, 164 (3881): 788–798 [2015-07-16 ] . Bibcode:1969Sci...164..788L PMID 5767777 doi:10.1126/science.164.3881.788 原始 げんし 内容 ないよう 存 そん (PDF) 于2019-12-31). ^ Nei, M. Selectionism and Neutralism in Molecular Evolution . Molecular Biology and Evolution. 2005, 22 (12): 2318–2342. PMC 1513187 PMID 16120807 doi:10.1093/molbev/msi242 ^ Sueoka, N. On the evolution of informational macromolecules. In: Bryson, V. and Vogel, H.J. (编). Evolving genes and proteins. Academic Press, New-York. 1964: 479–496. ^ 30.0 30.1 Thomas Jr, C. A. The genetic organization of chromosomes (PDF) . Annual review of genetics. 1971, 5 (1): 237-256 [2019-01-02 ] . (原始 げんし 内容 ないよう (PDF) 存 そん ^ Sean Eddy (2012) The C-value paradox, junk DNA, and ENCODE (页面存 そん ,存 そん 互联网档案 あん ), Curr Biol 22(21):R898–R899.
^ Organ, C. L.; Shedlock, A. M.; Meade, A.; Pagel, M.; Edwards, S. V. Origin of avian genome size and structure in nonavian dinosaurs. Nature. 2007, 446 : 180–184. doi:10.1038/nature05621 ^ Medical Subject Headings . National Library of Medicine. 2011 [2015-07-15 ] . (原始 げんし 内容 ないよう 存 そん ^ 鄭 てい 杏 あんず 微 ほろ 衛星 えいせい 科技 かぎ 部 ぶ 高 だか 自然 しぜん 科學 かがく 教學 きょうがく 資源 しげん 平台 ひらだい [2015-07-16 ] . (原始 げんし 内容 ないよう 存 そん ^ Thomas Wicker, François Sabot, Aurélie Hua-Van, Jeffrey L. Bennetzen, Pierre Capy, Boulos Chalhoub, Andrew Flavell, Philippe Leroy, Michele Morgante, Olivier Panaud, Etienne Paux, Phillip SanMiguel & Alan H. Schulman. A unified classification system for eukaryotic transposable elements . Nature Reviews Genetics. December 2007, 8 : 973-982 [2015-07-16 ] . doi:10.1038/nrg2165 原始 げんし 内容 ないよう 存 そん ^ Crosland, M.W.J., Crozier, R.H. Myrmecia pilosula , an ant with only one pair of chromosomes. Science. 1986, 231 (4743): 1278. Bibcode:1986Sci...231.1278C PMID 17839565 doi:10.1126/science.231.4743.1278 ^ Gerardus J. H. Grubben. Vegetables . PROTA. 2004: 404 [10 March 2013] . ISBN 978-90-5782-147-9原始 げんし 内容 ないよう 存 そん ^ Robert S. Coyne, Douglas L. Chalker, and and Meng-Chao Yao. GENOME DOWNSIZING DURING CILIATE DEVELOPMENT:Nuclear Division of Labor through Chromosome Restructuring . Annual Review of Genetics. December 1996, 30 : 557-578 [2015-07-16 ] . doi:10.1146/annurev.genet.30.1.557 原始 げんし 内容 ないよう 存 そん ^ Ijdo, J. W., Baldini, A., Ward, D. C., Reeders, S. T., & Wells, R. A. Origin of human chromosome 2: an ancestral telomere-telomere fusion. Proceedings of the National Academy of Sciences. 1991, 88 (20): 9051-9055. ^ Nikolai P. Kandul, Vladimir A. Lukhtanov, Naomi E. Pierce, KARYOTYPIC DIVERSITY AND SPECIATION IN AGRODIAETUS BUTTERFLIES, The Society for the Study of Evolution, 2007,, 61(3):546-559, doi:10.1111/j.1558-5646.2007.00046.x ^ Sophie Breton, Hélène Doucet Beaupré, Donald T. Stewart, Walter R. Hoeh, Pierre U. Blier. The unusual system of doubly uniparental inheritance of mtDNA: isn't one enough? . Trends in Genetics. September 2007, 23 (9): 465-474, [2015-07-16 ] . ISSN 0168-9525 doi:10.1016/j.tig.2007.05.011 原始 げんし 内容 ないよう 存 そん ^ D. V. Babushok, E. M. Ostertag, H. H. Kazazian Jr. Current topics in genome evolution: Molecular mechanisms of new gene formation . Cellular and Molecular Life Sciences. 2006-12-27, 64 (5): 542-554. ^ Cai J; Zhao R; Jiang H; et al. De novo origination of a new protein-coding gene in Saccharomyces cerevisiae . Genetics. 2008, 179 (1): 487–496. PMC 2390625 PMID 18493065 doi:10.1534/genetics.107.084491 ^ Xiao W; Liu H; Li Y; et al. El-Shemy, Hany A , 编. A rice gene of de novo origin negatively regulates pathogen- induced defense response . PLoS ONE. 2009, 4 (2): e4603. Bibcode:2009PLoSO...4.4603X PMC 2643483 PMID 19240804 doi:10.1371/journal.pone.0004603 ^ Knowles DG, McLysaght A. Recent de novo origin of human protein-coding genes . Genome Res. 2009, 19 (10): 1752–1759. PMC 2765279 PMID 19726446 doi:10.1101/gr.095026.109 ^ Wilson, Ben A.; Joanna Masel. Putatively Noncoding Transcripts Show Extensive Association with Ribosomes. Genome Biology & Evolution. 2011, 3 : 1245–1252. doi:10.1093/gbe/evr099 ^ Tautz, Diethard and Domazet-Lošo, Tomislav. The evolutionary origin of orphan genes. Nature Reviews Genetics. 2011, 12 (10): 692–702. PMID 21878963 doi:10.1038/nrg3053 ^ Levine MT; Jones CD; Kern AD; et al. Novel genes derived from noncoding DNA in Drosophila melanogaster are frequently X-linked and exhibit testis-biased expression . Proc Natl Acad Sci USA. 2006, 103 (26): 9935–9939. Bibcode:2006PNAS..103.9935L PMC 1502557 PMID 16777968 doi:10.1073/pnas.0509809103 ^ Zhou Q; Zhang G; Zhang Y; et al. On the origin of new genes in Drosophila . Genome Res. 2008, 18 (9): 1446–1455. PMC 2527705 PMID 18550802 doi:10.1101/gr.076588.108 ^ Suárez-Díaz, Edna and Anaya-Muñoz, Victor H. History, objectivity, and the construction of molecular phylogenies. Stud. Hist. Phil. Biol. & Biomed. Sci. 2008, 39 (4): 451–468. PMID 19026976 doi:10.1016/j.shpsc.2008.09.002 ^ Erika Check Hayden. Technology: The $1,000 genome. Nature. 2014-03-20, 507 : 294-295. doi:10.1038/507294a ^ Lahr, D. J.; Laughinghouse, H. D.; Oliverio, A. M.; Gao, F.; Katz, L. A. How discordant morphological and molecular evolution among microorganisms can revise our notions of biodiversity on Earth . BioEssays. 2014, 36 (10): 950–959 [2015-07-16 ] . doi:10.1002/bies.201400056 原始 げんし 内容 ないよう 存 そん ^ Society for Molecular Biology and Evolution . [2015-07-16 ] . (原始 げんし 内容 ないよう 存 そん




 . PMID 17248176.
. PMID 17248176.
 . PMID 5968643.
. PMID 5968643.
 . PMID 16120807. doi:10.1093/molbev/msi242.
. PMID 16120807. doi:10.1093/molbev/msi242.
 . PMID 18493065. doi:10.1534/genetics.107.084491.
. PMID 18493065. doi:10.1534/genetics.107.084491.
 . PMID 19240804. doi:10.1371/journal.pone.0004603.
. PMID 19240804. doi:10.1371/journal.pone.0004603.
 . PMID 19726446. doi:10.1101/gr.095026.109.
. PMID 19726446. doi:10.1101/gr.095026.109.
 . PMID 16777968. doi:10.1073/pnas.0509809103.
. PMID 16777968. doi:10.1073/pnas.0509809103.
 . PMID 18550802. doi:10.1101/gr.076588.108.
. PMID 18550802. doi:10.1101/gr.076588.108.