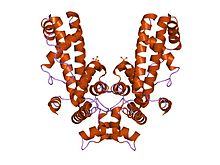
RNAポリメラーゼ[注釈 ちゅうしゃく (RNA polymerase ) とは、リボヌクレオチド を重合 じゅうごう RNA を合成 ごうせい 酵素 こうそ RNA合成 ごうせい 酵素 こうそ )。
DNAの鋳型 いがた 鎖 くさり 一本 いっぽん 鎖 くさり 塩基 えんき 配列 はいれつ 読 よ 取 と 相補 そうほ 的 てき 合成 ごうせい 反応 はんのう 転写 てんしゃ 触媒 しょくばい 中心 ちゅうしん 酵素 こうそ DNA依存 いぞん 性 せい という(単 たん 呼 よ 真 ま 核 かく 生物 せいぶつ DNA を鋳型 いがた mRNA やsnRNA 遺伝子 いでんし 多 おお 転写 てんしゃ RNAポリメラーゼII 知 し rRNA 前駆 ぜんく 体 たい 転写 てんしゃ RNAポリメラーゼI 、tRNA とU6 snRNA 、5S rRNA 前駆 ぜんく 体 たい 等 とう 転写 てんしゃ RNAポリメラーゼIII 三種 さんしゅ 依存 いぞん 性 せい 呼 よ
また、RNAを鋳型 いがた 合成 ごうせい RNA依存 いぞん 性 せい 多 おお RNAウイルス で重要 じゅうよう 機能 きのう 果 は 以外 いがい microRNA の増幅 ぞうふく 過程 かてい 利用 りよう
鋳型 いがた 必要 ひつよう 物 もの 初 はじ 発見 はっけん 酵素 こうそ 実際 じっさい 細菌 さいきん 細胞 さいぼう 内 うち ヌクレアーゼ として働 はたら 試験管 しけんかん 内 ない 合成 ごうせい 利用 りよう 一 いち 種類 しゅるい ヌクレオチド からなるRNAを合成 ごうせい 翻訳 ほんやく タンパク質 たんぱくしつ 調 しら 初 はじ 遺伝 いでん 暗号 あんごう 決定 けってい 行 おこな 真 ま 核 かく 生物 せいぶつ 同様 どうよう 鋳型 いがた 必要 ひつよう 転写 てんしゃ 産物 さんぶつ 末端 まったん 鎖 くさり 付加 ふか 転写 てんしゃ 後 ご 遺伝子 いでんし 発現 はつげん 制御 せいぎょ 機構 きこう 一端 いったん 担 にな
真 ま 核 かく 生物 せいぶつ 転写 てんしゃ 装置 そうち RNAポリメラーゼ )は、Pol I、Pol II、Pol IIIの3種 しゅ 種類 しゅるい 以上 いじょう 構成 こうせい 基本 きほん 的 てき 種 しゅ 古 こ 細菌 さいきん 数 すう 多 おお 種 しゅ 構成 こうせい ユーリ古 こ 細菌 さいきん ではいくつかのサブユニットが省 はぶ 一部 いちぶ クレン古 こ 細菌 さいきん には真 ま 核 かく 生物 せいぶつ 種類 しゅるい 全 すべ 保存 ほぞん 真 ま 核 かく 生物 せいぶつ 持 も 種 しゅ 祖先 そせん 型 がた 考 かんが 古 こ 細菌 さいきん 分 わ 特徴 とくちょう
一方 いっぽう 真正 しんしょう 細菌 さいきん 全体 ぜんたい 的 てき 真 ま 核 かく 生物 せいぶつ 古 こ 細菌 さいきん 単純 たんじゅん 構成 こうせい ω おめが 種 しゅ σ しぐま 会合 かいごう 呼 よ 形態 けいたい 正常 せいじょう 認識 にんしき シグマ因子 いんし は遺伝子 いでんし 上流 じょうりゅう プロモーター 配列 はいれつ 認識 にんしき 転写 てんしゃ 開始 かいし 役割 やくわり 担 にな
真正 しんしょう 細菌 さいきん [ 編集 へんしゅう ] 大腸菌 だいちょうきん RNAポリメラーゼホロ酵素 こうそ RNA polymerase holoenzyme は2分子 ぶんし α あるふぁ α あるふぁ 分子 ぶんし β べーた β べーた σ しぐま ω おめが サブユニット を含 ふく [1] σ しぐま 以外 いがい 複 ふく 合体 がったい 形成 けいせい RNAポリメラーゼコア酵素 こうそ (RNA polymerase core enzyme, コアポリメラーゼ (core polymerase))と呼 よ 酵素 こうそ 実際 じっさい 合成 ごうせい 部位 ぶい σ しぐま σ しぐま 因子 いんし 酵素 こうそ 特定 とくてい 遺伝子 いでんし 導 みちび 酵素 こうそ 特異 とくい 性 せい σ しぐま ギリシャ文字 もじ のs) を担 にな [2]
それぞれの項 こう 各 かく 紹介 しょうかい
RNAポリメラーゼホロ酵素 こうそ 存在 そんざい α あるふぁ 開始 かいし 段階 だんかい プロモーターのUPエレメント の認識 にんしき 担 にな 一方 いっぽう 伸長 しんちょう 段階 だんかい 酵素 こうそ 会合 かいごう 含 ふく 様々 さまざま 活性 かっせい 示 しめ
リチャード・グルース (Richard Grouse) らはα あるふぁ 末端 まったん アミノ酸 あみのさん 欠損 けっそん 正常 せいじょう α あるふぁ アミノ酸 あみのさん 数 すう アミノ酸 あみのさん 失 うしな 末端 まったん 番目 ばんめ アルギニン をシステイン に置換 ちかん α あるふぁ 変異 へんい 体 たい 実験 じっけん 行 おこな 酵素 こうそ 認識 にんしき 明 あき [3] リチャード・エブライト (Richard Ebright) らはタンパク質 たんぱくしつ 限定 げんてい 分解 ぶんかい 法 ほう 用 もち α あるふぁ 末端 まったん 末端 まったん 独立 どくりつ α あるふぁ α あるふぁ α あるふぁ α あるふぁ ドメイン を形成 けいせい 突 つ 止 と [4] 実験 じっけん 用 もち 生物 せいぶつ 大腸菌 だいちょうきん 末端 まったん 付近 ふきん 含 ふく kD 、C末端 まったん 末端 まったん 付近 ふきん 含 ふく 両者 りょうしゃ 明確 めいかく 構造 こうぞう モチーフ ) をとらない、少 すく アミノ酸 あみのさん 連結 れんけつ 鎖 くさり 発見 はっけん [5]
このことから、α あるふぁ 機能 きのう 一 ひと 仮説 かせつ 考 かんが 酵素 こうそ タンパク質 たんぱくしつ 相互 そうご 作用 さよう α あるふぁ α あるふぁ 連結 れんけつ 鎖 くさり 先 さき 酵素 こうそ 離 はな 対 たい 強力 きょうりょく 結合 けつごう 酵素 こうそ 強固 きょうこ 補 おぎな [5] [6] 後述 こうじゅつ RF複 ふく 合体 がったい 立体 りったい 構造 こうぞう 解析 かいせき から、2つあるUPエレメントのうち-40付近 ふきん α あるふぁ 付近 ふきん α あるふぁ 連結 れんけつ 示 しめ
β べーた 転写 てんしゃ 産物 さんぶつ 伸長 しんちょう 担 にな 結合 けつごう 部位 ぶい 持 も β べーた 末端 まったん [注釈 ちゅうしゃく 近 ちか Met 30〜Met102の領域 りょういき [7] 静 しずか 電 でん 相互 そうご 作用 さよう 弱 よわ 結合 けつごう エフゲニー・ナドラー (Evgeny Nudler) の1996年 ねん 実験 じっけん 結合 けつごう 標的 ひょうてき 転写 てんしゃ 中 ちゅう 部位 ぶい 融解 ゆうかい [8] 接続 せつぞく 中心 ちゅうしん 別 べつ β べーた 結合 けつごう 部位 ぶい β べーた 上流 じょうりゅう 位置 いち 上流 じょうりゅう 吐 は 出 だ 転写 てんしゃ 産物 さんぶつ 鋳型 いがた 鎖 くさり 結合 けつごう 脅 おど 活性 かっせい 大 おお 影響 えいきょう 別 べつ 実験 じっけん β べーた β べーた 結合 けつごう 関 かか [7]
ホロ酵素 こうそ 活性 かっせい 部位 ぶい 構成 こうせい タンパク質 たんぱくしつ 一 ひと 補 ほ 因子 いんし Mg+ と結合 けつごう アスパラギン酸 さん を持 も
β べーた 微生物 びせいぶつ 対 たい 代表 だいひょう 的 てき 抗生 こうせい 物質 ぶっしつ リファンピシン とストレプトリジギン の直接的 ちょくせつてき 作用 さよう 標的 ひょうてき [9] 抗生 こうせい 物質 ぶっしつ 転写 てんしゃ 伸長 しんちょう 阻害 そがい 開始 かいし 段階 だんかい 効果 こうか 開始 かいし 段階 だんかい 転写 てんしゃ 産物 さんぶつ 合成 ごうせい 過程 かてい [注釈 ちゅうしゃく 阻害 そがい [9]
β べーた 転写 てんしゃ 開始 かいし 段階 だんかい 酵素 こうそ 位 い 巻 ま 戻 もど 助 たす [9] 巻 ま 戻 もど 開放 かいほう 型 がた 複 ふく 合体 がったい 形成 けいせい [注釈 ちゅうしゃく 際 さい 非 ひ 鋳型 いがた 鎖 くさり [注釈 ちゅうしゃく 領域 りょういき 中 ちゅう 結合 けつごう 必要 ひつよう キャロル・グロス (Carol Gross) らの研究 けんきゅう 結合 けつごう β べーた アミノ酸 あみのさん 領域 りょういき 促 うなが [3]
伸長 しんちょう 段階 だんかい 酵素 こうそ 結合 けつごう 担 にな 末端 まったん [注釈 ちゅうしゃく 近 ちか Met 1230〜Met1273で+2〜+11の領域 りょういき 強 つよ 疎水 そすい 性 せい 相互 そうご 作用 さよう [8] 領域 りょういき β べーた 結合 けつごう 部位 ぶい 異 こと 転写 てんしゃ 中 ちゅう 二 に 重 じゅう
特 とく 転写 てんしゃ 開始 かいし 段階 だんかい [注釈 ちゅうしゃく 活躍 かつやく σ しぐま 因子 いんし 不 ふ 特定 とくてい 部位 ぶい 緩 ゆる 結合 けつごう 部位 ぶい 弱 よわ 結合 けつごう 滑 すべ 移動 いどう 出会 であ 遊離 ゆうり 転写 てんしゃ 行 おこな 遺伝子 いでんし 発見 はっけん 加速 かそく 速度 そくど 定数 ていすう 10 M-1 s-1 で、滑 すべ 無 む 差別 さべつ 結合 けつごう 解離 かいり 繰 く 返 かえ 場合 ばあい 倍 ばい [10] [11] 結合 けつごう 時 とき 安定 あんてい 性 せい 解離 かいり 半減 はんげん 期 き 約 やく 分 ふん 長 なが [11] σ しぐま 因子 いんし 秒 びょう 以下 いか [11] σ しぐま プロモーター を、半減 はんげん 期 き 数時間 すうじかん 強固 きょうこ 結合 けつごう 酵素 こうそ 会合 かいごう 定数 ていすう 配列 はいれつ 比較 ひかく 平均 へいきん 約 やく 7 倍 ばい 酵素 こうそ 平均 へいきん 倍 ばい [11] 結合 けつごう 定数 ていすう 6 〜1012 と幅広 はばひろ rRNA のような約 やく 秒 びょう 回 かい lacI 遺伝子 いでんし 約 やく 分 ふん 回 かい 転写 てんしゃ 頻度 ひんど 違 ちが 生 う 出 だ [12] 伸長 しんちょう 段階 だんかい 移行 いこう 必要 ひつよう 巻 ま 戻 もど 担 にな [注釈 ちゅうしゃく [13]
伸長 しんちょう 段階 だんかい 移行 いこう 構造 こうぞう 変 か σ しぐま 因子 いんし 結合 けつごう 極端 きょくたん 弱 よわ 研究 けんきゅう σ しぐま 因子 いんし 伸長 しんちょう 促進 そくしん [14] 二人 ふたり 年 ねん 論文 ろんぶん 離 はな σ しぐま 因子 いんし 別 べつ 酵素 こうそ 結合 けつごう 正常 せいじょう 転写 てんしゃ 行 おこな 証明 しょうめい [14] σ しぐま 因子 いんし 再 さい 利用 りよう 考 かんが σ しぐま 循環 じゅんかん 中 なか 当初 とうしょ 伸長 しんちょう 前 まえ 必 かなら 離 はな 考 かんが 現在 げんざい 結合 けつごう 弱 よわ 説 せつ 有力 ゆうりょく [15] 実際 じっさい 伸長 しんちょう 段階 だんかい 至 いた 酵素 こうそ σ しぐま 因子 いんし 保有 ほゆう [13] σ しぐま 因子 いんし 通常 つうじょう 伸長 しんちょう 止 と 別 べつ 酵素 こうそ 利用 りよう 離 はな
特別 とくべつ 遺伝子 いでんし 専任 せんにん σ しぐま 因子 いんし 真正 しんせい 細菌 さいきん 成長 せいちょう 機能 きのう 関 かん 遺伝子 いでんし 通常 つうじょう 増殖 ぞうしょく 必要 ひつよう 遺伝子 いでんし 転写 てんしゃ 主要 しゅよう σ しぐま 因子 いんし σ しぐま σ しぐま A ) を持 も 例 たと 大腸菌 だいちょうきん σ しぐま 70 であり、枯草 かれくさ 菌 きん σ しぐま 43 である[16] kD と43 kDで、右 みぎ 上 じょう 番号 ばんごう 分子 ぶんし 量 りょう 由来 ゆらい 熱 ねつ 遺伝子 いでんし 胞子 ほうし 形成 けいせい 遺伝子 いでんし 特別 とくべつ σ しぐま 因子 いんし 担当 たんとう [16] 多 おお 種類 しゅるい 環境 かんきょう 条件 じょうけん 適切 てきせつ 遺伝子 いでんし 群 ぐん 発現 はつげん 使 つか 分 わ 特 とく 枯草 かれくさ 菌 きん 用 もち 研究 けんきゅう 明 あき 普段 ふだん σ しぐま 43 が転写 てんしゃ 制御 せいぎょ 当 あ 栄養 えいよう 状態 じょうたい 悪 わる 場合 ばあい 他 た σ しぐま 因子 いんし σ しぐま H など)が発現 はつげん 胞子 ほうし 形成 けいせい 準備 じゅんび 始 はじ 後 ご 母 はは 細胞 さいぼう 変化 へんか 胞子 ほうし 使用 しよう
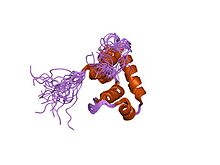
σ しぐま 因子 いんし 領域 りょういき [ 編集 へんしゅう ] あらゆる真正 しんせい 細菌 さいきん σ しぐま 因子 いんし アミノ酸 あみのさん 配列 はいれつ 領域 りょういき 分類 ぶんるい 各 かく 領域 りょういき 機能 きのう 以下 いか 提唱 ていしょう [17]
領域 りょういき 主要 しゅよう σ しぐま 因子 いんし 存在 そんざい σ しぐま 因子 いんし 伴 ともな 結合 けつごう 阻害 そがい 結合 けつごう 酵素 こうそ 結合 けつごう 後述 こうじゅつ 領域 りょういき ドメイン を露出 ろしゅつ σ しぐま 因子 いんし 単独 たんどく 結合 けつごう 酵素 こうそ 間 あいだ 結合 けつごう 阻害 そがい 機能 きのう 重要 じゅうよう
領域 りょういき 全 すべ σ しぐま 因子 いんし 存在 そんざい 生物 せいぶつ 最 もっと 共通 きょうつう 性 せい 高 たか [18] 領域 りょういき 分類 ぶんるい 特 とく 重要 じゅうよう 領域 りょういき 特異 とくい 的 てき 強 つよ 結合 けつごう 結合 けつごう 最適 さいてき α あるふぁ 形成 けいせい 予測 よそく アミノ酸 あみのさん 配列 はいれつ 含 ふく 実際 じっさい 認識 にんしき リチャード・ロジック (Richard Losick) が代償 だいしょう 変異 へんい 実験 じっけん 証明 しょうめい [18]
領域 りょういき 酵素 こうそ 両方 りょうほう 結合 けつごう 関与 かんよ 領域 りょういき 連結 れんけつ 鎖 くさり 転写 てんしゃ 最初 さいしょ 合成 ごうせい アデニン との特異 とくい 的 てき 結合 けつごう 関 かか [19] 出口 でぐち 通路 つうろ 塞 ふさ #真正 しんしょう 細菌 さいきん 酵素 こうそ 複 ふく 合体 がったい の(3)で詳述 しょうじゅつ 合成 ごうせい 本 ほん 弱 よわ 水素 すいそ 結合 けつごう 支 ささ 酵素 こうそ 特異 とくい 的 てき 結合 けつごう 必要 ひつよう 連結 れんけつ 鎖 くさり 欠 か 酵素 こうそ 用 もち 実験 じっけん 最初 さいしょ リボヌクレオチド の一方 いっぽう 両方 りょうほう 通常 つうじょう 高 こう 濃度 のうど 転写 てんしゃ 始 はじ 確認 かくにん [19]
領域 りょういき 分 わ 酵素 こうそ 認識 にんしき 重要 じゅうよう 考 かんが 領域 りょういき ヘリックスターンヘリックス というDNA結合 けつごう ドメイン を含 ふく 強 つよ 結合 けつごう
真正 しんしょう 細菌 さいきん 伸長 しんちょう 複 ふく 合体 がったい [ 編集 へんしゅう ] 伸長 しんちょう 段階 だんかい 実行 じっこう 中心 ちゅうしん 複 ふく 合体 がったい 立体 りったい 構造 こうぞう 研究 けんきゅう 年 ねん セス・ダースト (Seth Darst) によるThermus aquaticus 結晶 けっしょう 線 せん 回折 かいせつ 像 ぞう 基 もと 年 ねん 現在 げんざい 真正 しんしょう 細菌 さいきん 生物 せいぶつ 大腸菌 だいちょうきん 線 せん 結晶 けっしょう 構造 こうぞう 解析 かいせき 成功 せいこう 二 に 次元 じげん 結晶 けっしょう 電子 でんし 顕微鏡 けんびきょう 観察 かんさつ 大腸菌 だいちょうきん 全体 ぜんたい 形状 けいじょう 酷似 こくじ 詳細 しょうさい 構造 こうぞう 似 に 考 かんが [20]
真正 しんしょう 細菌 さいきん 酵素 こうそ [ 編集 へんしゅう ] T. Aquaticus のRNAポリメラーゼコア酵素 こうそ カニ のはさみ のようである。主 おも 一 ひと β べーた 一 ひと β べーた 占 し α あるふぁ ヒンジ にあり、それぞれβ べーた β べーた 結合 けつごう 小 ちい ω おめが β べーた 末端 まったん [注釈 ちゅうしゃく 巻 ま 峰 みね 存在 そんざい 触媒 しょくばい 活性 かっせい 中心 ちゅうしん 活性 かっせい 部位 ぶい β べーた β べーた 内部 ないぶ 活性 かっせい 中心 ちゅうしん 溝 みぞ 付 つ 根 ね [21] 広 ひろ 約 やく Å の空間 くうかん [22] [23] マグネシウム イオン (Mg2+ ) がβ べーた 中 ちゅう アスパラギン酸 さん にキレート 結合 けつごう アミノ酸 あみのさん 配列 はいれつ N A DF DG D(Dがアスパラギン酸 さん 含 ふく 全 すべ 細菌 さいきん 保存 ほぞん [20]
真正 しんしょう 細菌 さいきん 酵素 こうそ [ 編集 へんしゅう ] 2002年 ねん X線 せん 結晶 けっしょう 構造 こうぞう 解析 かいせき から3つの結論 けつろん 出 だ [24] σ しぐま 因子 いんし σ しぐま A )とβ べーた β べーた 間 あいだ 広 ひろ 範囲 はんい 相互 そうご 作用 さよう σ しぐま 因子 いんし 末端 まったん [注釈 ちゅうしゃく 1個 いっこ アミノ酸 あみのさん ドメイン 1.1欠損 けっそん 酵素 こうそ 通 とお 割 わ 目 め 小 ちい 1個 いっこ アミノ酸 あみのさん 割 わ 目 め 開 あ 結合 けつごう 推測 すいそく σ しぐま 因子 いんし 中 ちゅう 明確 めいかく 三 さん 次 じ 構造 こうぞう ループ はRNAポリメラーゼホロ酵素 こうそ 活性 かっせい 部位 ぶい 近 ちか 転写 てんしゃ 産物 さんぶつ 出口 でぐち 存在 そんざい
2番目 ばんめ 欠損 けっそん 部位 ぶい 解釈 かいしゃく 完全 かんぜん 酵素 こうそ 結晶 けっしょう 化 か 欠損 けっそん σ しぐま 撮影 さつえい 用 もち [25] 完全 かんぜん 構造 こうぞう 明 あき 予測 よそく 例 たと 回折 かいせつ 像 ぞう 切断 せつだん 末端 まったん α あるふぁ 端 はし 位置 いち 活性 かっせい 部位 ぶい 向 む 中性 ちゅうせい 約 やく 分 ぶん 残 ざん 基 もと 負 ふ 電荷 でんか 酸性 さんせい アミノ酸 あみのさん 非常 ひじょう 多 おお 塩基 えんき 性 せい アミノ酸 あみのさん 並 なら 活性 かっせい 部位 ぶい 強 つよ 結合 けつごう 小 ちい 入口 いりくち 開 あ 内部 ないぶ 結合 けつごう 考 かんが [25] 内部 ないぶ 融解 ゆうかい 酵素 こうそ 閉鎖 へいさ 型 がた 複 ふく 合体 がったい [注釈 ちゅうしゃく 考 かんが 際 さい 解離 かいり 内部 ないぶ 周辺 しゅうへん 活性 かっせい 部位 ぶい 閉 と 考 かんが 解離 かいり 閉鎖 へいさ 型 がた 複 ふく 合体 がったい 保護 ほご 開放 かいほう 型 がた 複 ふく 合体 がったい 移行 いこう ヒドロキシルラジカル にさらされるためのようである。リチャード・エブライト は閉鎖 へいさ 型 がた 複 ふく 合体 がったい 開放 かいほう 型 がた 複 ふく 合体 がったい 消 き 蛍光 けいこう 共鳴 きょうめい 移動 いどう 実験 じっけん 証明 しょうめい [25]
3番目 ばんめ 見解 けんかい 解釈 かいしゃく 第 だい 一 いち σ しぐま 因子 いんし 活性 かっせい 部位 ぶい 近 ちか リン酸 さん 結合 けつごう の形成 けいせい 携 たずさ 第 だい 二 に 連結 れんけつ 鎖 くさり 転写 てんしゃ 産物 さんぶつ 出口 でぐち 塞 ふさ 転写 てんしゃ 産物 さんぶつ 形成 けいせい 行 おこな [注釈 ちゅうしゃく 転写 てんしゃ 産物 さんぶつ 形成 けいせい 連結 れんけつ 鎖 くさり 開始 かいし 段階 だんかい 合成 ごうせい 出口 でぐち 占有 せんゆう 競合 きょうごう 仮説 かせつ [26] 連結 れんけつ 鎖 くさり 勝 か 伸長 しんちょう 中断 ちゅうだん 短 みじか 転写 てんしゃ 産物 さんぶつ 放出 ほうしゅつ 転写 てんしゃ 産物 さんぶつ 完成 かんせい 転写 てんしゃ 産物 さんぶつ 過剰 かじょう 合成 ごうせい 大腸菌 だいちょうきん 倍 ばい 過剰 かじょう [26] 過程 かてい 何 なん 度 ど 繰 く 返 かえ 約 やく 以上 いじょう 成長 せいちょう 競合 きょうごう 勝 か 連結 れんけつ 鎖 くさり 結果 けっか 酵素 こうそ σ しぐま 因子 いんし 結合 けつごう 弱 よわ 酵素 こうそ 解離 かいり 伸長 しんちょう 移行 いこう 備 そな 連結 れんけつ 鎖 くさり 欠損 けっそん σ しぐま 因子 いんし 転写 てんしゃ 産物 さんぶつ 多量 たりょう 生産 せいさん 確認 かくにん [26] 転写 てんしゃ 産物 さんぶつ σ しぐま 因子 いんし 活性 かっせい 部位 ぶい 存在 そんざい 副産物 ふくさんぶつ 推測 すいそく 伸長 しんちょう 礎 いしずえ 短 みじか 結合 けつごう σ しぐま 因子 いんし 活性 かっせい 部位 ぶい 接近 せっきん 必然 ひつぜん 的 てき 連結 れんけつ 鎖 くさり 出口 でぐち 塞 ふさ 考 かんが [26]
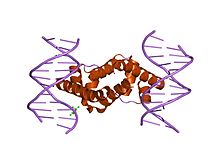
真正 しんしょう 細菌 さいきん 酵素 こうそ 複 ふく 合体 がったい [ 編集 へんしゅう ] T. aquaticus のRNAポリメラーゼ伸長 しんちょう 複 ふく 合体 がったい 青 あお 赤 あか 活性 かっせい 部位 ぶい マグネシウム イオン は黄色 おうしょく 示 しめ ホロ酵素 こうそ 形成 けいせい 複 ふく 合体 がったい 転写 てんしゃ 時 じ 状態 じょうたい RF複 ふく 合体 がったい (replicative form complex、RFは複製 ふくせい 型 がた 呼 よ 下図 したず フォークジャンクションDNA にT. aquaticus のDNAポリメラーゼホロ酵素 こうそ 結合 けつごう 複 ふく 合体 がったい 作成 さくせい -35ボックス を含 ふく 二 に 本 ほん 鎖 くさり -10ボックス 中 なか 非 ひ 鋳型 いがた 鎖 くさり [注釈 ちゅうしゃく 位 い 始 はじ 一本 いっぽん 鎖 くさり 突出 とっしゅつ 部分 ぶぶん 持 も 開放 かいほう 型 がた 複 ふく 合体 がったい 状態 じょうたい 模倣 もほう 詳 くわ #真正 しんしょう 細菌 さいきん β べーた )。
RF複 ふく 合体 がったい 立体 りったい 構造 こうぞう 様々 さまざま 事実 じじつ 判明 はんめい 酵素 こうそ 結合 けつごう σ しぐま 場所 ばしょ 横切 よこぎ 大腸菌 だいちょうきん プロモーター においては、-12位 い 塩基 えんき σ しぐま 70 因子 いんし 領域 りょういき Gln 437およびThr 440と相互 そうご 作用 さよう T. aquaticus のσ しぐま A で2つのアミノ酸 あみのさん Asn 263とに相当 そうとう
Trp 256は-10ボックス 直前 ちょくぜん 位 い 非常 ひじょう 近 ちか T. aquaticus σ しぐま A のPhe 248、Tyr 253、Trp256や大腸菌 だいちょうきん σ しぐま 70 における一部 いちぶ 芳香 ほうこう 族 ぞく アミノ酸 あみのさん 高度 こうど 保存 ほぞん 開放 かいほう 型 がた 複 ふく 合体 がったい 非 ひ 鋳型 いがた 鎖 くさり 結合 けつごう 融解 ゆうかい 関与 かんよ 予測 よそく 観察 かんさつ 位置 いち 位 い 塩基 えんき 対 たい 代 か 融解 ゆうかい 促進 そくしん 可能 かのう 性 せい 高 たか
σ しぐま 領域 りょういき 保存 ほぞん 塩基 えんき 性 せい アミノ酸 あみのさん Arg 237とLys 241)が 静 しずか 電 でん 相互 そうご 作用 さよう 結合 けつごう 観察 かんさつ 領域 りょういき 残 ざん 基 もと 35ボックス に結合 けつごう 複 ふく 合体 がったい 結晶 けっしょう 化 か 際 さい 領域 りょういき 対 たい 正常 せいじょう 位置 いち 押 お 出 だ 結論 けつろん 付 つ [27] 自身 じしん 撮影 さつえい 複 ふく 合体 がったい 構造 こうぞう 他 た 証拠 しょうこ 以下 いか 仮説 かせつ 提唱 ていしょう [27] 上流 じょうりゅう 二 に 本 ほん 鎖 くさり 曲 ま DNaseⅠ の標的 ひょうてき 部位 ぶい 生 しょう 一方 いっぽう 下流 かりゅう 領域 りょういき 二 に 重 じゅう 融解 ゆうかい 閉鎖 へいさ 型 がた 開放 かいほう 型 がた 複 ふく 合体 がったい 移行 いこう 開放 かいほう 型 がた 複 ふく 合体 がったい 各 かく タンパク質 たんぱくしつ 相互 そうご 作用 さよう 立体 りったい 的 てき 解析 かいせき β べーた β べーた 間 あいだ 融解 ゆうかい β べーた 舵 かじ 型 がた 構造 こうぞう 維持 いじ 構造 こうぞう β べーた 表面 ひょうめん 隣接 りんせつ β べーた 向 む 分離 ぶんり 鎖 くさり 間隙 かんげき 突 つ 出 だ 再 さい 会合 かいごう 阻止 そし
活性 かっせい 部位 ぶい + が3つのアスパラギン酸 さん によって支 ささ
非 ひ 鋳型 いがた 鎖 くさり 拡張 かくちょう 鋳型 いがた 鎖 くさり 図 ず 複 ふく 合体 がったい 作成 さくせい 使用 しよう
少 すく 開放 かいほう 型 がた 複 ふく 合体 がったい 時点 じてん 酵素 こうそ 内部 ないぶ 通 つう 通路 つうろ [21] NTP取 と 込 こ 通路 つうろ は基質 きしつ 触媒 しょくばい 活性 かっせい 中心 ちゅうしん 迎 むか 入 い RNA出口 でぐち 通路 つうろ は後 ご 伸長 しんちょう 段階 だんかい 合成 ごうせい 鎖 くさり 部分 ぶぶん 出 だ 通路 つうろ 出入 でい 使 つか 下流 かりゅう 下流 かりゅう 用 よう 通路 つうろ 二 に 重 じゅう 活性 かっせい 中心 ちゅうしん 溝 みぞ 入 はい 本 ほん 一本 いっぽん 鎖 くさり 分 わ [28] 非 ひ 鋳型 いがた 鎖 くさり 非 ひ 鋳型 いがた 鎖 くさり 用 よう 通路 つうろ 通路 つうろ 抜 ぬ 酵素 こうそ 表面 ひょうめん 沿 そ 進 すす 一方 いっぽう 鋳型 いがた 鎖 くさり 触媒 しょくばい 活性 かっせい 溝 みぞ 突 つ 進 すす 鋳型 いがた 鎖 くさり 用 よう 通路 つうろ 通路 つうろ 外 そと 出 で 一本 いっぽん 鎖 くさり 酵素 こうそ 後方 こうほう 上流 じょうりゅう 位置 いち 二 に 重 じゅう 戻 もど [28]
真 ま 核 かく 生物 せいぶつ [ 編集 へんしゅう ] α あるふぁ 赤 あか 結合 けつごう 真 ま 核 かく 生物 せいぶつ 毒 どく 合成 ごうせい 阻害 そがい 真 ま 核 かく 生物 せいぶつ I 、II 、III といった3種類 しゅるい 年 ねん ロバート・ローダー (Robert Roeder) とウィリアム・ラター William Rutter が発見 はっけん [29] 合成 ごうせい 異 こと rRNA 前駆 ぜんく 体 たい 合成 ごうせい タンパク質 たんぱくしつ 謎 なぞ 多 おお ヘテロ核 かく 内 ない (heterogeneous nuclear RNA, hnRNA ) や大 だい 部分 ぶぶん 核 かく 内 ない 低 てい 分子 ぶんし snRNA ) を合成 ごうせい 成熟 せいじゅく 合成 ごうせい 関 かか tRNA や5S rRNA、前述 ぜんじゅつ 別 べつ 前駆 ぜんく 体 たい 担 にな 細胞 さいぼう 内 ない 分布 ぶんぷ 別 べつ 核 かく 小体 こてい 核質 かくしつ 存在 そんざい
細菌 さいきん 開始 かいし 因子 いんし σ しぐま 因子 いんし 真 ま 核 かく 生物 せいぶつ 複数 ふくすう 基本 きほん 転写 てんしゃ 因子 いんし GTF ) を必要 ひつよう 実際 じっさい ヌクレオソーム があるためさらにDNA結合 けつごう 調節 ちょうせつ 介在 かいざい 複 ふく 合体 がったい 修飾 しゅうしょく 酵素 こうそ タンパク質 たんぱくしつ 必要 ひつよう [30]
RNAポリメラーゼIIのサブユニット [ 編集 へんしゅう ] RNAポリメラーゼIIのサブユニット構成 こうせい 年 ねん ピエール・シャンボン (Pierre Chambon) らとラターらのグループから独立 どくりつ 報告 ほうこく [31] 時 とき 不完全 ふかんぜん 年 ねん マウス 由来 ゆらい 全 すべ 完全 かんぜん 情報 じょうほう 明 あき [31] 現在 げんざい 全 ぜん 種 しゅ 正確 せいかく 判明 はんめい
ヒト と酵母 こうぼ 個 こ 下 した 表 ひょう 各々 おのおの 単独 たんどく 遺伝子 いでんし 各 かく 名前 なまえ 遺伝子 いでんし 名前 なまえ 由来 ゆらい 名称 めいしょう 用 もち 呼 よ 名 な
リチャード・ヤング (Richard Young) はエピトープタグ法 ほう で同定 どうてい 個 こ 分類 ぶんるい [31] 真正 しんしょう 細菌 さいきん 酵素 こうそ 構造 こうぞう 機能 きのう 類似 るいじ コアサブユニット 、少 すく 酵母 こうぼ 種類 しゅるい 核 かく 内 ない 全 すべ 共通 きょうつう 必 かなら 酵素 こうそ 活性 かっせい 必要 ひつよう 非 ひ 必須 ひっす
電気 でんき 泳 およげ 動 どう 結果 けっか IIa と240 kDと測定 そくてい IIo の2つの形態 けいたい 存在 そんざい a のC末端 まったん CTD (carboxyl-terminal domain) と呼 よ 個 こ アミノ酸 あみのさん heptad )から成 な 共通 きょうつう 配列 はいれつ Tyr -Ser -Pro -Thr -Ser-Pro-Ser が反復 はんぷく 配列 はいれつ o はCTDのヒドロキシ基 もと を持 も アミノ酸 あみのさん リン酸化 さんか したものである。しかし、哺乳類 ほにゅうるい 回 かい 反復 はんぷく 全 すべ 酸化 さんか a とIIo の分子 ぶんし 量 りょう 差 さ 埋 う 実際 じっさい 分子 ぶんし 量 りょう 大 おお 見 み 泳 およげ 動 どう 度 ど 遅 おそ 酸化 さんか 立体 りったい 構造 こうぞう 変化 へんか 引 ひ 起 お 考 かんが 異 こと 所有 しょゆう RNAポリメラーゼIIA (RNA polymerase IIA) およびRNAポリメラーゼIIO (RNA polymerase IIO) と区別 くべつ 前者 ぜんしゃ プロモーター に最初 さいしょ 結合 けつごう 形態 けいたい 後者 こうしゃ 伸長 しんちょう 反応 はんのう 行 おこな
ヒトと酵母 こうぼ [32]
サブユニット
酵母 こうぼ 遺伝子 いでんし 酵母 こうぼ タンパク質 たんぱくしつ モル質量 しつりょう (kD)特徴 とくちょう
hRPB1
RPb1 192
コアサブユニット。CTDを含 ふく 結合 けつごう 選別 せんべつ 関与 かんよ β べーた 相 あい 同 どう
hRPB2
RPb2 139
活性 かっせい 部位 ぶい 含 ふく 認識 にんしき 伸長 しんちょう 速度 そくど 関与 かんよ β べーた 相 あい 同 どう
hRPB3
RPb3 35
コアサブユニット。原核 げんかく 生物 せいぶつ α あるふぁ 相 あい 同 どう 機能 きのう 可能 かのう 性 せい
hRPB4
RPb4 25
非 ひ 必須 ひっす 複 ふく 合体 がったい 形成 けいせい 応答 おうとう 関与 かんよ
hRPB5
RPb5 25
共通 きょうつう 転写 てんしゃ アクチベーター の標的 ひょうてき
hRPB6
RPb6 18
共通 きょうつう 複 ふく 合体 がったい 形成 けいせい 安定 あんてい 化 か 寄与 きよ
hRPB7
RPb7 19
定常 ていじょう 期 き 複 ふく 合体 がったい 形成 けいせい
hRPB8
RPb8 17
共通 きょうつう オリゴヌクレオチド /オリゴ糖 とう 結合 けつごう ドメイン 。
hRPB9
RPb9 14
伸長 しんちょう 関与 かんよ 可能 かのう 性 せい Znリボンモチーフ を含 ふく 認識 にんしき
hRPB10
RPb10 8
共通 きょうつう
hRPB11
RPb11 14
原核 げんかく 生物 せいぶつ α あるふぁ 相 あい 同 どう 機能 きのう 可能 かのう 性 せい
hRPB12
RPb12 8
共通 きょうつう
RNAポリメラーゼIIの立体 りったい 構造 こうぞう [ 編集 へんしゅう ] ロジャー・コーンバーグらは2001年 ねん X線 せん 構造 こうぞう 解析 かいせき の結果 けっか 発表 はっぴょう [33] 結晶 けっしょう 化 か 難 むずか 撮影 さつえい 用 もち 欠 か 酵母 こうぼ 変異 へんい 株 かぶ Δ でるた 転写 てんしゃ 開始 かいし 伸長 しんちょう 反応 はんのう 問題 もんだい
全体 ぜんたい 構造 こうぞう 巨大 きょだい 顎 あご 酸性 さんせい 深 ふか 溝 みぞ 残 のこ 酵素 こうそ 表面 ひょうめん 酸性 さんせい 対 たい 溝 みぞ 塩基 えんき 性 せい 残 ざん 基 もと 並 なら 上顎 じょうがく 下 しも 顎 あご 底 そこ 触媒 しょくばい 活性 かっせい 中心 ちゅうしん 個 こ Mg2+ があり、コーンバーグらはメタルA (metal A) とメタルB (metal B) に区別 くべつ [34] 個 こ アスパラギン酸 さん と強固 きょうこ 結合 けつごう 一方 いっぽう E 836とD837に囲 かこ 配 はい 位 い 結合 けつごう 距離 きょり 触媒 しょくばい 反応 はんのう 過程 かてい 酸性 さんせい アミノ酸 あみのさん 近 ちか 考 かんが 基質 きしつ 三 さん 酸 さん 結合 けつごう
真正 しんしょう 細菌 さいきん 同様 どうよう ポア1 (pore 1) という、合成 ごうせい 出 だ 出口 でぐち 存在 そんざい 漏斗 ろうと 状 じょう 外縁 がいえん 出 で 切断 せつだん TFIIS と結合 けつごう アミノ酸 あみのさん 並 なら [34] 一方 いっぽう 入 い 口 くち 及 およ クランプモジュール (clamp module) が回転 かいてん 開閉 かいへい [35] 酵素 こうそ 表面 ひょうめん 相補 そうほ 鎖 くさり 外 そと 残 のこ 鋳型 いがた 鎖 くさり 溝 みぞ 中 なか 誘導 ゆうどう
RNAポリメラーゼIIの伸長 しんちょう 複 ふく 合体 がったい [ 編集 へんしゅう ] コーンバーグらはDNAと合成 ごうせい 両方 りょうほう 結合 けつごう 撮影 さつえい 成功 せいこう [35] 単独 たんどく 開 ひら 外 そと 活性 かっせい 中心 ちゅうしん 近 ちか 伸長 しんちょう 複 ふく 合体 がったい 閉 と 鋳型 いがた 鎖 くさり 転写 てんしゃ 産物 さんぶつ 覆 おお [36] 後述 こうじゅつ 転写 てんしゃ 中 ちゅう 内部 ないぶ 折 お 曲 ま 転写 てんしゃ 開始 かいし 前 まえ 比較的 ひかくてき 強固 きょうこ 構造 こうぞう [37] 最初 さいしょ 入 い 開 ひら 途中 とちゅう 酵素 こうそ 離 はな 閉 と 最近 さいきん 付加 ふか 間 あいだ リン酸 さん に結合 けつごう 位置 いち [36] 活性 かっせい 中心 ちゅうしん 近 ちか 溝 みぞ ブリッジヘリックス (bridge helix) が観察 かんさつ 伸 の 状態 じょうたい 基質 きしつ 三 さん 酸 さん 入 い 開 ひら 一方 いっぽう Ala 832の付近 ふきん 曲 ま 状態 じょうたい 活性 かっせい 中心 ちゅうしん 閉 と [36]
内部 ないぶ 入口 いりくち 所 ところ 先 さき 壁 かべ 無理 むり 曲 ま 酵素 こうそ 表面 ひょうめん 鋳型 いがた 鎖 くさり 二 に 重 じゅう 形成 けいせい 長 なが ラダー (rudder, 舵 かじ 呼 よ タンパク質 たんぱくしつ 障害 しょうがい 物 ぶつ 制限 せいげん [37] 以上 いじょう 付加 ふか 塩基 えんき 対 たい 形成 けいせい 最後 さいご 離 はな 出口 でぐち 抜 ぬ 出 だ 別 べつ 出口 でぐち 脱出 だっしゅつ 鋳型 いがた 鎖 くさり 非 ひ 鋳型 いがた 鎖 くさり 二 に 重 じゅう 戻 もど 進路 しんろ 下流 かりゅう 前 まえ 後 うし 壁 かべ 上 うえ 出口 でぐち 下 した 開 ひら [37]
^ 「ポリメラーゼ」は、より英語 えいご 発音 はつおん 近 ちか 表記 ひょうき
^ a b c d タンパク質 たんぱくしつ 様々 さまざま 立体 りったい 構造 こうぞう 本来 ほんらい アミノ酸 あみのさん 鎖 くさり 直 ちょく 鎖 くさり 状 じょう 高分子 こうぶんし 直 ちょく 鎖 くさり 末端 まったん 残 ざん 基 もと アミノ基 もと か酢酸 さくさん 末端 まったん 末端 まったん 区別 くべつ 酵素 こうそ 活性 かっせい 転写 てんしゃ 複製 ふくせい 末端 まったん 末端 まったん 進 すす タンパク質 たんぱくしつ アミノ酸 あみのさん 構成 こうせい 示 しめ 末端 まったん 左 ひだり 順番 じゅんばん アミノ酸 あみのさん 書 か 並 なら 中 なか 特定 とくてい アミノ酸 あみのさん 位置 いち 区間 くかん 末端 まったん 数 かぞ 番号 ばんごう 示 しめ
^ a b c d 転写 てんしゃ 開始 かいし 伸長 しんちょう 終了 しゅうりょう 段階 だんかい 開始 かいし 段階 だんかい 酵素 こうそ 形成 けいせい プロモーター に結合 けつごう 初 はじ 二 に 重 じゅう 形成 けいせい 酵素 こうそ 閉鎖 へいさ 型 がた 複 ふく 合体 がったい 呼 よ 後 ご 二 に 重 じゅう 開放 かいほう 型 がた 複 ふく 合体 がったい 転写 てんしゃ 産物 さんぶつ 呼 よ 数 すう 合成 ごうせい 伸長 しんちょう 段階 だんかい 入 はい 遺伝子 いでんし 本格 ほんかく 的 てき 転写 てんしゃ
^ a b 転写 てんしゃ 開始 かいし 段階 だんかい 合成 ごうせい 本来 ほんらい 二 に 重 じゅう 一本 いっぽん 巻 ま 戻 もど 二 に 重 じゅう 結合 けつごう 次 つぎ 巻 ま 戻 もど 行 おこな 一 いち 本 ほん 鎖 くさり 酵素 こうそ 開放 かいほう 型 がた 複 ふく 合体 がったい 呼 よ
^ a b DNAは二 に 重 じゅう 形成 けいせい 転写 てんしゃ 行 おこな 本 ほん 転写 てんしゃ 鋳型 いがた 鎖 くさり 非 ひ 鋳型 いがた 鎖 くさり 呼 よ
^ 『ウィーバー 分子生物学 ぶんしせいぶつがく 化学 かがく 同人 どうじん 著者 ちょしゃ 監訳 かんやく 者 しゃ 杉山 すぎやま 弘 ひろし
^ 『ウィーバー 分子生物学 ぶんしせいぶつがく
^ a b 『ウィーバー 分子生物学 ぶんしせいぶつがく
^ 『ウィーバー 分子生物学 ぶんしせいぶつがく
^ a b 『ウィーバー 分子生物学 ぶんしせいぶつがく
^ 『ワトソン 遺伝子 いでんし 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b 『ウィーバー 生化学 せいかがく
^ a b 『ウィーバー 生化学 せいかがく
^ a b c 『ウィーバー 分子生物学 ぶんしせいぶつがく
^ 『ストライヤー生化学 せいかがく 第 だい 版 はん 東京 とうきょう 化学 かがく 同人 どうじん 著者 ちょしゃ 監訳 かんやく 者 しゃ 入村 いりむら 達郎 たつお
^ a b c d 『遺伝子 いでんし 第 だい 版 はん 著者 ちょしゃ 訳者 やくしゃ 菊池 きくち 菊池 きくち 東京 とうきょう 化学 かがく 同人 どうじん
^ 『遺伝子 いでんし 第 だい 版 はん
^ a b 『遺伝子 いでんし 第 だい 版 はん
^ a b 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b 『ワトソン 遺伝子 いでんし 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b 『ウィーバー 生化学 せいかがく 第 だい 版 はん
^ a b 『ワトソン 遺伝子 いでんし 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ 『エッセンシャル遺伝子 いでんし 著者 ちょしゃ 訳者 やくしゃ 菊池 きくち 発行 はっこう 東京 とうきょう 化学 かがく 同人 どうじん
^ 『遺伝子 いでんし 第 だい 版 はん
^ 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b c 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b c d 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b 『ワトソン 遺伝子 いでんし 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ 『ワトソン 遺伝子 いでんし 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b c 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ the Annual Review of Genetics, Volume 34, 2000 by Annual Reviews
^ 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b c 『ウィーバー 分子生物学 ぶんしせいぶつがく 第 だい 版 はん
^ a b c 『エッセンシャル遺伝子 いでんし
活性 かっせい 調整 ちょうせい 分類 ぶんるい 動力 どうりょく 学 がく 種類 しゅるい