化学 かがく 上 じょう 未 み 解決 かいけつ 問題 もんだい
配列 はいれつ 環境 かんきょう 情報 じょうほう 基 もと ポリペプチド 配列 はいれつ 二 に 次 じ 構造 こうぞう 三 さん 次 じ 構造 こうぞう 四 よん 次 じ 構造 こうぞう 予測 よそく 可能 かのう 逆 ぎゃく タンパク質 たんぱくしつ 問題 もんだい 特定 とくてい 環境 かんきょう 条件下 じょうけんか 所与 しょよ 構造 こうぞう 採用 さいよう 配列 はいれつ 設計 せっけい 可能 かのう [1] [2] 近年 きんねん 小 しょう 球状 きゅうじょう タンパク質 たんぱくしつ 達成 たっせい [3]
フォールディング前 まえ 後 ご タンパク質 たんぱくしつ タンパク質 たんぱくしつ 結果 けっか タンパク質 たんぱくしつ フォールディング (英語 えいご Protein folding ) とは、タンパク質 たんぱくしつ 鎖 くさり 本来 ほんらい 三 さん 次元 じげん 構造 こうぞう 通常 つうじょう 生物 せいぶつ 学 がく 的 てき 機能 きのう コンホメーション (立体 りったい 構造 こうぞう 迅速 じんそく 再現 さいげん 性 せい 方法 ほうほう 獲得 かくとく 物理 ぶつり 的 てき ポリペプチド がランダムコイル からその特徴 とくちょう 的 てき 機能 きのう 的 てき 三 さん 次元 じげん 構造 こうぞう 折 お 物理 ぶつり 的 てき 過程 かてい [4] タンパク質 たんぱくしつ mRNA の配列 はいれつ アミノ酸 あみのさん 直 ちょく 鎖 くさり 翻訳 ほんやく 折 お 存在 そんざい 安定 あんてい 長続 ながつづ 立体 りったい 構造 こうぞう 欠 か 第 だい 図 ず 左側 ひだりがわ 鎖 くさり リボソーム で合成 ごうせい 過程 かてい 直 ちょく 鎖 くさり 三 さん 次元 じげん 構造 こうぞう 折 お 鎖 くさり 翻訳 ほんやく 中 ちゅう 始 はじ アミノ酸 あみのさん 互 たが 相互 そうご 作用 さよう 明確 めいかく 定義 ていぎ 三 さん 次元 じげん 構造 こうぞう 天然 てんねん 状態 じょうたい 知 し 折 お タンパク質 たんぱくしつ 図 ず 右側 みぎがわ 生成 せいせい 結果 けっか 生 しょう 三 さん 次元 じげん 構造 こうぞう アミノ酸 あみのさん 配列 はいれつ 一 いち 次 じ 構造 こうぞう アンフィンセンのドグマ ) によって決定 けってい [5]
タンパク質 たんぱくしつ 機能 きのう 発揮 はっき 正 ただ 三 さん 次元 じげん 構造 こうぞう 不可欠 ふかけつ 機能 きのう 性 せい タンパク質 たんぱくしつ 一部 いちぶ 折 お 状態 じょうたい [6] タンパク質 たんぱくしつ 動力 どうりょく 学 がく (英語 えいご 版 ばん 重要 じゅうよう 本来 ほんらい 構造 こうぞう 折 お 一般 いっぱん 不 ふ 活性 かっせい タンパク質 たんぱくしつ 生成 せいせい 場合 ばあい 誤 あやま 折 お タンパク質 たんぱくしつ 機能 きのう 改変 かいへん 毒性 どくせい 機能 きのう 性 せい 持 も 神経 しんけい 変性 へんせい 疾患 しっかん 他 た 疾患 しっかん 誤 あやま 折 お タンパク質 たんぱくしつ 形成 けいせい アミロイド 原 はら 線維 せんい 蓄積 ちくせき 起因 きいん 考 かんが [7] 多 おお アレルギー は、一部 いちぶ タンパク質 たんぱくしつ 正 まさ 折 お 原因 げんいん 免疫 めんえき 系 けい 特定 とくてい タンパク質 たんぱくしつ 構造 こうぞう 対 たい 抗体 こうたい 産 さん 生 せい 引 ひ 起 お [8]
タンパク質 たんぱくしつ 変性 へんせい 折 お 状態 じょうたい 折 お 状態 じょうたい 移行 いこう 調理 ちょうり 火傷 かしょう プロテオパチー 、その他 た 状況 じょうきょう 起 お
フォールディング・プロセスの所要 しょよう 時間 じかん 目的 もくてき タンパク質 たんぱくしつ 劇的 げきてき 異 こと 細胞 さいぼう 外 がい 調 しら 最 もっと 遅 おそ 折 お タンパク質 たんぱくしつ 主 おも プロリン 異性 いせい 化 か 折 お 数 すう 分 ふん 数時間 すうじかん 要 よう 完了 かんりょう 中 なか 間 あいだ 状態 じょうたい 通過 つうか 必要 ひつよう [9] 一方 いっぽう 長 なが アミノ酸 あみのさん 非常 ひじょう 小 ちい ドメイン タンパク質 たんぱくしつ 通常 つうじょう 回 かい 折 お [10] 時間 じかん 秒 びょう 一般 いっぱん 的 てき 非常 ひじょう 速 はや 既知 きち タンパク質 たんぱくしつ 反応 はんのう 数 すう 秒 びょう 以内 いない 完了 かんりょう [11]
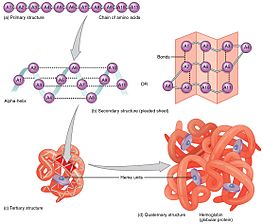
タンパク質 たんぱくしつ 過程 かてい [ 編集 へんしゅう ] タンパク質 たんぱくしつ 一 いち 次 じ 構造 こうぞう 直線 ちょくせん 的 てき アミノ酸 あみのさん 配列 はいれつ 本来 ほんらい 決定 けってい [12] 特定 とくてい アミノ酸 あみのさん 残 ざん 基 もと 鎖 くさり 内 ない 位置 いち タンパク質 たんぱくしつ 部分 ぶぶん 密接 みっせつ 折 お 重 かさ 三 さん 次元 じげん 構造 こうぞう 形成 けいせい 決定 けってい 要因 よういん アミノ酸 あみのさん 組成 そせい 配列 はいれつ 重要 じゅうよう [13] 本質 ほんしつ 的 てき 事実 じじつ 各 かく タンパク質 たんぱくしつ アミノ酸 あみのさん 配列 はいれつ 本来 ほんらい 構造 こうぞう 状態 じょうたい 到達 とうたつ 経路 けいろ 両方 りょうほう 指定 してい 情報 じょうほう 含 ふく 同 おな アミノ酸 あみのさん 配列 はいれつ 常 つね 同 おな 折 お [14] 類似 るいじ タンパク質 たんぱくしつ 環境 かんきょう 要因 よういん 異 こと 見 み 異 こと 方法 ほうほう 折 お
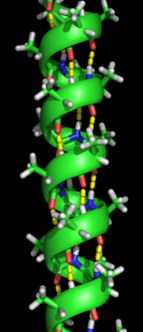
α あるふぁ 螺旋 らせん 形成 けいせい 主 しゅ 鎖 くさり 内 ない 水素 すいそ 結合 けつごう 有 ゆう 反 はん 平行 へいこう β べーた 二 に 次 じ 構造 こうぞう 形成 けいせい タンパク質 たんぱくしつ 本来 ほんらい 構造 こうぞう 取 と 最初 さいしょ 二 に 次 じ 構造 こうぞう 特徴 とくちょう α あるふぁ β べーた 知 し 構造 こうぞう ライナス・ポーリング によって最初 さいしょ 述 の 分子 ぶんし 内 ない (英語 えいご 版 ばん 水素 すいそ 結合 けつごう 安定 あんてい 化 か 急速 きゅうそく 折 お 分子 ぶんし 内 ない 水素 すいそ 結合 けつごう 形成 けいせい タンパク質 たんぱくしつ 安定 あんてい 性 せい 別 べつ 重要 じゅうよう 貢献 こうけん [15] α あるふぁ 主 しゅ 鎖 くさり 水素 すいそ 結合 けつごう 螺旋 らせん 状 じょう 形成 けいせい 右 みぎ 図 ず 参照 さんしょう [13] β べーた 水素 すいそ 結合 けつごう 形成 けいせい 主 しゅ 鎖 くさり 折 お 曲 ま 形成 けいせい 構造 こうぞう 左 ひだり 図 ず 参照 さんしょう 水素 すいそ 結合 けつごう ペプチド結合 けつごう のアミド水素 すいそ 酸素 さんそ 間 あいだ 逆 ぎゃく 平行 へいこう β べーた 平行 へいこう β べーた 存在 そんざい 平行 へいこう β べーた 形成 けいせい 傾斜 けいしゃ 水素 すいそ 結合 けつごう 比較 ひかく 逆 ぎゃく 平行 へいこう β べーた 方 ほう 理想 りそう 的 てき 度 ど 角度 かくど 水素 すいそ 結合 けつごう 形成 けいせい 水素 すいそ 結合 けつごう 安定 あんてい 性 せい 高 たか [13]
α あるふぁ β べーた 本質 ほんしつ 的 てき 両親 りょうしん 媒 なかだち 性 せい 親水 しんすい 性 せい 部分 ぶぶん 疎水 そすい 性 せい 部分 ぶぶん 含 ふく 二 に 次 じ 構造 こうぞう 性質 せいしつ タンパク質 たんぱくしつ 三 さん 次 じ 構造 こうぞう 形成 けいせい 助 たす 親水 しんすい 性 せい 側 がわ タンパク質 たんぱくしつ 取 と 囲 かこ 水溶 すいよう 性 せい 環境 かんきょう 面 めん 疎水 そすい 性 せい 側 がわ タンパク質 たんぱくしつ 疎水 そすい 性 せい 面 めん 起 お [16] 二 に 次 じ 構造 こうぞう 階層 かいそう 的 てき 三 さん 次 じ 構造 こうぞう 形成 けいせい 取 と 代 か タンパク質 たんぱくしつ 三 さん 次 じ 構造 こうぞう 形成 けいせい 疎水 そすい 性 せい 相互 そうご 作用 さよう 安定 あんてい 化 か システイン 残 ざん 基 もと 間 あいだ 形成 けいせい ジスルフィド結合 けつごう の形 かたち 共有 きょうゆう 結合 けつごう 存在 そんざい タンパク質 たんぱくしつ 三 さん 次 じ 構造 こうぞう 単一 たんいつ 鎖 くさり 含 ふく 折 お 鎖 くさり 追加 ついか 相互 そうご 作用 さよう 四 よん 次 じ 構造 こうぞう 形成 けいせい [17]
三 さん 次 じ 構造 こうぞう タンパク質 たんぱくしつ 四 よん 次 じ 構造 こうぞう 形成 けいせい 取 と 代 か 通常 つうじょう 折 お 集合 しゅうごう 会合 かいごう 含 ふく 換 いか 複数 ふくすう 鎖 くさり 相互 そうご 作用 さよう 完全 かんぜん 機能 きのう 四 よん 次 じ タンパク質 たんぱくしつ 形成 けいせい 可能 かのう 性 せい [13]
タンパク質 たんぱくしつ 推進 すいしん 力 りょく [ 編集 へんしゅう ] タンパク質 たんぱくしつ 構造 こうぞう 全 ぜん 形態 けいたい フォールディングは、主 おも 疎水 そすい 性 せい 相互 そうご 作用 さよう 分子 ぶんし 内 ない 水素 すいそ 結合 けつごう 形成 けいせい ファンデルワールス力 りょく によって導 みちび 自発 じはつ 的 てき 過程 かてい (英語 えいご 版 ばん 配 はい 座 ざ 対抗 たいこう 受 う [18] 多 おお 場合 ばあい 共 きょう 翻訳 ほんやく 的 てき 部分 ぶぶん 的 てき 翻訳 ほんやく 開始 かいし タンパク質 たんぱくしつ C末端 まったん 部分 ぶぶん 合成 ごうせい 間 あいだ 先 さき 翻訳 ほんやく タンパク質 たんぱくしつ N末端 まったん が折 お 始 はじ [19] タンパク質 たんぱくしつ 分子 ぶんし 生 なま 合成 ごうせい 中 ちゅう 生 なま 合成 ごうせい 後 のち 自発 じはつ 的 てき 折 お [20] 高分子 こうぶんし 自分 じぶん 自身 じしん 折 お 考 かんが 溶媒 ようばい 水 みず 脂質 ししつ 二 に 重層 じゅうそう [21] 塩 しお 濃度 のうど pH 、温度 おんど 補助 ほじょ 因子 いんし 分子 ぶんし シャペロン の存在 そんざい 依存 いぞん
タンパク質 たんぱくしつ 可能 かのう 曲 ま 角度 かくど 制限 せいげん 能力 のうりょく 制限 せいげん タンパク質 たんぱくしつ 許容 きょよう 角度 かくど ラマチャンドランプロット として知 し 二 に 次元 じげん 表 あらわ 許容 きょよう 回転 かいてん 角度 かくど 示 しめ [22]
疎水 そすい 性 せい 効果 こうか [ 編集 へんしゅう ] 疎水 そすい 性 せい 凝集 ぎょうしゅう (英語 えいご 版 ばん 右側 みぎがわ 疎水 そすい 性 せい アミノ酸 あみのさん 黒 くろ 球体 きゅうたい 表示 ひょうじ 中央 ちゅうおう 向 む 凝集 ぎょうしゅう 水性 すいせい 環境 かんきょう 遮蔽 しゃへい 状態 じょうたい タンパク質 たんぱくしつ 自発 じはつ 的 てき 反応 はんのう 細胞 さいぼう 内 ない 熱 ねつ 力学 りきがく 的 てき 有利 ゆうり タンパク質 たんぱくしつ 自発 じはつ 的 てき 反応 はんのう 知 し 負 まけ ギブス自由 じゆう 値 ね 必要 ひつよう タンパク質 たんぱくしつ 自由 じゆう エンタルピー とエントロピー に直接 ちょくせつ 関係 かんけい [13] 負 まけ 発生 はっせい タンパク質 たんぱくしつ 熱 ねつ 力学 りきがく 的 てき 有利 ゆうり 両方 りょうほう 有利 ゆうり
水分 すいぶん 子 こ 疎水 そすい 性 せい 溶質 ようしつ 近 ちか 整然 せいぜん 減少 げんしょう 水 みず 疎水 そすい 性 せい 側 がわ 鎖 くさり 数 かず 最小限 さいしょうげん 背後 はいご 重要 じゅうよう 推進 すいしん 力 りょく [23] 疎水 そすい 効果 こうか タンパク質 たんぱくしつ 疎水 そすい 性 せい 鎖 くさり タンパク質 たんぱくしつ 中心 ちゅうしん 部 ぶ 親水 しんすい 性 せい 環境 かんきょう 離 はな 凝集 ぎょうしゅう 現象 げんしょう [13] 水性 すいせい 環境 かんきょう 下 か 水分 すいぶん 子 こ タンパク質 たんぱくしつ 疎水 そすい 性 せい 領域 りょういき 側 がわ 鎖 くさり 周囲 しゅうい 凝集 ぎょうしゅう 秩序 ちつじょ 水分 すいぶん 子 こ 水 みず 殻 から 形成 けいせい 傾向 けいこう [24] 疎水 そすい 性 せい 領域 りょういき 中心 ちゅうしん 水分 すいぶん 子 こ 秩序 ちつじょ 系 けい 内 ない 秩序 ちつじょ 増大 ぞうだい 負 まけ 変化 へんか 寄与 きよ 系 けい 内 ない 減少 げんしょう 水分 すいぶん 子 こ 水分 すいぶん 子 こ 固定 こてい 疎水 そすい 性 せい 凝集 ぎょうしゅう (英語 えいご 版 ばん 疎水 そすい 性 せい 基 もと 内側 うちがわ 促進 そくしん 疎水 そすい 性 せい 凝集 ぎょうしゅう 秩序 ちつじょ 水分 すいぶん 子 こ 解放 かいほう 水 みず 破壊 はかい 介 かい 導入 どうにゅう [13] 球状 きゅうじょう 折 お タンパク質 たんぱくしつ 内 ない 相互 そうご 作用 さよう 多数 たすう 疎水 そすい 性 せい 基 もと 膨大 ぼうだい 蓄積 ちくせき 力 りょく 特 とく ロンドン分散 ぶんさん 力 りょく ) のため、折 お 後 ご タンパク質 たんぱくしつ 安定 あんてい 性 せい 大 おお 貢献 こうけん [13] 疎水 そすい 性 せい 効果 こうか 大 おお 疎水 そすい 性 せい 領域 りょういき 含 ふく 両親 りょうしん 媒 なかだち 性 せい 分子 ぶんし 含 ふく 水性 すいせい 媒体 ばいたい 存在 そんざい 場合 ばあい 熱 ねつ 力学 りきがく 推進 すいしん 力 りょく 存在 そんざい [25] 水素 すいそ 結合 けつごう 強 つよ 環境 かんきょう 依存 いぞん 疎水 そすい 性 せい 包 つつ 水素 すいそ 結合 けつごう 水性 すいせい 環境 かんきょう 水素 すいそ 結合 けつごう 天然 てんねん 状態 じょうたい 安定 あんてい 性 せい 寄与 きよ [26]
球状 きゅうじょう 折 お 持 も タンパク質 たんぱくしつ 疎水 そすい 性 せい アミノ酸 あみのさん 分布 ぶんぷ 一緒 いっしょ 化 か 一 いち 次 じ 配列 はいれつ 沿 そ 散在 さんざい 傾向 けいこう [27] [28] 天然 てんねん 変成 へんせい 傾向 けいこう 新生 しんせい de novo タンパク質 たんぱくしつ [29] [30] 一 いち 次 じ 配列 はいれつ 沿 そ 疎水 そすい 性 せい アミノ酸 あみのさん 化 か 逆 ぎゃく 示 しめ [31]
小型 こがた 真 ま 核 かく 生物 せいぶつ 熱 ねつ 質 しつ 例 れい 分子 ぶんし 英 えい molecular chaperone ) は、生体 せいたい 内 ない in vivo 他 た タンパク質 たんぱくしつ 正 まさ 折 お 役立 やくだ タンパク質 たんぱくしつ 一種 いっしゅ 細胞 さいぼう 内 ない 区画 くかく 存在 そんざい 鎖 くさり 相互 そうご 作用 さよう タンパク質 たんぱくしつ 本来 ほんらい 三 さん 次元 じげん 形成 けいせい 自体 じたい 補助 ほじょ タンパク質 たんぱくしつ 最終 さいしゅう 構造 こうぞう 含 ふく [32] 新生 しんせい 合成 ごうせい 場合 ばあい 助 たす [33] 分子 ぶんし 結合 けつごう 機能 きのう 経路 けいろ タンパク質 たんぱくしつ 不安定 ふあんてい 構造 こうぞう 安定 あんてい 化 か 補助 ほじょ タンパク質 たんぱくしつ 正 ただ 本来 ほんらい 構造 こうぞう 知 し 必要 ひつよう 情報 じょうほう 含 ふく 誤 あやま 折 お 構造 こうぞう 防 ふせ 機能 きのう [33] 実際 じっさい 本来 ほんらい 構造 こうぞう 向 む 経路 けいろ 関与 かんよ 個々 ここ 速度 そくど 増加 ぞうか 代 か 適切 てきせつ 中 なか 間 あいだ 体 たい 探索 たんさく 遅 おそ 可能 かのう 性 せい 鎖 くさり 不要 ふよう 凝集 ぎょうしゅう 減 へ 機能 きのう 鎖 くさり 正 ただ 効率 こうりつ 的 てき 経路 けいろ 提供 ていきょう [32] 触媒 しょくばい 混同 こんどう 経路 けいろ 遅 おそ 実際 じっさい 触媒 しょくばい 触媒 しょくばい 例 れい タンパク質 たんぱくしつ 異性 いせい 化 か 酵素 こうそ 異性 いせい 化 か 酵素 こうそ 結合 けつごう 形成 けいせい 立体 りったい 異性 いせい 体 たい 間 あいだ 相互 そうご 変換 へんかん 関与 かんよ 可能 かのう 性 せい [33] 生体 せいたい 内 ない タンパク質 たんぱくしつ 重要 じゅうよう 示 しめ タンパク質 たんぱくしつ 生物 せいぶつ 学 がく 的 てき 適切 てきせつ 状態 じょうたい 十分 じゅうぶん 効率 こうりつ 的 てき 適切 てきせつ 配列 はいれつ 必要 ひつよう 手助 てだす タンパク質 たんぱくしつ 提供 ていきょう [34] in vitro 行 おこな タンパク質 たんぱくしつ 実験 じっけん 実証 じっしょう 鎖 くさり 理論 りろん 的 てき 助 たす 本来 ほんらい 構造 こうぞう 折 お 意味 いみ [34] 非 ひ 効率 こうりつ 的 てき 遅 おそ 生物 せいぶつ 学 がく 的 てき 存在 そんざい 判明 はんめい 生体 せいたい 内 ない タンパク質 たんぱくしつ 必要 ひつよう 本来 ほんらい 構造 こうぞう 形成 けいせい 助 たす 役割 やくわり 加 くわ タンパク質 たんぱくしつ 輸送 ゆそう 分解 ぶんかい 外部 がいぶ 変性 へんせい 因子 いんし 変性 へんせい タンパク質 たんぱくしつ 正 ただ 本来 ほんらい 構造 こうぞう 再 さい 折 お 機会 きかい 与 あた 様々 さまざま 役割 やくわり 関与 かんよ 明 あき [35]
完全 かんぜん 変性 へんせい タンパク質 たんぱくしつ 三 さん 次 じ 構造 こうぞう 二 に 次 じ 構造 こうぞう 両方 りょうほう 欠 か ランダムコイル として存在 そんざい 特定 とくてい 条件下 じょうけんか 一部 いちぶ タンパク質 たんぱくしつ 再 さい 折 お 可能 かのう 性 せい 多 おお 場合 ばあい 変性 へんせい 不 ふ 可逆 かぎゃく 的 てき [36] 細胞 さいぼう 熱 ねつ 変性 へんせい 影響 えいきょう タンパク質 たんぱくしつ 守 まも 熱 ねつ 質 しつ 一種 いっしゅ 知 し 酵素 こうそ 用 もち 他 た タンパク質 たんぱくしつ 折 お 状態 じょうたい 維持 いじ 助 たす 熱 ねつ 質 しつ 細菌 さいきん 調査 ちょうさ 種 たね 発見 はっけん 非常 ひじょう 早 はや 段階 だんかい 進化 しんか 重要 じゅうよう 機能 きのう 持 も 示唆 しさ 一部 いちぶ タンパク質 たんぱくしつ 助 たす 借 か 他 た タンパク質 たんぱくしつ 相互 そうご 作用 さよう 中断 ちゅうだん 個々 ここ タンパク質 たんぱくしつ 分離 ぶんり 誤 あやま 折 お タンパク質 たんぱくしつ 展開 てんかい 本来 ほんらい 正 ただ 構造 こうぞう 限 かぎ 細胞 さいぼう 内 ない 全 まった 折 お [37] 機能 きのう 不溶性 ふようせい 凝集 ぎょうしゅう 体 たい 沈殿 ちんでん 防 ふせ 非常 ひじょう 重要 じゅうよう タンパク質 たんぱくしつ 変性 へんせい 天然 てんねん 状態 じょうたい 破壊 はかい 関与 かんよ 外部 がいぶ 要因 よういん 温度 おんど 外部 がいぶ 磁場 じば 電場 でんじょう 磁場 じば [38] 分子 ぶんし [39] タンパク質 たんぱくしつ 大 おお 影響 えいきょう 与 あた 可能 かのう 性 せい 空間 くうかん 制限 せいげん 閉 と 込 こ 含 ふく [40] 高 こう 濃度 のうど 溶質 ようしつ 極端 きょくたん pH 、機械 きかい 的 てき 力 ちから 化学 かがく 的 てき 変性 へんせい 剤 ざい 存在 そんざい 同様 どうよう タンパク質 たんぱくしつ 変性 へんせい 寄与 きよ 個々 ここ 要因 よういん 分類 ぶんるい 細胞 さいぼう 時 じ 高 こう 濃度 のうど 存在 そんざい 変性 へんせい タンパク質 たんぱくしつ 誤 あやま 折 お タンパク質 たんぱくしつ 新生 しんせい タンパク質 たんぱくしつ 適切 てきせつ 助 たす 示 しめ [32]
一部 いちぶ 条件下 じょうけんか タンパク質 たんぱくしつ 生化学 せいかがく 的 てき 機能 きのう 形 かたち 折 お 細胞 さいぼう 通常 つうじょう 生存 せいぞん 温度 おんど 範囲 はんい 高 たか 温度 おんど 低 ひく 温度 おんど 熱 ねつ 的 てき 不安定 ふあんてい タンパク質 たんぱくしつ 折 お 変性 へんせい 煮沸 しゃふつ 卵白 らんぱく 不透明 ふとうめい 理由 りゆう タンパク質 たんぱくしつ 熱 ねつ 安定 あんてい 性 せい 一定 いってい 限 かぎ 例 たと 超 ちょう 好 こう 熱性 ねっせい 細菌 さいきん 高温 こうおん 生育 せいいく 確認 かくにん [41] 重要 じゅうよう タンパク質 たんぱくしつ タンパク質 たんぱくしつ 集合 しゅうごう 体 たい 完全 かんぜん 補 ほ 体 たい 温度 おんど 以上 いじょう 安定 あんてい 必要 ひつよう
大腸菌 だいちょうきん バクテリオファージT4 の宿主 しゅくしゅ タンパク質 たんぱくしつ 大腸菌 だいちょうきん シャペロンタンパク質 しつ GroES (英語 えいご 版 ばん 機能 きのう 的 てき 相 あい 同 どう 見 み 感染 かんせん 時 じ ウイルス 粒子 りゅうし 組 く 立 た 置 お 換 か [42] 同様 どうよう GroEL (英語 えいご 版 ばん 安定 あんてい 複 ふく 合体 がったい 形成 けいせい 質 しつ in vivo でのフォールディングおよび組 く 立 た 絶対 ぜったい 的 てき 必要 ひつよう [42]
タンパク質 たんぱくしつ 誤 あやま 神経 しんけい 変性 へんせい 疾患 しっかん [ 編集 へんしゅう ] タンパク質 たんぱくしつ 通常 つうじょう 天然 てんねん 状態 じょうたい 得 え 場合 ばあい 誤 あやま 折 お 考 かんが アミノ酸 あみのさん 配列 はいれつ 突然変異 とつぜんへんい 外部 がいぶ 要因 よういん 通常 つうじょう 混乱 こんらん 原因 げんいん 可能 かのう 性 せい [43] タンパク質 たんぱくしつ 通常 つうじょう β べーた 構造 こうぞう 知 し 超 ちょう 分子 ぶんし 配列 はいれつ 組織 そしき 化 か β べーた 含 ふく β べーた 豊富 ほうふ 含 ふく 集合 しゅうごう 体 たい 非常 ひじょう 安定 あんてい 極 きわ 不溶性 ふようせい 一般 いっぱん タンパク質 たんぱくしつ 分解 ぶんかい 対 たい 耐 たい 性 せい [44] フィブリル 状 じょう 集合 しゅうごう 体 たい 構造 こうぞう 的 てき 安定 あんてい 性 せい β べーた 間 あいだ 主 しゅ 鎖 くさり 水素 すいそ 結合 けつごう 形成 けいせい タンパク質 たんぱくしつ 間 あいだ 広範 こうはん 相互 そうご 作用 さよう [44] タンパク質 たんぱくしつ 他 た タンパク質 たんぱくしつ 凝集 ぎょうしゅう 体 たい オリゴマー へのさらなるミスフォールディングや蓄積 ちくせき 引 ひ 起 お 可能 かのう 性 せい 細胞 さいぼう 内 ない 凝集 ぎょうしゅう タンパク質 たんぱくしつ 上昇 じょうしょう 変性 へんせい 疾患 しっかん 細胞 さいぼう 死 し 引 ひ 起 お 可能 かのう 性 せい アミロイド 様 よう 構造 こうぞう 形成 けいせい [43] 分子 ぶんし 間 あいだ 水素 すいそ 結合 けつごう 含 ふく 線維状 せんいじょう 構造 こうぞう 極 きわ 不溶性 ふようせい 転換 てんかん タンパク質 たんぱくしつ 集合 しゅうごう 体 たい 作 つく [43] 経路 けいろ 凝集 ぎょうしゅう 前 まえ タンパク質 たんぱくしつ 分解 ぶんかい 十分 じゅうぶん 効率 こうりつ 得 え 場合 ばあい タンパク質 たんぱくしつ 互 たが 相互 そうご 作用 さよう 構造 こうぞう 化 か 凝集 ぎょうしゅう 体 たい 形成 けいせい 分子 ぶんし 間 あいだ 相互 そうご 作用 さよう 通 つう 毒性 どくせい 獲得 かくとく 可能 かのう 性 せい [43]
凝集 ぎょうしゅう タンパク質 たんぱくしつ クロイツフェルト・ヤコブ病 びょう 、牛 うし 海綿 かいめん 状 じょう 脳症 のうしょう 狂牛病 きょきょうぎゅうびょう プリオン 関連 かんれん 疾患 しっかん アルツハイマー病 びょう および家族 かぞく 性 せい (英語 えいご 版 ばん 心筋 しんきん 症 しょう 多 た 神経症 しんけいしょう アミロイド 関連 かんれん 疾患 しっかん [45] ハンチントン病 びょう およびパーキンソン病 びょう などの細胞 さいぼう 内 ない 凝集 ぎょうしゅう 性 せい 疾患 しっかん 関連 かんれん [7] [46] 加 か 齢 よわい 性 せい 変性 へんせい 疾患 しっかん 不溶性 ふようせい 細胞 さいぼう 外 がい 凝集 ぎょうしゅう 体 たい β べーた アミロイド 原 はら 線維 せんい 含 ふく 細胞 さいぼう 内 ない 封入 ふうにゅう 体 たい 質 しつ 凝集 ぎょうしゅう 関連 かんれん 凝集 ぎょうしゅう 体 たい 原因 げんいん 単 たん タンパク質 たんぱくしつ 恒常 こうじょう 性 せい 喪失 そうしつ 合成 ごうせい 凝集 ぎょうしゅう タンパク質 たんぱくしつ 代謝 たいしゃ 回転 かいてん 反映 はんえい 完全 かんぜん 明 あき 最近 さいきん 欧州 おうしゅう 医薬品 いやくひん 庁 ちょう トランスサイレチン アミロイド疾患 しっかん 治療 ちりょう タファミディス (英語 えいご 版 ばん 四 よん 量 りょう 体 からだ 動態 どうたい 安定 あんてい 化 か 剤 ざい 使用 しよう 承認 しょうにん 疾患 しっかん 原 げん 線維 せんい 形成 けいせい 原 はら 線維 せんい 自体 じたい 有 ゆう 糸 いと 分裂 ぶんれつ 後 ご 組織 そしき 変性 へんせい 引 ひ 起 お 示唆 しさ [47] 機能 きのう 過度 かど 分解 ぶんかい アンチトリプシン 関連 かんれん 肺気腫 はいきしゅ 嚢胞 のうほう 性 せい 線維 せんい 症 しょう リソソーム蓄積 ちくせき 症 しょう などの多 おお プロテオパチー 疾患 しっかん 引 ひ 起 お 機能 きのう 喪失 そうしつ 障害 しょうがい 根源 こんげん 後者 こうしゃ 疾患 しっかん 修正 しゅうせい タンパク質 たんぱくしつ 補充 ほじゅう 療法 りょうほう 歴史 れきし 的 てき 使用 しよう 新 あら 薬理 やくり (英語 えいご 版 ばん 使用 しよう 変異 へんい タンパク質 たんぱくしつ 折 お 機能 きのう 状態 じょうたい 挙 あ
タンパク質 たんぱくしつ 研究 けんきゅう 実験 じっけん 技術 ぎじゅつ [ 編集 へんしゅう ] タンパク質 たんぱくしつ 関 かん 推論 すいろん 突然変異 とつぜんへんい 研究 けんきゅう (英語 えいご 版 ばん を通 つう 行 おこな タンパク質 たんぱくしつ 研究 けんきゅう 実験 じっけん 技術 ぎじゅつ 通常 つうじょう タンパク質 たんぱくしつ 段階 だんかい 的 てき (英語 えいご 版 ばん 標準 ひょうじゅん 的 てき 非 ひ 結晶 けっしょう 学 がく 的 てき 技術 ぎじゅつ 用 もち 変化 へんか 観察 かんさつ 依存 いぞん
X線 せん 結晶 けっしょう 構造 こうぞう 解析 かいせき [ 編集 へんしゅう ] X線 せん 結晶 けっしょう 構造 こうぞう 解析 かいせき のステップX線 せん 結晶 けっしょう 構造 こうぞう 解析 かいせき は、折 お タンパク質 たんぱくしつ 三 さん 次元 じげん 構造 こうぞう 解読 かいどく 効率 こうりつ 的 てき 重要 じゅうよう 方法 ほうほう 一 ひと [48] 線 せん 結晶 けっしょう 構造 こうぞう 解析 かいせき 行 おこな 対象 たいしょう タンパク質 たんぱくしつ 結晶 けっしょう 格子 こうし 内 ない 配置 はいち 必要 ひつよう タンパク質 たんぱくしつ 結晶 けっしょう 格子 こうし 内 ない 配置 はいち 結晶 けっしょう 化 か 適 てき 溶媒 ようばい 用意 ようい 溶液 ようえき 中 ちゅう 過飽和 かほうわ 状態 じょうたい 純粋 じゅんすい タンパク質 たんぱくしつ 得 え 溶液 ようえき 中 ちゅう 結晶 けっしょう 析出 せきしゅつ 必要 ひつよう [49] タンパク質 たんぱくしつ 結晶 けっしょう 化 か 線 せん 結晶 けっしょう 格子 こうし 介 かい 集中 しゅうちゅう 回折 かいせつ 様々 さまざま 方向 ほうこう 外側 そとがわ 向 む 発射 はっしゃ 出射 しゅっしゃ 内包 ないほう タンパク質 たんぱくしつ 特定 とくてい 三 さん 次元 じげん 構成 こうせい 相関 そうかん 線 せん タンパク質 たんぱくしつ 結晶 けっしょう 格子 こうし 内 ない 個々 ここ 原子 げんし 取 と 囲 かこ 電子 でんし 雲 くも 特異 とくい 的 てき 相互 そうご 作用 さよう 識別 しきべつ 可能 かのう 回折 かいせつ 生成 せいせい [16] 電子 でんし 密度 みつど 雲 くも 線 せん 振幅 しんぷく 関連付 かんれんづ 読 よ 取 と 方法 ほうほう 複雑 ふくざつ 位相 いそう 位相 いそう 角 かく 仮定 かてい 導 みちび [50] フーリエ変換 へんかん という数学 すうがく 的 てき 基礎 きそ 確立 かくりつ 関係 かんけい 位相 いそう 問題 もんだい 回折 かいせつ 予測 よそく 非常 ひじょう 困難 こんなん [16] 多重 たじゅう 同型 どうけい 置換 ちかん (英語 えいご 版 ばん 新 あたら 方法 ほうほう 重金属 じゅうきんぞく 存在 そんざい 利用 りよう 線 せん 予測 よそく 可能 かのう 方法 ほうほう 回折 かいせつ 関与 かんよ 変数 へんすう 数 かず 減 へ 位相 いそう 問題 もんだい 解決 かいけつ [48]
蛍光 けいこう 分光 ぶんこう 法 ほう [ 編集 へんしゅう ] 蛍光 けいこう 分光 ぶんこう 法 ほう タンパク質 たんぱくしつ 折 お 状態 じょうたい 調 しら 高 こう 感度 かんど 手法 しゅほう アミノ酸 あみのさん 固有 こゆう 蛍光 けいこう 特性 とくせい 持 も 量子 りょうし 収 おさむ 率 りつ 高 たか 良好 りょうこう 蛍光 けいこう 得 え 実験 じっけん 的 てき 使用 しよう 共 とも 波長 はちょう 励起 れいき 対 たい 波長 はちょう 励起 れいき 芳香 ほうこう 族 ぞく 性 せい 残 ざん 基 もと タンパク質 たんぱくしつ 疎水 そすい 性 せい タンパク質 たんぱくしつ 間 あいだ 界面 かいめん 質 しつ 間 あいだ 界面 かいめん 完全 かんぜん 部分 ぶぶん 的 てき 埋 うず 無 む 極性 きょくせい 環境 かんきょう 残 ざん 基 もと 高 たか 量子 りょうし 収 おさむ 率 りつ 持 も 高 たか 蛍光 けいこう 強度 きょうど 示 しめ タンパク質 たんぱくしつ 次 じ 構造 こうぞう 次 じ 構造 こうぞう 破壊 はかい 側 がわ 鎖 くさり 溶媒 ようばい 親水 しんすい 性 せい 環境 かんきょう 量子 りょうし 収 おさむ 率 りつ 低下 ていか 低 ひく 蛍光 けいこう 強度 きょうど 残 ざん 基 もと 最大 さいだい 蛍光 けいこう 発光 はっこう 波長 はちょう 環境 かんきょう 依存 いぞん
蛍光 けいこう 分光 ぶんこう 法 ほう 変性 へんせい 剤 ざい 値 ね 関数 かんすう 蛍光 けいこう 発光 はっこう 強度 きょうど 最大 さいだい 発光 はっこう 波長 はちょう 変動 へんどう 測定 そくてい タンパク質 たんぱくしつ 平衡 へいこう (英語 えいご 版 ばん 特徴付 とくちょうづ 使用 しよう [51] [52] 変性 へんせい 剤 ざい 化学 かがく 分子 ぶんし 尿素 にょうそ 塩酸 えんさん 温度 おんど 圧力 あつりょく 互 たが 異 こと 離散 りさん 的 てき タンパク質 たんぱくしつ 状態 じょうたい 天然 てんねん 状態 じょうたい 中間 なかま 状態 じょうたい 状態 じょうたい 間 あいだ 平衡 へいこう 変性 へんせい 剤 ざい 値 ね 依存 いぞん 平衡 へいこう 混合 こんごう 物 ぶつ 全体 ぜんたい 的 てき 蛍光 けいこう 値 ね 依存 いぞん 全体 ぜんたい 的 てき タンパク質 たんぱくしつ 変性 へんせい 剤 ざい 値 ね 関連付 かんれんづ 得 え [53] [54] 平衡 へいこう 中 なか 間 あいだ 体 たい 検出 けんしゅつ 識別 しきべつ 可能 かのう 一般 いっぱん 的 てき 方程式 ほうていしき 開発 かいはつ タンパク質 たんぱくしつ 平衡 へいこう 特徴 とくちょう 熱 ねつ 力学 りきがく 的 てき 三 さん 量 りょう 体 からだ 潜在 せんざい 的 てき 四 よん 量 りょう 体 からだ 得 え [51] 蛍光 けいこう 分光 ぶんこう 法 ほう ストップフロー (英語 えいご 版 ばん 高速 こうそく 混合 こんごう 装置 そうち 組 く 合 あ タンパク質 たんぱくしつ 動態 どうたい 測定 そくてい [55] シェブロンプロット (英語 えいご 版 ばん 生成 せいせい 値 ち 分析 ぶんせき (英語 えいご 版 ばん を導出 どうしゅつ
円 えん 偏 へん 光 こう 二色 にしき 性 せい [ 編集 へんしゅう ] 円 えん 偏 へん 光 こう 二色 にしき 性 せい タンパク質 たんぱくしつ 研究 けんきゅう 最 もっと 一般 いっぱん 的 てき 基本 きほん 的 てき 一 ひと 円 えん 二 に 色 しょく 性 せい 分光 ぶんこう 法 ほう 円 えん 偏 へん 光 こう 吸収 きゅうしゅう 測定 そくてい タンパク質 たんぱくしつ α あるふぁ β べーた 構造 こうぞう 不 ふ 斉 ひとし 光 ひかり 吸収 きゅうしゅう 光 ひかり 吸収 きゅうしゅう タンパク質 たんぱくしつ 折 お 度合 どあ 機能 きのう 技術 ぎじゅつ 変性 へんせい 剤 ざい 濃度 のうど 温度 おんど 関数 かんすう 吸収 きゅうしゅう 変化 へんか 測定 そくてい タンパク質 たんぱくしつ 平衡 へいこう (英語 えいご 版 ばん 測定 そくてい 使用 しよう 変性 へんせい 溶解 ようかい タンパク質 たんぱくしつ 値 ね 変性 へんせい 剤 ざい 依存 いぞん 性 せい 同様 どうよう 自由 じゆう 測定 そくてい 温度 おんど 溶解 ようかい タンパク質 たんぱくしつ 変性 へんせい 温度 おんど (英語 えいご 版 ばん 測定 そくてい [51] 蛍光 けいこう 分光 ぶんこう 法 ほう 関 かん 円 えん 二 に 色 しょく 性 せい 分光 ぶんこう 法 ほう 高速 こうそく 混合 こんごう 装置 そうち 組 く 合 あ タンパク質 たんぱくしつ 動態 どうたい 測定 そくてい シェブロンプロット (英語 えいご 版 ばん 生成 せいせい
タンパク質 たんぱくしつ 振動 しんどう 円 えん 二 に 色 しょく 性 せい [ 編集 へんしゅう ] 最近 さいきん 開発 かいはつ タンパク質 たんぱくしつ 振動 しんどう 円 えん 二 に 色 しょく 性 せい (英語 えいご 版 ばん 技術 ぎじゅつ 現在 げんざい フーリエ変換 へんかん (FT)機器 きき 用 もち 非常 ひじょう 大 おお タンパク質 たんぱくしつ 分子 ぶんし 溶液 ようえき 中 ちゅう タンパク質 たんぱくしつ 構造 こうぞう 決定 けってい 強力 きょうりょく 手段 しゅだん 提供 ていきょう タンパク質 たんぱくしつ 研究 けんきゅう タンパク質 たんぱくしつ 結晶 けっしょう X線 せん 回折 かいせつ 、重水 じゅうすい 2 O)中 ちゅう タンパク質 たんぱくしつ 溶液 ようえき FT-IR データ、または第 だい 一 いち 原理 げんり 量子 りょうし 計算 けいさん 組 く 合 あ 円 えん 偏 へん 光 こう 二色 にしき 性 せい 得 え 明確 めいかく 構造 こうぞう 決定 けってい 提供 ていきょう [要 よう 出典 しゅってん 。
タンパク質 たんぱくしつ 核 かく 磁気 じき 共鳴 きょうめい 分光 ぶんこう 法 ほう [ 編集 へんしゅう ] タンパク質 たんぱくしつ NMR分光 ぶんこう 法 ほう を使用 しよう 日常 にちじょう 的 てき 研究 けんきゅう 例 たと 天然 てんねん 状態 じょうたい タンパク質 たんぱくしつ 主 しゅ 鎖 くさり 水素 すいそ 重水素 じゅうすいそ 交換 こうかん (英語 えいご 版 ばん を監視 かんし タンパク質 たんぱくしつ 残 ざん 基 もと 固有 こゆう 安定 あんてい 性 せい 全体 ぜんたい 的 てき 安定 あんてい 性 せい 両方 りょうほう 提供 ていきょう [56]
二 に 重 じゅう 偏 へん 光 こう 干渉 かんしょう 法 ほう [ 編集 へんしゅう ] 二 に 重 じゅう 偏 へん 光 こう 干渉 かんしょう 法 ほう 分子 ぶんし 層 そう 光学 こうがく 特性 とくせい 測定 そくてい 表面 ひょうめん 技術 ぎじゅつ タンパク質 たんぱくしつ 特徴 とくちょう 使用 しよう 場合 ばあい タンパク質 たんぱくしつ 単 たん 層 そう 全体 ぜんたい 的 てき 密度 みつど 分解能 ぶんかいのう 測定 そくてい コンホメーション を測定 そくてい [57] タンパク質 たんぱくしつ 速度 そくど 論 ろん 測定 そくてい Hz へるつ 遅 おそ 限 かぎ 円 えん 偏 へん 光 こう 二色 にしき 性 せい 同様 どうよう 刺激 しげき 変性 へんせい 剤 ざい 温度 おんど 可能 かのう 性 せい
高 こう 時間 じかん 分解能 ぶんかいのう 研究 けんきゅう [ 編集 へんしゅう ] タンパク質 たんぱくしつ 研究 けんきゅう 高速 こうそく 時間 じかん 分解 ぶんかい 技術 ぎじゅつ 開発 かいはつ 近年 きんねん 大 おお 進展 しんてん 実験 じっけん 者 しゃ 折 お タンパク質 たんぱくしつ 迅速 じんそく 誘発 ゆうはつ 結果 けっか 生 しょう タンパク質 たんぱくしつ 動力 どうりょく 学 がく (英語 えいご 版 ばん 観察 かんさつ 使用 しよう 高速 こうそく 化 か 技術 ぎじゅつ 中性子 ちゅうせいし 散乱 さんらん [58] 超 ちょう 高速 こうそく 溶液 ようえき 混合 こんごう 光化学 こうかがく 的 てき 手法 しゅほう 温度 おんど (英語 えいご 版 ばん 分光 ぶんこう 法 ほう 技術 ぎじゅつ 開発 かいはつ 貢献 こうけん 多 おお 科学 かがく 者 しゃ 中 なか
タンパク質 たんぱくしつ 分解 ぶんかい [ 編集 へんしゅう ] タンパク質 たんぱくしつ 分解 ぶんかい 広範囲 こうはんい 溶液 ようえき 条件下 じょうけんか 例 れい 高速 こうそく 並列 へいれつ タンパク質 たんぱくしつ 分解 ぶんかい (英語 えいご 版 ばん 画 が 分 ぶん 探索 たんさく 日常 にちじょう 的 てき 使用 しよう [59] [60]
単一 たんいつ 分子 ぶんし 力 りょく 分光 ぶんこう 法 ほう [ 編集 へんしゅう ] 単 たん 離 はな タンパク質 たんぱくしつ 持 も タンパク質 たんぱくしつ 機構 きこう 理解 りかい 光 ひかり 単一 たんいつ 分子 ぶんし 力 りょく 技術 ぎじゅつ 用 もち [61] 光 ひかり 単一 たんいつ タンパク質 たんぱくしつ 分子 ぶんし 末端 まったん 末端 まったん 引 ひ 伸 の 展開 てんかい 後 ご 研究 けんきゅう 使用 しよう [62] 手法 しゅほう 単一 たんいつ 分子 ぶんし 率 りつ 測定 そくてい 例 たと 光 ひかり 最近 さいきん 血液 けつえき 凝固 ぎょうこ 関与 かんよ タンパク質 たんぱくしつ 研究 けんきゅう 応用 おうよう ヴォン・ヴィレブランド因子 いんし (von Willebrand factor (vWF)) は、血液 けつえき 凝固 ぎょうこ 不可欠 ふかけつ 役割 やくわり 持 も タンパク質 たんぱくしつ 単一 たんいつ 分子 ぶんし 光 ひかり 測定 そくてい 使用 しよう 結合 けつごう 血液 けつえき 中 ちゅう 断 だん 力 りょく 働 はたら 発見 はっけん 断 だん 力 りょく 速度 そくど 存在 そんざい 下 か 劇的 げきてき 向上 こうじょう [63] 最近 さいきん 単純 たんじゅん SH3ドメイン が、力 ちから 受 う 複数 ふくすう 経路 けいろ 明 あき [64]
ビオチン標識 ひょうしき 未 ひつじ 質 しつ 状態 じょうたい 特異 とくい 的 てき 細胞 さいぼう 可能 かのう 標識 ひょうしき 予測 よそく 天然 てんねん 変成 へんせい タンパク質 たんぱくしつ 偏 かたよ 示 しめ [65]
タンパク質 たんぱくしつ 計算 けいさん 科学 かがく 的 てき 研究 けんきゅう [ 編集 へんしゅう ] タンパク質 たんぱくしつ 計算 けいさん 科学 かがく 的 てき 研究 けんきゅう タンパク質 たんぱくしつ 安定 あんてい 性 せい 速度 そくど 論 ろん 構造 こうぞう 予測 よそく 関連 かんれん 主要 しゅよう 側面 そくめん 含 ふく 以下 いか 最近 さいきん タンパク質 たんぱくしつ 利用 りよう 可能 かのう 計算 けいさん 手法 しゅほう [66]
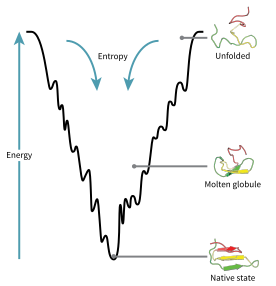
レヴィンタールのパラドックス [ 編集 へんしゅう ] 1969年 ねん サイラス・レヴィンタール (英語 えいご 版 ばん 折 お 鎖 くさり 自由 じゆう 度 ど 非常 ひじょう 大 おお タンパク質 たんぱくしつ 分子 ぶんし 天文学 てんもんがく 的 てき 数 かず 起 お 持 も 着目 ちゃくもく 彼 かれ 論文 ろんぶん 中 なか 300 または10143 という推定 すいてい [67] レヴィンタールのパラドックス は、タンパク質 たんぱくしつ 起 お 順次 じゅんじ 折 お 場合 ばあい 高速 こうそく ナノ秒 びょう またはピコ秒 びょう スケール) でサンプリングされたとしても、天文学 てんもんがく 的 てき 量 りょう 時間 じかん 観察 かんさつ 基 もと 思考 しこう 実験 じっけん [68] タンパク質 たんぱくしつ 速 はや 折 お 観測 かんそく 基 もと 探索 たんさく 発生 はっせい タンパク質 たんぱくしつ 一連 いちれん 準 じゅん 安定 あんてい 中間 ちゅうかん 状態 じょうたい 経 へ 折 お 提案 ていあん
タンパク質 たんぱくしつ 地形 ちけい [ 編集 へんしゅう ] 折 お 鎖 くさり 本来 ほんらい 構造 こうぞう 取 と 漏斗 ろうと フォールディング中 ちゅう タンパク質 たんぱくしつ 配 はい 位 い 空間 くうかん (英語 えいご 版 ばん エネルギー地形 ちけい (energy landscape) として可視 かし 化 か Peter Wolynes (英語 えいご 版 ばん タンパク質 たんぱくしつ 最小 さいしょう 原理 げんり 従 したが 自然 しぜん 進化 しんか タンパク質 たんぱくしつ 時 じ 地形 ちけい 最適 さいてき 化 か [69] 自然 しぜん タンパク質 たんぱくしつ 折 お 状態 じょうたい 十分 じゅうぶん 安定 あんてい アミノ酸 あみのさん 配列 はいれつ 選択 せんたく 意味 いみ 折 お 状態 じょうたい 獲得 かくとく 十分 じゅうぶん 高速 こうそく 自然 しぜん タンパク質 たんぱくしつ 減 へ タンパク質 たんぱくしつ 地形 ちけい 局所 きょくしょ 的 てき 最小 さいしょう 値 ち 存在 そんざい 程度 ていど 今 いま 残 のこ
これらの進化 しんか 的 てき 選択 せんたく 配列 はいれつ 結果 けっか タンパク質 たんぱくしつ 天然 てんねん 状態 じょうたい 向 む 漏斗 ろうと 状 じょう 地形 ちけい José Onuchic (英語 えいご 版 ばん 造語 ぞうご [70] 持 も 一般 いっぱん 的 てき 考 かんが フォールディング・ファンネル (英語 えいご 版 ばん 地形 ちけい タンパク質 たんぱくしつ 単一 たんいつ 限定 げんてい 多数 たすう 経路 けいろ 中 ちゅう 間 あいだ 体 たい 介 かい 天然 てんねん 状態 じょうたい 理論 りろん タンパク質 たんぱくしつ 計算 けいさん 格子 こうし タンパク質 たんぱくしつ (英語 えいご 版 ばん 実験 じっけん 的 てき 研究 けんきゅう 両方 りょうほう 支持 しじ [69] タンパク質 たんぱくしつ 構造 こうぞう 予測 よそく タンパク質 たんぱくしつ 構造 こうぞう 設計 せっけい 方法 ほうほう 改善 かいぜん 使用 しよう [69] 平準 へいじゅん 化 か 自由 じゆう 地形 ちけい タンパク質 たんぱくしつ 説明 せつめい 熱 ねつ 力学 りきがく 第 だい 法則 ほうそく 合致 がっち [71] 物理 ぶつり 的 てき 地形 ちけい 地理 ちり 的 てき 地形 ちけい 単 たん 最大 さいだい 値 ち 鞍点 あんてん 最小 さいしょう 値 ち 持 も 可視 かし 化 か 可能 かのう 曲面 きょくめん 全 ぜん 曲面 きょくめん 観点 かんてん 考 かんが 誤解 ごかい 招 まね 可能 かのう 性 せい 妥当 だとう 記述 きじゅつ 実際 じっさい 多様 たよう 体 たい 様々 さまざま 複雑 ふくざつ 位相 いそう 形態 けいたい 可能 かのう 性 せい 高 こう 次元 じげん 位相 いそう 空間 くうかん [72]
折 お 鎖 くさり 一番 いちばん 上 じょう 位置 いち 折 お 数 かず 最 もっと 多 おお 状態 じょうたい 高 こう 高 たか 地形 ちけい 初期 しょき 可能 かのう 性 せい 多数 たすう 示 しめ 可能 かのう 単一 たんいつ 天然 てんねん 状態 じょうたい 可能 かのう 多 おお 経路 けいろ 明 あき 同 おな 正確 せいかく タンパク質 たんぱくしつ 異 こと 分子 ぶんし 同 おな 天然 てんねん 構造 こうぞう 到達 とうたつ 限 かぎ 異 こと 経路 けいろ 異 こと 低 てい 中間 なかま 体 たい 探 さが 場合 ばあい [73] 異 こと 経路 けいろ 各 かく 経路 けいろ 熱 ねつ 力学 りきがく 的 てき 有利 ゆうり 性 せい 応 おう 異 こと 利用 りよう 頻度 ひんど 持 も 可能 かのう 性 せい 経路 けいろ 他 た 経路 けいろ 熱 ねつ 力学 りきがく 的 てき 有利 ゆうり 分 わ 場合 ばあい 本来 ほんらい 構造 こうぞう 追求 ついきゅう 頻繁 ひんぱん 使用 しよう 可能 かのう 性 せい 高 たか 意味 いみ [73] タンパク質 たんぱくしつ 折 お 始 はじ 常 つね 以前 いぜん 熱 ねつ 力学 りきがく 的 てき 有利 ゆうり 構造 こうぞう 求 もと 通過 つうか 続 つづ 二 に 次 じ 構造 こうぞう 形成 けいせい タンパク質 たんぱくしつ 内 ない 安定 あんてい 性 せい 向上 こうじょう 強 つよ 示 しめ 骨格 こっかく 想定 そうてい 二 に 次 じ 構造 こうぞう 一 ひと 組 く 合 あ 最低 さいてい 持 も タンパク質 たんぱくしつ 天然 てんねん 状態 じょうたい 存在 そんざい [73] 折 お 開始 かいし 形成 けいせい 最初 さいしょ 構造 こうぞう 中 なか α あるふぁ β べーた α あるふぁ 秒 びょう 形成 けいせい β べーた 秒 びょう 形成 けいせい [32]
エネルギー・ファンネル地形 ちけい 特定 とくてい タンパク質 たんぱくしつ 遷移 せんい 状態 じょうたい 見 み 鞍点 あんてん 存在 そんざい [32] 図 ず 遷移 せんい 状態 じょうたい タンパク質 たんぱくしつ 最終 さいしゅう 的 てき 本来 ほんらい 構造 こうぞう 想定 そうてい 場合 ばあい タンパク質 たんぱくしつ 分子 ぶんし タンパク質 たんぱくしつ 最初 さいしょ 遷移 せんい 状態 じょうたい 通過 つうか 本来 ほんらい 構造 こうぞう [32] 遷移 せんい 状態 じょうたい 単 たん 別 べつ 中 なか 間 あいだ 段階 だんかい 天然 てんねん 状態 じょうたい 変化 へんか 形 がた 未熟 みじゅく 形 かたち 呼 よ [74] 遷移 せんい 状態 じょうたい 律 りつ 速 そく 示 しめ 本来 ほんらい 高 たか 状態 じょうたい 存在 そんざい 本来 ほんらい 構造 こうぞう 極 きわ 類似 るいじ 遷移 せんい 状態 じょうたい 中 なか タンパク質 たんぱくしつ 折 お 核 かく 足場 あしば 構造 こうぞう 存在 そんざい 核 かく 上 うえ 構造 こうぞう 段階 だんかい 的 てき 完成 かんせい 凝縮 ぎょうしゅく 核 かく 形成 けいせい 呼 よ 形成 けいせい [74]
タンパク質 たんぱくしつ [ 編集 へんしゅう ] Folding@home は、ここに示 しめ マルコフ状態 じょうたい を使用 しよう タンパク質 たんぱくしつ 初期 しょき 状 じょう 状態 じょうたい 左 ひだり 自然 しぜん 三 さん 次元 じげん 構造 こうぞう 右 みぎ 凝集 ぎょうしゅう 取 と 起 お 形状 けいじょう 経路 けいろ 化 か 計算 けいさん タンパク質 たんぱくしつ 構造 こうぞう 予測 よそく デ・ノボ (de novo ) または第 だい 一 いち 原理 げんり ab initio ) 的 てき 手法 しゅほう タンパク質 たんぱくしつ 実験 じっけん 的 てき 研究 けんきゅう 関連 かんれん 厳密 げんみつ 区別 くべつ 分子 ぶんし 動力 どうりょく 学 がく 法 ほう タンパク質 たんぱくしつ 動力 どうりょく 学 がく イン・シリコ (in silico) で研究 けんきゅう 重要 じゅうよう [75] 最初 さいしょ 平衡 へいこう 暗黙 あんもく 溶媒 ようばい アンブレラ・サンプリング法 ほう を用 もち 行 おこな [76] 計算 けいさん 高 たか 明示 めいじ 的 てき 水 みず 用 もち 第 だい 一 いち 原理 げんり 計算 けいさん 非常 ひじょう 小 ちい タンパク質 たんぱくしつ 限定 げんてい [77] [78] 大 おお タンパク質 たんぱくしつ 実験 じっけん 的 てき 構造 こうぞう 動力 どうりょく 学 がく 高温 こうおん 限定 げんてい 小 ちい タンパク質 たんぱくしつ 約 やく 残 ざん 基 もと 以上 いじょう 長時間 ちょうじかん 約 やく 秒 びょう 超 こ 粗 あら 視 し 化 か (英語 えいご 版 ばん 用 もち 解析 かいせき [79] [80] [81]
スタンフォード大学 だいがく 教授 きょうじゅ ビジェイ・S・パンデ のグループが作成 さくせい 級 きゅう 分散 ぶんさん Folding@home は、ボランティアのパーソナルコンピュータのCPU とGPU のアイドル処理 しょり 時間 じかん 利用 りよう タンパク質 たんぱくしつ タンパク質 たんぱくしつ 誤 あやま 折 お 理解 りかい 疾患 しっかん 研究 けんきゅう 創 そう 薬 やく ドラッグデザイン を加速 かそく 目的 もくてき
D. E. Shaw Research社 しゃ (英語 えいご 版 ばん のカスタムASIC と相互 そうご 接続 せつぞく 中心 ちゅうしん 設計 せっけい 構築 こうちく 超 ちょう 並列 へいれつ Anton (アントン)で、長時間 ちょうじかん 連続 れんぞく 軌道 きどう 実行 じっこう 使用 しよう 実行 じっこう 公開 こうかい 最長 さいちょう 結果 けっか 秒 びょう [82]
注釈 ちゅうしゃく 出典 しゅってん [ 編集 へんしゅう ]
^ “So much more to know” . Science 309 (5731): 78–102. (July 2005). doi :10.1126/science.309.5731.78b . PMID 15994524 . http://www.sciencemag.org/cgi/content/full/309/5731/78b . ^ King (2007年 ねん MIT OpenCourseWare - 7.88J / 5.48J / 7.24J / 10.543J Protein Folding Problem, Fall 2007 Lecture Notes - 1 ”. MIT OpenCourseWare 年 ねん 月 がつ 日 にち 時点 じてん オリジナル よりアーカイブ。2013年 ねん 月 がつ 日 にち 閲覧 えつらん ^ Dill KA (June 2008). “The Protein Folding Problem” . Annu Rev Biophys 37 : 289–316. doi :10.1146/annurev.biophys.37.092707.153558 . PMC 2443096 . PMID 18573083 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2443096/ . ^ Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walters, Peter (2002). “The Shape and Structure of Proteins” . Molecular Biology of the Cell; Fourth Edition . New York and London: Garland Science. ISBN 978-0-8153-3218-3 . https://www.ncbi.nlm.nih.gov/books/NBK26830/ ^ Anfinsen CB (July 1972). “The formation and stabilization of protein structure” . The Biochemical Journal 128 (4): 737–49. doi :10.1042/bj1280737 . PMC 1173893 . PMID 4565129 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1173893/ . ^ Berg, Jeremy M.; Tymoczko, John L.; Stryer, Lubert (2002). “3. Protein Structure and Function” . Biochemistry . San Francisco: W. H. Freeman. ISBN 978-0-7167-4684-3 . https://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Search&db=books&doptcmdl=GenBookHL&term=stryer%5Bbook%5D+AND+215168%5Buid%5D&rid=stryer.chapter.280 ^ a b Selkoe DJ (December 2003). “Folding proteins in fatal ways”. Nature 426 (6968): 900–4. Bibcode : 2003Natur.426..900S . doi :10.1038/nature02264 . PMID 14685251 .
^ Alberts, Bruce; Bray, Dennis; Hopkin, Karen; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter (2010). “Protein Structure and Function”. Essential cell biology (Third ed.). New York, NY: Garland Science. pp. 120–70. ISBN 978-0-8153-4454-4 ^ Kim PS, Baldwin RL (1990). “Intermediates in the folding reactions of small proteins”. Annual Review of Biochemistry 59 : 631–60. doi :10.1146/annurev.bi.59.070190.003215 . PMID 2197986 . ^ Jackson SE (1998). “How do small single-domain proteins fold?”. Folding & Design 3 (4): R81-91. doi :10.1016/S1359-0278(98)00033-9 . PMID 9710577 . ^ Kubelka J, Hofrichter J, Eaton WA (February 2004). “The protein folding 'speed limit'” . Current Opinion in Structural Biology 14 (1): 76–88. doi :10.1016/j.sbi.2004.01.013 . PMID 15102453 . https://zenodo.org/record/1259347 . ^ Anfinsen CB (July 1973). “Principles that govern the folding of protein chains”. Science 181 (4096): 223–30. Bibcode : 1973Sci...181..223A . doi :10.1126/science.181.4096.223 . PMID 4124164 . ^ a b c d e f g h Voet, Donald; Voet, Judith G.; Pratt, Charlotte W. (2016). Principles of Biochemistry (Fifth ed.). Wiley. ISBN 978-1-118-91840-1
^ Alexander PA, He Y, Chen Y, Orban J, Bryan PN (July 2007). “The design and characterization of two proteins with 88% sequence identity but different structure and function” . Proceedings of the National Academy of Sciences of the United States of America 104 (29): 11963–8. Bibcode : 2007PNAS..10411963A . doi :10.1073/pnas.0700922104 . PMC 1906725 . PMID 17609385 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1906725/ . ^ Rose GD, Fleming PJ, Banavar JR, Maritan A (November 2006). “A backbone-based theory of protein folding” . Proceedings of the National Academy of Sciences of the United States of America 103 (45): 16623–33. Bibcode : 2006PNAS..10316623R . doi :10.1073/pnas.0606843103 . PMC 1636505 . PMID 17075053 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1636505/ . ^ a b c Fersht, Alan (1999). Structure and Mechanism in Protein Science: A Guide to Enzyme Catalysis and Protein Folding ISBN 978-0-7167-3268-6 . https://books.google.com/books?id=QdpZz_ahA5UC&pg=PR20
^ “Protein Structure ”. Scitable . Nature Education. 2016年 ねん 日 にち 閲覧 えつらん ^ Pratt, Charlotte; Cornely, Kathleen (2004). “Thermodynamics” . Essential Biochemistry ISBN 978-0-471-39387-0 . http://www.wiley.com/college/pratt/0471393878/instructor/review/thermodynamics/7_relationship.html 2016年 ねん 日 にち 閲覧 えつらん ^ Fedorov AN (1997). “Cotranslational Protein Folding”. J. Biol. Chem. 272 (52): 32715-32718. doi :10.1074/jbc.272.52.32715 . ^ Zhang, Gong; Ignatova, Zoya (2011-02-01). “Folding at the birth of the nascent chain: coordinating translation with co-translational folding”. Current Opinion in Structural Biology 21 (1): 25–31. doi :10.1016/j.sbi.2010.10.008 . ISSN 0959-440X . PMID 21111607 . ^ van den Berg B, Wain R, Dobson CM, Ellis RJ (August 2000). “Macromolecular crowding perturbs protein refolding kinetics: implications for folding inside the cell” . The EMBO Journal 19 (15): 3870–5. doi :10.1093/emboj/19.15.3870 . PMC 306593 . PMID 10921869 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC306593/ . ^ Al-Karadaghi, Salam. “Torsion Angles and the Ramachnadran Plot in Protein Structures ”. www.proteinstructures.com . 2016年 ねん 日 にち 閲覧 えつらん ^ Pace CN, Shirley BA, McNutt M, Gajiwala K (January 1996). “Forces contributing to the conformational stability of proteins”. FASEB Journal 10 (1): 75–83. doi :10.1096/fasebj.10.1.8566551 . PMID 8566551 . ^ Cui D, Ou S, Patel S (December 2014). “Protein-spanning water networks and implications for prediction of protein-protein interactions mediated through hydrophobic effects”. Proteins 82 (12): 3312–26. doi :10.1002/prot.24683 . PMID 25204743 . ^ Tanford C (June 1978). “The hydrophobic effect and the organization of living matter”. Science 200 (4345): 1012–8. Bibcode : 1978Sci...200.1012T . doi :10.1126/science.653353 . PMID 653353 . ^ Deechongkit S, Nguyen H, Powers ET, Dawson PE, Gruebele M, Kelly JW (July 2004). “Context-dependent contributions of backbone hydrogen bonding to beta-sheet folding energetics”. Nature 430 (6995): 101–5. Bibcode : 2004Natur.430..101D . doi :10.1038/nature02611 . PMID 15229605 . ^ Irbäck, Anders; Sandelin, Erik (November 2000). “On Hydrophobicity Correlations in Protein Chains” . Biophysical Journal 79 (5): 2252–2258. arXiv :cond-mat/0010390 . Bibcode : 2000BpJ....79.2252I . doi :10.1016/S0006-3495(00)76472-1 . PMC 1301114 . PMID 11053106 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1301114/ . ^ Irbäck, A.; Peterson, C.; Potthast, F. (3 September 1996). “Evidence for nonrandom hydrophobicity structures in protein chains.” . Proceedings of the National Academy of Sciences 93 (18): 9533–9538. arXiv :chem-ph/9512004 . Bibcode : 1996PNAS...93.9533I . doi :10.1073/pnas.93.18.9533 . PMC 38463 . PMID 8790365 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC38463/ . ^ Wilson, Benjamin A.; Foy, Scott G.; Neme, Rafik; Masel, Joanna (24 April 2017). “Young genes are highly disordered as predicted by the preadaptation hypothesis of de novo gene birth” . Nature Ecology & Evolution 1 (6): 0146–146. doi :10.1038/s41559-017-0146 . PMC 5476217 . PMID 28642936 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5476217/ . ^ Willis, Sara; Masel, Joanna (September 2018). “Gene Birth Contributes to Structural Disorder Encoded by Overlapping Genes” . Genetics 210 (1): 303–313. doi :10.1534/genetics.118.301249 . PMC 6116962 . PMID 30026186 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6116962/ . ^ Foy, Scott G.; Wilson, Benjamin A.; Bertram, Jason; Cordes, Matthew H. J.; Masel, Joanna (April 2019). “A Shift in Aggregation Avoidance Strategy Marks a Long-Term Direction to Protein Evolution” . Genetics 211 (4): 1345–1355. doi :10.1534/genetics.118.301719 . PMC 6456324 . PMID 30692195 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6456324/ . ^ a b c d e f Dobson CM (December 2003). “Protein folding and misfolding”. Nature 426 (6968): 884–90. Bibcode : 2003Natur.426..884D . doi :10.1038/nature02261 . PMID 14685248 .
^ a b c Hartl FU (June 1996). “Molecular chaperones in cellular protein folding”. Nature 381 (6583): 571–9. Bibcode : 1996Natur.381..571H . doi :10.1038/381571a0 . PMID 8637592 .
^ a b Hartl FU, Bracher A, Hayer-Hartl M (July 2011). “Molecular chaperones in protein folding and proteostasis”. Nature 475 (7356): 324–32. doi :10.1038/nature10317 . PMID 21776078 .
^ Kim YE, Hipp MS, Bracher A, Hayer-Hartl M, Hartl FU (2013). “Molecular chaperone functions in protein folding and proteostasis”. Annual Review of Biochemistry 82 : 323–55. doi :10.1146/annurev-biochem-060208-092442 . PMID 23746257 . ^ Shortle D (January 1996). “The denatured state (the other half of the folding equation) and its role in protein stability”. FASEB Journal 10 (1): 27–34. doi :10.1096/fasebj.10.1.8566543 . PMID 8566543 . ^ Lee S, Tsai FT (2005). “Molecular chaperones in protein quality control”. Journal of Biochemistry and Molecular Biology 38 (3): 259–65. doi :10.5483/BMBRep.2005.38.3.259 . PMID 15943899 . ^ Ojeda-May P, Garcia ME (July 2010). “Electric field-driven disruption of a native beta-sheet protein conformation and generation of a helix-structure” . Biophysical Journal 99 (2): 595–9. Bibcode : 2010BpJ....99..595O . doi :10.1016/j.bpj.2010.04.040 . PMC 2905109 . PMID 20643079 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2905109/ . ^ van den Berg B, Ellis RJ, Dobson CM (December 1999). “Effects of macromolecular crowding on protein folding and aggregation” . The EMBO Journal 18 (24): 6927–33. doi :10.1093/emboj/18.24.6927 . PMC 1171756 . PMID 10601015 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1171756/ . ^ Ellis RJ (July 2006). “Molecular chaperones: assisting assembly in addition to folding”. Trends in Biochemical Sciences 31 (7): 395–401. doi :10.1016/j.tibs.2006.05.001 . PMID 16716593 . ^ Takai K, Nakamura K, Toki T, Tsunogai U, Miyazaki M, Miyazaki J, Hirayama H, Nakagawa S, Nunoura T, Horikoshi K (August 2008). “Cell proliferation at 122 degrees C and isotopically heavy CH4 production by a hyperthermophilic methanogen under high-pressure cultivation” . Proceedings of the National Academy of Sciences of the United States of America 105 (31): 10949–54. Bibcode : 2008PNAS..10510949T . doi :10.1073/pnas.0712334105 . PMC 2490668 . PMID 18664583 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2490668/ . ^ a b Marusich EI, Kurochkina LP, Mesyanzhinov VV. Chaperones in bacteriophage T4 assembly. Biochemistry (Mosc). 1998;63(4):399-406
^ a b c d Chaudhuri TK, Paul S (April 2006). “Protein-misfolding diseases and chaperone-based therapeutic approaches”. The FEBS Journal 273 (7): 1331–49. doi :10.1111/j.1742-4658.2006.05181.x . PMID 16689923 .
^ a b Soto C, Estrada L, Castilla J (March 2006). “Amyloids, prions and the inherent infectious nature of misfolded protein aggregates”. Trends in Biochemical Sciences 31 (3): 150–5. doi :10.1016/j.tibs.2006.01.002 . PMID 16473510 .
^ Hammarström P, Wiseman RL, Powers ET, Kelly JW (January 2003). “Prevention of transthyretin amyloid disease by changing protein misfolding energetics”. Science 299 (5607): 713–6. Bibcode : 2003Sci...299..713H . doi :10.1126/science.1079589 . PMID 12560553 . ^ Chiti F, Dobson CM (2006). “Protein misfolding, functional amyloid, and human disease”. Annual Review of Biochemistry 75 : 333–66. doi :10.1146/annurev.biochem.75.101304.123901 . PMID 16756495 . ^ Johnson SM, Wiseman RL, Sekijima Y, Green NS, Adamski-Werner SL, Kelly JW (December 2005). “Native state kinetic stabilization as a strategy to ameliorate protein misfolding diseases: a focus on the transthyretin amyloidoses”. Accounts of Chemical Research 38 (12): 911–21. doi :10.1021/ar020073i . PMID 16359163 . ^ a b Cowtan K (2001). "Phase Problem in X-ray Crystallography, and Its Solution" (PDF) . Encyclopedia of Life Sciences . Macmillan Publishers Ltd, Nature Publishing Group. 2016年 ねん 日 にち 閲覧 えつらん 。
^ Drenth, Jan (2007-04-05). Principles of Protein X-Ray Crystallography ISBN 978-0-387-33746-3 . https://books.google.com/books?id=Jobr7svN0IIC&pg=PR5 ^ Taylor, Garry (2003). “The phase problem”. Acta Crystallographica Section D 59 (11): 1881–90. doi :10.1107/S0907444903017815 . PMID 14573942 . ^ a b c Bedouelle H (February 2016). “Principles and equations for measuring and interpreting protein stability: From monomer to tetramer”. Biochimie 121 : 29–37. doi :10.1016/j.biochi.2015.11.013 . PMID 26607240 .
^ Monsellier E, Bedouelle H (September 2005). “Quantitative measurement of protein stability from unfolding equilibria monitored with the fluorescence maximum wavelength”. Protein Engineering, Design & Selection 18 (9): 445–56. doi :10.1093/protein/gzi046 . PMID 16087653 . ^ Park YC, Bedouelle H (July 1998). “Dimeric tyrosyl-tRNA synthetase from Bacillus stearothermophilus unfolds through a monomeric intermediate. A quantitative analysis under equilibrium conditions”. The Journal of Biological Chemistry 273 (29): 18052–9. doi :10.1074/jbc.273.29.18052 . PMID 9660761 . ^ Ould-Abeih MB, Petit-Topin I, Zidane N, Baron B, Bedouelle H (June 2012). “Multiple folding states and disorder of ribosomal protein SA, a membrane receptor for laminin, anticarcinogens, and pathogens”. Biochemistry 51 (24): 4807–21. doi :10.1021/bi300335r . PMID 22640394 . ^ Royer CA (May 2006). “Probing protein folding and conformational transitions with fluorescence”. Chemical Reviews 106 (5): 1769–84. doi :10.1021/cr0404390 . PMID 16683754 . ^ Beatrice M.P. Huyghues-Despointes, C. Nick Pace, S. Walter Englander, and J. Martin Scholtz. "Measuring the Conformational Stability of a Protein by Hydrogen Exchange." Methods in Molecular Biology. Kenneth P. Murphy Ed. Humana Press, Totowa, New Jersey, 2001. pp. 69–92
^ Cross, Graham H.; Freeman, Neville J.; Swann, Marcus J. (2008). “Dual Polarization Interferometry: A Real-Time Optical Technique for Measuring (Bio)molecular Orientation, Structure and Function at the Solid/Liquid Interface”. Handbook of Biosensors and Biochips . doi :10.1002/9780470061565.hbb055 . ISBN 978-0-470-01905-4 ^ Bu Z, Cook J, Callaway DJ (September 2001). “Dynamic regimes and correlated structural dynamics in native and denatured alpha-lactalbumin”. Journal of Molecular Biology 312 (4): 865–73. doi :10.1006/jmbi.2001.5006 . PMID 11575938 . ^ Minde DP, Maurice MM, Rüdiger SG (2012). “Determining biophysical protein stability in lysates by a fast proteolysis assay, FASTpp” . PLOS ONE 7 (10): e46147. Bibcode : 2012PLoSO...746147M . doi :10.1371/journal.pone.0046147 . PMC 3463568 . PMID 23056252 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3463568/ . ^ Park C, Marqusee S (March 2005). “Pulse proteolysis: a simple method for quantitative determination of protein stability and ligand binding”. Nature Methods 2 (3): 207–12. doi :10.1038/nmeth740 . PMID 15782190 . ^ Mashaghi A, Kramer G, Lamb DC, Mayer MP, Tans SJ (January 2014). “Chaperone action at the single-molecule level”. Chemical Reviews 114 (1): 660–76. doi :10.1021/cr400326k . PMID 24001118 . ^ Jagannathan B, Marqusee S (November 2013). “Protein folding and unfolding under force” . Biopolymers 99 (11): 860–9. doi :10.1002/bip.22321 . PMC 4065244 . PMID 23784721 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4065244/ . ^ Jakobi AJ, Mashaghi A, Tans SJ, Huizinga EG (July 2011). “Calcium modulates force sensing by the von Willebrand factor A2 domain” . Nature Communications 2 : 385. Bibcode : 2011NatCo...2..385J . doi :10.1038/ncomms1385 . PMC 3144584 . PMID 21750539 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3144584/ . ^ Jagannathan B, Elms PJ, Bustamante C, Marqusee S (October 2012). “Direct observation of a force-induced switch in the anisotropic mechanical unfolding pathway of a protein” . Proceedings of the National Academy of Sciences of the United States of America 109 (44): 17820–5. Bibcode : 2012PNAS..10917820J . doi :10.1073/pnas.1201800109 . PMC 3497811 . PMID 22949695 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3497811/ . ^ Minde DP, Ramakrishna M, Lilley KS (2018). “Biotinylation by proximity labelling favours unfolded proteins”. bioRxiv . doi :10.1101/274761 . ^ Compiani M, Capriotti E (December 2013). “Computational and theoretical methods for protein folding”. Biochemistry 52 (48): 8601–24. doi :10.1021/bi4001529 . PMID 24187909 . ^ “Structural Biochemistry/Proteins/Protein Folding - Wikibooks, open books for an open world ”. en.wikibooks.org . 2016年 ねん 日 にち 閲覧 えつらん ^ Levinthal, Cyrus (1968). “Are there pathways for protein folding?” . Journal de Chimie Physique et de Physico-Chimie Biologique 65 : 44–45. Bibcode : 1968JCP....65...44L . doi :10.1051/jcp/1968650044 . オリジナル の2009-09-02時点 じてん . https://web.archive.org/web/20090902211239/http://www.biochem.wisc.edu/courses/biochem704/Reading/Levinthal1968.pdf . ^ a b c Bryngelson JD, Onuchic JN, Socci ND, Wolynes PG (March 1995). “Funnels, pathways, and the energy landscape of protein folding: a synthesis”. Proteins 21 (3): 167–95. arXiv :chem-ph/9411008 . doi :10.1002/prot.340210302 . PMID 7784423 .
^ Leopold PE; Montal M; Onuchic JN (September 1992). “Protein folding funnels: a kinetic approach to the sequence-structure relationship” . Proceedings of the National Academy of Sciences of the United States of America 89 (18): 8721–5. Bibcode : 1992PNAS...89.8721L . doi :10.1073/pnas.89.18.8721 . PMC 49992 . PMID 1528885 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC49992/ . ^ Sharma, Vivek; Kaila, Ville R.I.; Annila, Arto (2009). “Protein folding as an evolutionary process”. Physica A: Statistical Mechanics and Its Applications 388 (6): 851–62. Bibcode : 2009PhyA..388..851S . doi :10.1016/j.physa.2008.12.004 . ^ Robson, Barry; Vaithilingam, Andy (2008). “Protein Folding Revisited”. Molecular Biology of Protein Folding, Part B . Progress in Molecular Biology and Translational Science. 84 . pp. 161–202. doi :10.1016/S0079-6603(08)00405-4 . ISBN 978-0-12-374595-8 . PMID 19121702 ^ a b c Dill KA, MacCallum JL (November 2012). “The protein-folding problem, 50 years on”. Science 338 (6110): 1042–6. Bibcode : 2012Sci...338.1042D . doi :10.1126/science.1219021 . PMID 23180855 .
^ a b Fersht AR (February 2000). “Transition-state structure as a unifying basis in protein-folding mechanisms: contact order, chain topology, stability, and the extended nucleus mechanism” . Proceedings of the National Academy of Sciences of the United States of America 97 (4): 1525–9. Bibcode : 2000PNAS...97.1525F . doi :10.1073/pnas.97.4.1525 . PMC 26468 . PMID 10677494 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC26468/ .
^ Rizzuti B, Daggett V (March 2013). “Using simulations to provide the framework for experimental protein folding studies” . Archives of Biochemistry and Biophysics 531 (1–2): 128–35. doi :10.1016/j.abb.2012.12.015 . PMC 4084838 . PMID 23266569 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4084838/ . ^ Schaefer M, Bartels C, Karplus M (December 1998). “Solution conformations and thermodynamics of structured peptides: molecular dynamics simulation with an implicit solvation model”. Journal of Molecular Biology 284 (3): 835–48. doi :10.1006/jmbi.1998.2172 . PMID 9826519 . ^ Jones, David. “Fragment-based Protein Folding Simulations ”. University College London. 2020年 ねん 月 がつ 日 にち 閲覧 えつらん ^ “Protein folding ” (by Molecular Dynamics). 2020年 ねん 月 がつ 日 にち 閲覧 えつらん ^ Kmiecik S, Gront D, Kolinski M, Wieteska L, Dawid AE, Kolinski A (July 2016). “Coarse-Grained Protein Models and Their Applications”. Chemical Reviews 116 (14): 7898–936. doi :10.1021/acs.chemrev.6b00163 . PMID 27333362 . ^ Kmiecik S, Kolinski A (July 2007). “Characterization of protein-folding pathways by reduced-space modeling” . Proceedings of the National Academy of Sciences of the United States of America 104 (30): 12330–5. Bibcode : 2007PNAS..10412330K . doi :10.1073/pnas.0702265104 . PMC 1941469 . PMID 17636132 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1941469/ . ^ Adhikari AN, Freed KF, Sosnick TR (October 2012). “De novo prediction of protein folding pathways and structure using the principle of sequential stabilization” . Proceedings of the National Academy of Sciences of the United States of America 109 (43): 17442–7. Bibcode : 2012PNAS..10917442A . doi :10.1073/pnas.1209000109 . PMC 3491489 . PMID 23045636 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3491489/ . ^ Lindorff-Larsen K, Piana S, Dror RO, Shaw DE (October 2011). “How fast-folding proteins fold”. Science 334 (6055): 517–20. Bibcode : 2011Sci...334..517L . doi :10.1126/science.1208351 . PMID 22034434 .
藤 ふじ 博幸 ひろゆき 編 へん タンパク質 たんぱくしつ 立体 りったい 構造 こうぞう 入門 にゅうもん 講談社 こうだんしゃ Voet & Voet "Biochemistry" 3rd edition, Wiley (2004) 全般 ぜんぱん α あるふぁ β べーた α あるふぁ β べーた α あるふぁ β べーた イレギュラードメイン