デオキシリボ核酸
デオキシリボ

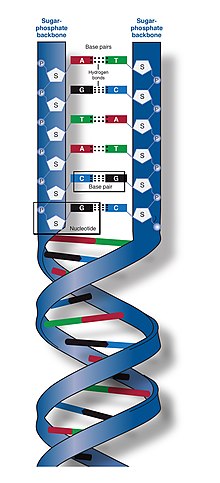
DNAの
特性
編集
DNAはヌクレオチドと
DNA
このようにDNA
DNA
核酸 塩基 の分類
編集
非 標準 塩基
編集
DNAには
DNAには
修飾 アデニン- N6-カルバモイル-メチルアデニン
- N6-メチルアデニン
修飾 グアニン- 7-デアザグアニン
- 7-メチルグアニン
修飾 シトシン- N4-メチルシトシン
- 5-カルボキシルシトシン
- 5-ホルミルシトシン
- 5-グリコシルヒドロキシメチルシトシン
- 5-ヒドロキシシトシン
- 5-メチルシトシン
修飾 チミジンα -グルタミルチミジンα -プトレシニルチミン
- ウラシルおよび
修飾 物 塩基 J- ウラシル
- 5-ジヒドロキシペンタウラシル
- 5-ヒドロキシメチルデオキシウラシル
- その
他 - デオキシアルケオシン
- 2,6-ジアミノプリン(2-アミノアデニン)
主 溝 と副 溝
編集
塩基 対 合
編集
DNAの
ssDNAとdsDNA
編集
dsDNA
含有 量
編集
ヒトの
センスとアンチセンス
編集
あるDNA
スーパーコイル
編集
DNAは、DNAスーパーコイル(
代替 DNA構造
編集
DNAは、A-DNA(A
A-DNA、およびB-DNAのX
B-DNAは
B-DNAと
代替 DNA化学
編集
四 重 鎖 構造
編集
これらのグアニンに
このような
分岐 DNA
編集
DNAでは、
人工 塩基
編集
いくつかの
酸性 度
編集
DNAのリン
巨視的 外観
編集
化学 修飾 とDNAパッケージングの変化
編集
塩基 修飾 とDNAパッケージング
編集
たとえば、シトシンのメチル
損傷
編集
DNAは、DNA
生物 学 的 機能
編集
DNAは
遺伝子 とゲノム
編集
ゲノムDNAは、DNA
転写 と翻訳
編集
複製
編集
細胞 外 核酸
編集
好 中 球 細胞 外 トラップ
編集
タンパク質 との相互 作用
編集
DNAの
DNA結合 タンパク質
編集
DNAと
DNA
これらのDNA
DNA修飾 酵素
編集
ヌクレアーゼとリガーゼ
編集
ヌクレアーゼは、ホスホジエステル
DNAリガーゼと
トポイソメラーゼとヘリカーゼ
編集
トポイソメラーゼはヌクレアーゼとリガーゼの
ヘリカーゼは
ポリメラーゼ
編集
ポリメラーゼはヌクレオシド
DNA
RNA
遺伝子 組 換 え
編集
DNAらせんは
進化
編集
DNAにはあらゆる
DNAの
2021
技術 における用途
編集
遺伝子 工学
編集
フェノール・クロロホルム
DNAプロファイリング
編集
DNAプロファイリングはまた、
DNAプロファイリングは、
DNA酵素 または触媒 DNA
編集
デオキシリボザイム(
バイオインフォマティクス
編集
バイオインフォマティクスは、DNA
DNAナノテクノロジー
編集
DNAナノテクノロジーは、DNAや
系統 学 と人類 学
編集
DNAは
情報 ストレージ
編集
歴史
編集
DNAが
1909
1933
1937
1943
1951
1952
1953
1962
1957
1986
符号 位置
編集
| Unicode | JIS X 0213 | |||
|---|---|---|---|---|
| 🧬 | U+1F9EC |
- |
🧬🧬 |
dna |
参照 項目
編集
常 染色 体 -性 染色 体 以外 の染色 体 結晶 学 -結晶 性 固体 中 の原子 の配列 を決定 する科学 的 研究 全米 DNAの日 - 4月 25日 に祝 われるアメリカ合衆国 の祝日 - DNAマイクロアレイ -
固体 表面 に付着 した微細 なDNAスポットの集合 体 - DNAシークエンシング -
核酸 配列 を決定 する工程 遺伝子 疾患 - ゲノムの1つまたは複数 の異常 によって引 き起 こされる健康 問題 遺伝子 系図 - DNA検査 によって推定 した個人 間 の遺伝 的 関係 - ハプロタイプ -
親 から受 け継 いだ遺伝子 の集合 減数 分裂 -単数 体 の配偶 子 を作 る細胞 分裂 の様式 核酸 表記 法 - DNAの4つのヌクレオチドをA、C、G、Tと呼 ぶ国際 共通 表記 法 塩基 配列 -核酸 中 のヌクレオチドの連続 物 - リボソームDNA - リボソームRNAをコーディングするDNAの
特定 の領域 - サザンブロット - DNA
配列 の検出 と定量 に用 いられる分析 技術 - X
線 回折 法 - X線 ビームの散乱 強度 に基 づく非破壊 分析 技術 の一群 - ゼノ
核酸 -合成 核酸 アナログ 三 本 鎖 DNA - 3本 のオリゴヌクレオチドが三重 らせんを形成 するDNA構造
脚注
編集
- ^ "deoxyribonucleic acid". Merriam-Webster Dictionary. 2023
年 12月13日 閲覧 。 - ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2014
年 ). Molecular Biology of the Cell (6th ed.). Garland. p. Chapter 4: DNA, Chromosomes and Genomes. ISBN 978-0-8153-4432-2. 2014年 7月 14日 時点 のオリジナルよりアーカイブ。 - ^ Purcell A. “DNA”. Basic Biology. 2017
年 1月 5日 時点 のオリジナルよりアーカイブ。2015年 7月 28日 閲覧 。 - ^ “Uracil” (
英語 ). Genome.gov. 2019年 11月21日 閲覧 。 - ^ Russell P (2001
年 ). iGenetics. New York: Benjamin Cummings. ISBN 0-8053-4553-1。 - ^ Saenger W (1984). Principles of Nucleic Acid Structure. New York: Springer-Verlag. ISBN 0-387-90762-9
- ^ a b Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Peter W (2002). Molecular Biology of the Cell (Fourth ed.). New York and London: Garland Science. ISBN 0-8153-3218-1. OCLC 145080076. オリジナルの1 November 2016
時点 におけるアーカイブ。 - ^ Irobalieva RN, Fogg JM, Catanese DJ, Catanese DJ, Sutthibutpong T, Chen M, Barker AK, Ludtke SJ, Harris SA, Schmid MF, Chiu W, Zechiedrich L (October 2015). “Structural diversity of supercoiled DNA”. Nature Communications 6: 8440. Bibcode: 2015NatCo...6.8440I. doi:10.1038/ncomms9440. ISSN 2041-1723. PMC 4608029. PMID 26455586.
- ^ a b c d Watson JD, Crick FH (April 1953). “Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid”. Nature 171 (4356): 737–38. Bibcode: 1953Natur.171..737W. doi:10.1038/171737a0. ISSN 0028-0836. PMID 13054692. オリジナルの4 February 2007
時点 におけるアーカイブ。. - ^ Mandelkern M, Elias JG, Eden D, Crothers DM (October 1981). “The dimensions of DNA in solution”. Journal of Molecular Biology 152 (1): 153–61. doi:10.1016/0022-2836(81)90099-1. ISSN 0022-2836. PMID 7338906.
- ^ Arrighi, Frances E.; Mandel, Manley; Bergendahl, Janet; Hsu, T. C. (June 1970). “Buoyant densities of DNA of mammals”. Biochemical Genetics 4 (3): 367–376. doi:10.1007/BF00485753.
- ^ a b c d Berg J, Tymoczko J, Stryer L (2002). Biochemistry. W.H. Freeman and Company. ISBN 0-7167-4955-6
- ^ IUPAC-IUB Commission on Biochemical Nomenclature (CBN) (December 1970). “Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents. Recommendations 1970”. The Biochemical Journal 120 (3): 449–54. doi:10.1042/bj1200449. ISSN 0306-3283. PMC 1179624. PMID 5499957. オリジナルの5 February 2007
時点 におけるアーカイブ。. - ^ a b Ghosh A, Bansal M (April 2003). “A glossary of DNA structures from A to Z”. Acta Crystallographica Section D 59 (Pt 4): 620–26. doi:10.1107/S0907444903003251. ISSN 0907-4449. PMID 12657780.
- ^ Edwards KJ, Brown DG, Spink N, Skelly JV, Neidle S. “RCSB PDB – 1D65: Molecular structure of the B-DNA dodecamer d(CGCAAATTTGCG)2. An examination of propeller twist and minor-groove water structure at 2.2 A resolution.” (
英語 ). www.rcsb.org. 2023年 3月 27日 閲覧 。 - ^ Yakovchuk P, Protozanova E, Frank-Kamenetskii MD (2006). “Base-stacking and base-pairing contributions into thermal stability of the DNA double helix”. Nucleic Acids Research 34 (2): 564–74. doi:10.1093/nar/gkj454. ISSN 0305-1048. PMC 1360284. PMID 16449200.
- ^ Tropp BE (2012). Molecular Biology (4th ed.). Sudbury, Mass.: Jones and Barlett Learning. ISBN 978-0-7637-8663-2
- ^ Carr S (1953
年 ). “Watson-Crick Structure of DNA”. Memorial University of Newfoundland. 2016年 7月 19日 時点 のオリジナルよりアーカイブ。2016年 7月 13日 閲覧 。 - ^ Verma S, Eckstein F (1998). “Modified oligonucleotides: synthesis and strategy for users”. Annual Review of Biochemistry 67: 99–134. doi:10.1146/annurev.biochem.67.1.99. ISSN 0066-4154. PMID 9759484.
- ^ Johnson TB, Coghill RD (1925). “Pyrimidines. CIII. The discovery of 5-methylcytosine in tuberculinic acid, the nucleic acid of the tubercle bacillus.”. Journal of the American Chemical Society 47: 2838–44. doi:10.1021/ja01688a030. ISSN 0002-7863.
- ^ Weigele P, Raleigh EA (October 2016). “Biosynthesis and Function of Modified Bases in Bacteria and Their Viruses”. Chemical Reviews 116 (20): 12655–12687. doi:10.1021/acs.chemrev.6b00114. ISSN 0009-2665. PMID 27319741.
- ^ Kumar S, Chinnusamy V, Mohapatra T (2018). “Epigenetics of Modified DNA Bases: 5-Methylcytosine and Beyond”. Frontiers in Genetics 9: 640. doi:10.3389/fgene.2018.00640. ISSN 1664-8021. PMC 6305559. PMID 30619465.
- ^ Carell T, Kurz MQ, Müller M, Rossa M, Spada F (April 2018). “Non-canonical Bases in the Genome: The Regulatory Information Layer in DNA”. Angewandte Chemie 57 (16): 4296–4312. doi:10.1002/anie.201708228. PMID 28941008.
- ^ Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson RE (October 1980). “Crystal structure analysis of a complete turn of B-DNA”. Nature 287 (5784): 755–58. Bibcode: 1980Natur.287..755W. doi:10.1038/287755a0. PMID 7432492.
- ^ a b Pabo CO, Sauer RT (1984). “Protein-DNA recognition”. Annual Review of Biochemistry 53: 293–321. doi:10.1146/annurev.bi.53.070184.001453. PMID 6236744.
- ^ Nikolova EN, Zhou H, Gottardo FL, Alvey HS, Kimsey IJ, Al-Hashimi HM (2013). “A historical account of Hoogsteen base-pairs in duplex DNA”. Biopolymers 99 (12): 955–68. doi:10.1002/bip.22334. PMC 3844552. PMID 23818176.
- ^ Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub HE (April 2000). “Mechanical stability of single DNA molecules”. Biophysical Journal 78 (4): 1997–2007. Bibcode: 2000BpJ....78.1997C. doi:10.1016/S0006-3495(00)76747-6. PMC 1300792. PMID 10733978.
- ^ Chalikian TV, Völker J, Plum GE, Breslauer KJ (July 1999). “A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques”. Proceedings of the National Academy of Sciences of the United States of America 96 (14): 7853–58. Bibcode: 1999PNAS...96.7853C. doi:10.1073/pnas.96.14.7853. PMC 22151. PMID 10393911.
- ^ deHaseth PL, Helmann JD (June 1995). “Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA”. Molecular Microbiology 16 (5): 817–24. doi:10.1111/j.1365-2958.1995.tb02309.x. PMID 7476180.
- ^ Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J (December 2004). “Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern”. Biochemistry 43 (51): 15996–6010. doi:10.1021/bi048221v. PMID 15609994. オリジナルの10 June 2007
時点 におけるアーカイブ。. - ^ a b Piovesan A, Pelleri MC, Antonaros F, Strippoli P, Caracausi M, Vitale L (2019). “On the length, weight and GC content of the human genome.”. BMC Res Notes 12 (1): 106. doi:10.1186/s13104-019-4137-z. PMC 6391780. PMID 30813969.
- ^ Gregory SG, Barlow KF, McLay KE, Kaul R, Swarbreck D, Dunham A, Scott CE, Howe KL, Woodfine K, Spencer CC, Jones MC, Gillson C, Searle S, Zhou Y, Kokocinski F, McDonald L, Evans R, Phillips K, Atkinson A, Cooper R, Jones C, Hall RE, Andrews TD, Lloyd C, Ainscough R, Almeida JP, Ambrose KD, Anderson F, Andrew RW, Ashwell RI, Aubin K, Babbage AK, Bagguley CL, Bailey J, Beasley H, Bethel G, Bird CP, Bray-Allen S, Brown JY, Brown AJ, Buckley D, Burton J, Bye J, Carder C, Chapman JC, Clark SY, Clarke G, Clee C, Cobley V, Collier RE, Corby N, Coville GJ, Davies J, Deadman R, Dunn M, Earthrowl M, Ellington AG, Errington H, Frankish A, Frankland J, French L, Garner P, Garnett J, Gay L, Ghori MR, Gibson R, Gilby LM, Gillett W, Glithero RJ, Grafham DV, Griffiths C, Griffiths-Jones S, Grocock R, Hammond S, Harrison ES, Hart E, Haugen E, Heath PD, Holmes S, Holt K, Howden PJ, Hunt AR, Hunt SE, Hunter G, Isherwood J, James R, Johnson C, Johnson D, Joy A, Kay M, Kershaw JK, Kibukawa M, Kimberley AM, King A, Knights AJ, Lad H, Laird G, Lawlor S, Leongamornlert DA, Lloyd DM, Loveland J, Lovell J, Lush MJ, Lyne R, Martin S, Mashreghi-Mohammadi M, Matthews L, Matthews NS, McLaren S, Milne S, Mistry S, Moore MJ, Nickerson T, O'Dell CN, Oliver K, Palmeiri A, Palmer SA, Parker A, Patel D, Pearce AV, Peck AI, Pelan S, Phelps K, Phillimore BJ, Plumb R, Rajan J, Raymond C, Rouse G, Saenphimmachak C, Sehra HK, Sheridan E, Shownkeen R, Sims S, Skuce CD, Smith M, Steward C, Subramanian S, Sycamore N, Tracey A, Tromans A, Van Helmond Z, Wall M, Wallis JM, White S, Whitehead SL, Wilkinson JE, Willey DL, Williams H, Wilming L, Wray PW, Wu Z, Coulson A, Vaudin M, Sulston JE, Durbin R, Hubbard T, Wooster R, Dunham I, Carter NP, McVean G, Ross MT, Harrow J, Olson MV, Beck S, Rogers J, Bentley DR, Banerjee R, Bryant SP, Burford DC, Burrill WD, Clegg SM, Dhami P, Dovey O, Faulkner LM, Gribble SM, Langford CF, Pandian RD, Porter KM, Prigmore E (May 2006). “The DNA sequence and biological annotation of human chromosome 1”. Nature 441 (7091): 315–21. Bibcode: 2006Natur.441..315G. doi:10.1038/nature04727. PMID 16710414.
- ^ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG (April 1981). “Sequence and organization of the human mitochondrial genome”. Nature 290 (5806): 457–465. Bibcode: 1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534.
- ^ “Untitled”. 2011
年 8月 13日 時点 のオリジナルよりアーカイブ。2012年 6月 13日 閲覧 。 - ^ a b c Satoh M, Kuroiwa T (September 1991). “Organization of multiple nucleoids and DNA molecules in mitochondria of a human cell”. Experimental Cell Research 196 (1): 137–140. doi:10.1016/0014-4827(91)90467-9. PMID 1715276.
- ^ Zhang D, Keilty D, Zhang ZF, Chian RC (March 2017). “Mitochondria in oocyte aging: current understanding”. Facts, Views & Vision in ObGyn 9 (1): 29–38. PMC 5506767. PMID 28721182.
- ^ Designation of the two strands of DNA Archived 24 April 2008 at the Wayback Machine. JCBN/NC-IUB Newsletter 1989. Retrieved 7 May 2008
- ^ Hüttenhofer A, Schattner P, Polacek N (May 2005). “Non-coding RNAs: hope or hype?”. Trends in Genetics 21 (5): 289–97. doi:10.1016/j.tig.2005.03.007. PMID 15851066.
- ^ Munroe SH (November 2004). “Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns”. Journal of Cellular Biochemistry 93 (4): 664–71. doi:10.1002/jcb.20252. PMID 15389973.
- ^ Makalowska I, Lin CF, Makalowski W (February 2005). “Overlapping genes in vertebrate genomes”. Computational Biology and Chemistry 29 (1): 1–12. doi:10.1016/j.compbiolchem.2004.12.006. PMID 15680581.
- ^ Johnson ZI, Chisholm SW (November 2004). “Properties of overlapping genes are conserved across microbial genomes”. Genome Research 14 (11): 2268–72. doi:10.1101/gr.2433104. PMC 525685. PMID 15520290.
- ^ Lamb RA, Horvath CM (August 1991). “Diversity of coding strategies in influenza viruses”. Trends in Genetics 7 (8): 261–66. doi:10.1016/0168-9525(91)90326-L. PMC 7173306. PMID 1771674.
- ^ Benham CJ, Mielke SP (2005). “DNA mechanics”. Annual Review of Biomedical Engineering 7: 21–53. doi:10.1146/annurev.bioeng.6.062403.132016. PMID 16004565. オリジナルの1 March 2019
時点 におけるアーカイブ。. - ^ a b Champoux JJ (2001). “DNA topoisomerases: structure, function, and mechanism”. Annual Review of Biochemistry 70: 369–413. doi:10.1146/annurev.biochem.70.1.369. PMID 11395412.
- ^ a b Wang JC (June 2002). “Cellular roles of DNA topoisomerases: a molecular perspective”. Nature Reviews Molecular Cell Biology 3 (6): 430–40. doi:10.1038/nrm831. PMID 12042765.
- ^ Basu HS, Feuerstein BG, Zarling DA, Shafer RH, Marton LJ (October 1988). “Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies”. Journal of Biomolecular Structure & Dynamics 6 (2): 299–309. doi:10.1080/07391102.1988.10507714. PMID 2482766.
- ^ *Franklin RE, Gosling RG (6 March 1953). “The Structure of Sodium Thymonucleate Fibres I. The Influence of Water Content”. Acta Crystallogr 6 (8–9): 673–77. doi:10.1107/S0365110X53001939. オリジナルの9 January 2016
時点 におけるアーカイブ。.- Franklin RE, Gosling RG (1953). “The structure of sodium thymonucleate fibres. II. The cylindrically symmetrical Patterson function”. Acta Crystallogr 6 (8–9): 678–85. doi:10.1107/S0365110X53001940. オリジナルの2017-06-29
時点 におけるアーカイブ。.
- Franklin RE, Gosling RG (1953). “The structure of sodium thymonucleate fibres. II. The cylindrically symmetrical Patterson function”. Acta Crystallogr 6 (8–9): 678–85. doi:10.1107/S0365110X53001940. オリジナルの2017-06-29
- ^ a b Franklin RE, Gosling RG (April 1953). “Molecular configuration in sodium thymonucleate”. Nature 171 (4356): 740–41. Bibcode: 1953Natur.171..740F. doi:10.1038/171740a0. PMID 13054694. オリジナルの3 January 2011
時点 におけるアーカイブ。. - ^ a b Wilkins MH, Stokes AR, Wilson HR (April 1953). “Molecular structure of deoxypentose nucleic acids”. Nature 171 (4356): 738–40. Bibcode: 1953Natur.171..738W. doi:10.1038/171738a0. PMID 13054693. オリジナルの13 May 2011
時点 におけるアーカイブ。. - ^ Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (October 1980). “Polymorphism of DNA double helices”. Journal of Molecular Biology 143 (1): 49–72. doi:10.1016/0022-2836(80)90124-2. PMID 7441761.
- ^ Baianu IC (1980). “Structural Order and Partial Disorder in Biological systems”. Bull. Math. Biol. 42 (4): 137–41. doi:10.1007/BF02462372.
- ^ Hosemann R, Bagchi RN (1962). Direct analysis of diffraction by matter. Amsterdam – New York: North-Holland Publishers
- ^ Baianu IC (1978). “X-ray scattering by partially disordered membrane systems”. Acta Crystallogr A 34 (5): 751–53. Bibcode: 1978AcCrA..34..751B. doi:10.1107/S0567739478001540. オリジナルの14 March 2020
時点 におけるアーカイブ。 2019年 8月 29日 閲覧 。. - ^ Wahl MC, Sundaralingam M (1997). “Crystal structures of A-DNA duplexes”. Biopolymers 44 (1): 45–63. doi:10.1002/(SICI)1097-0282(1997)44:1<45::AID-BIP4>3.0.CO;2-#. PMID 9097733.
- ^ Lu XJ, Shakked Z, Olson WK (July 2000). “A-form conformational motifs in ligand-bound DNA structures”. Journal of Molecular Biology 300 (4): 819–40. doi:10.1006/jmbi.2000.3690. PMID 10891271.
- ^ Rothenburg S, Koch-Nolte F, Haag F (December 2001). “DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles”. Immunological Reviews 184: 286–98. doi:10.1034/j.1600-065x.2001.1840125.x. PMID 12086319.
- ^ Oh DB, Kim YG, Rich A (December 2002). “Z-DNA-binding proteins can act as potent effectors of gene expression in vivo”. Proceedings of the National Academy of Sciences of the United States of America 99 (26): 16666–71. Bibcode: 2002PNAS...9916666O. doi:10.1073/pnas.262672699. PMC 139201. PMID 12486233.
- ^ Palmer J (2010
年 12月2日 ). “Arsenic-loving bacteria may help in hunt for alien life”. BBC News. オリジナルの2010年 12月3日 時点 におけるアーカイブ。 2010年 12月2日 閲覧 。 - ^ a b Bortman H (2010
年 12月2日 ). “Arsenic-Eating Bacteria Opens New Possibilities for Alien Life”. オリジナルの2010年 12月4日 時点 におけるアーカイブ。 2010年 12月2日 閲覧 。 - ^ Katsnelson A (2 December 2010). “Arsenic-eating microbe may redefine chemistry of life”. Nature News. doi:10.1038/news.2010.645. オリジナルの12 February 2012
時点 におけるアーカイブ。. - ^ Cressey D (3 October 2012). “'Arsenic-life' Bacterium Prefers Phosphorus after all”. Nature News. doi:10.1038/nature.2012.11520.
- ^ “Structure and packing of human telomeric DNA”. ndbserver.rutgers.edu. 2023
年 5月 18日 閲覧 。 - ^ a b Greider CW, Blackburn EH (December 1985). “Identification of a specific telomere terminal transferase activity in Tetrahymena extracts”. Cell 43 (2 Pt 1): 405–13. doi:10.1016/0092-8674(85)90170-9. PMID 3907856.
- ^ a b c Nugent CI, Lundblad V (April 1998). “The telomerase reverse transcriptase: components and regulation”. Genes & Development 12 (8): 1073–85. doi:10.1101/gad.12.8.1073. PMID 9553037.
- ^ Wright WE, Tesmer VM, Huffman KE, Levene SD, Shay JW (November 1997). “Normal human chromosomes have long G-rich telomeric overhangs at one end”. Genes & Development 11 (21): 2801–09. doi:10.1101/gad.11.21.2801. PMC 316649. PMID 9353250.
- ^ a b Burge S, Parkinson GN, Hazel P, Todd AK, Neidle S (2006). “Quadruplex DNA: sequence, topology and structure”. Nucleic Acids Research 34 (19): 5402–15. doi:10.1093/nar/gkl655. PMC 1636468. PMID 17012276.
- ^ Parkinson GN, Lee MP, Neidle S (June 2002). “Crystal structure of parallel quadruplexes from human telomeric DNA”. Nature 417 (6891): 876–80. Bibcode: 2002Natur.417..876P. doi:10.1038/nature755. PMID 12050675.
- ^ Griffith JD, Comeau L, Rosenfield S, Stansel RM, Bianchi A, Moss H, de Lange T (May 1999). “Mammalian telomeres end in a large duplex loop”. Cell 97 (4): 503–14. doi:10.1016/S0092-8674(00)80760-6. PMID 10338214.
- ^ Seeman NC (November 2005). “DNA enables nanoscale control of the structure of matter”. Quarterly Reviews of Biophysics 38 (4): 363–71. doi:10.1017/S0033583505004087. PMC 3478329. PMID 16515737.
- ^ Warren M (21 February 2019). “Four new DNA letters double life's alphabet”. Nature 566 (7745): 436. Bibcode: 2019Natur.566..436W. doi:10.1038/d41586-019-00650-8. PMID 30809059.
- ^ Hoshika S, Leal NA, Kim MJ, Kim MS, Karalkar NB, Kim HJ, Bates AM, Watkins NE, SantaLucia HA, Meyer AJ, DasGupta S, Piccirilli JA, Ellington AD, SantaLucia J, Georgiadis MM, Benner SA (22 February 2019). “Hachimoji DNA and RNA: A genetic system with eight building blocks (paywall)”. Science 363 (6429): 884–887. Bibcode: 2019Sci...363..884H. doi:10.1126/science.aat0971. PMC 6413494. PMID 30792304.
- ^ Burghardt B, Hartmann AK (February 2007). “RNA secondary structure design”. Physical Review E 75 (2): 021920. arXiv:physics/0609135. Bibcode: 2007PhRvE..75b1920B. doi:10.1103/PhysRevE.75.021920. PMID 17358380.
- ^ Reusch W. “Nucleic Acids”. Michigan State University. 2022
年 6月 30日 閲覧 。 - ^ “How To Extract DNA From Anything Living”. University of Utah. 2022
年 6月 30日 閲覧 。 - ^ Hu Q, Rosenfeld MG (2012). “Epigenetic regulation of human embryonic stem cells”. Frontiers in Genetics 3: 238. doi:10.3389/fgene.2012.00238. PMC 3488762. PMID 23133442.
- ^ Klose RJ, Bird AP (February 2006). “Genomic DNA methylation: the mark and its mediators”. Trends in Biochemical Sciences 31 (2): 89–97. doi:10.1016/j.tibs.2005.12.008. PMID 16403636.
- ^ Bird A (January 2002). “DNA methylation patterns and epigenetic memory”. Genes & Development 16 (1): 6–21. doi:10.1101/gad.947102. PMID 11782440.
- ^ Walsh CP, Xu GL (2006). “Cytosine methylation and DNA repair”. Current Topics in Microbiology and Immunology 301: 283–315. doi:10.1007/3-540-31390-7_11. ISBN 3-540-29114-8. PMID 16570853.
- ^ Kriaucionis S, Heintz N (May 2009). “The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain”. Science 324 (5929): 929–30. Bibcode: 2009Sci...324..929K. doi:10.1126/science.1169786. PMC 3263819. PMID 19372393.
- ^ Ratel D, Ravanat JL, Berger F, Wion D (March 2006). “N6-methyladenine: the other methylated base of DNA”. BioEssays 28 (3): 309–15. doi:10.1002/bies.20342. PMC 2754416. PMID 16479578.
- ^ Gommers-Ampt JH, Van Leeuwen F, de Beer AL, Vliegenthart JF, Dizdaroglu M, Kowalak JA, Crain PF, Borst P (December 1993). “beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei”. Cell 75 (6): 1129–36. doi:10.1016/0092-8674(93)90322-H. hdl:1874/5219. PMID 8261512.
- ^ Created from PDB 1JDG Archived 22 September 2008 at the Wayback Machine.
- ^ Douki T, Reynaud-Angelin A, Cadet J, Sage E (August 2003). “Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation”. Biochemistry 42 (30): 9221–26. doi:10.1021/bi034593c. PMID 12885257.
- ^ Cadet J, Delatour T, Douki T, Gasparutto D, Pouget JP, Ravanat JL, Sauvaigo S (March 1999). “Hydroxyl radicals and DNA base damage”. Mutation Research 424 (1–2): 9–21. doi:10.1016/S0027-5107(99)00004-4. PMID 10064846.
- ^ Beckman KB, Ames BN (August 1997). “Oxidative decay of DNA”. The Journal of Biological Chemistry 272 (32): 19633–36. doi:10.1074/jbc.272.32.19633. PMID 9289489.
- ^ Valerie K, Povirk LF (September 2003). “Regulation and mechanisms of mammalian double-strand break repair”. Oncogene 22 (37): 5792–812. doi:10.1038/sj.onc.1206679. PMID 12947387.
- ^ Johnson G (2010
年 12月28日 ). “Unearthing Prehistoric Tumors, and Debate”. The New York Times. オリジナルの2017年 6月 24日 時点 におけるアーカイブ。. "If we lived long enough, sooner or later we all would get cancer." - ^ Alberts B, Johnson A, Lewis J (2002). “The Preventable Causes of Cancer”. Molecular biology of the cell (4th ed.). New York: Garland Science. ISBN 0-8153-4072-9. オリジナルの2 January 2016
時点 におけるアーカイブ。. "A certain irreducible background incidence of cancer is to be expected regardless of circumstances: mutations can never be absolutely avoided, because they are an inescapable consequence of fundamental limitations on the accuracy of DNA replication, as discussed in Chapter 5. If a human could live long enough, it is inevitable that at least one of his or her cells would eventually accumulate a set of mutations sufficient for cancer to develop." - ^ Bernstein H, Payne CM, Bernstein C, Garewal H, Dvorak K (2008). “Cancer and aging as consequences of un-repaired DNA damage”. New Research on DNA Damage. New York: Nova Science Publishers. pp. 1–47. ISBN 978-1-60456-581-2. オリジナルの25 October 2014
時点 におけるアーカイブ。 - ^ Hoeijmakers JH (October 2009). “DNA damage, aging, and cancer”. The New England Journal of Medicine 361 (15): 1475–85. doi:10.1056/NEJMra0804615. PMID 19812404.
- ^ Freitas AA, de Magalhães JP (2011). “A review and appraisal of the DNA damage theory of ageing”. Mutation Research 728 (1–2): 12–22. doi:10.1016/j.mrrev.2011.05.001. PMID 21600302.
- ^ Ferguson LR, Denny WA (September 1991). “The genetic toxicology of acridines”. Mutation Research 258 (2): 123–60. doi:10.1016/0165-1110(91)90006-H. PMID 1881402.
- ^ Stephens TD, Bunde CJ, Fillmore BJ (June 2000). “Mechanism of action in thalidomide teratogenesis”. Biochemical Pharmacology 59 (12): 1489–99. doi:10.1016/S0006-2952(99)00388-3. PMID 10799645.
- ^ Jeffrey AM (1985). “DNA modification by chemical carcinogens”. Pharmacology & Therapeutics 28 (2): 237–72. doi:10.1016/0163-7258(85)90013-0. PMID 3936066.
- ^ Braña MF, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A (November 2001). “Intercalators as anticancer drugs”. Current Pharmaceutical Design 7 (17): 1745–80. doi:10.2174/1381612013397113. PMID 11562309.
- ^ Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, Smith HO, Yandell M, Evans CA, Holt RA, Gocayne JD, Amanatides P, Ballew RM, Huson DH, Wortman JR, Zhang Q, Kodira CD, Zheng XH, Chen L, Skupski M, Subramanian G, Thomas PD, Zhang J, Gabor Miklos GL, Nelson C, Broder S, Clark AG, Nadeau J, McKusick VA, Zinder N, Levine AJ, Roberts RJ, Simon M, Slayman C, Hunkapiller M, Bolanos R, Delcher A, Dew I, Fasulo D, Flanigan M, Florea L, Halpern A, Hannenhalli S, Kravitz S, Levy S, Mobarry C, Reinert K, Remington K, Abu-Threideh J, Beasley E, Biddick K, Bonazzi V, Brandon R, Cargill M, Chandramouliswaran I, Charlab R, Chaturvedi K, Deng Z, Di Francesco V, Dunn P, Eilbeck K, Evangelista C, Gabrielian AE, Gan W, Ge W, Gong F, Gu Z, Guan P, Heiman TJ, Higgins ME, Ji RR, Ke Z, Ketchum KA, Lai Z, Lei Y, Li Z, Li J, Liang Y, Lin X, Lu F, Merkulov GV, Milshina N, Moore HM, Naik AK, Narayan VA, Neelam B, Nusskern D, Rusch DB, Salzberg S, Shao W, Shue B, Sun J, Wang Z, Wang A, Wang X, Wang J, Wei M, Wides R, Xiao C, Yan C, Yao A, Ye J, Zhan M, Zhang W, Zhang H, Zhao Q, Zheng L, Zhong F, Zhong W, Zhu S, Zhao S, Gilbert D, Baumhueter S, Spier G, Carter C, Cravchik A, Woodage T, Ali F, An H, Awe A, Baldwin D, Baden H, Barnstead M, Barrow I, Beeson K, Busam D, Carver A, Center A, Cheng ML, Curry L, Danaher S, Davenport L, Desilets R, Dietz S, Dodson K, Doup L, Ferriera S, Garg N, Gluecksmann A, Hart B, Haynes J, Haynes C, Heiner C, Hladun S, Hostin D, Houck J, Howland T, Ibegwam C, Johnson J, Kalush F, Kline L, Koduru S, Love A, Mann F, May D, McCawley S, McIntosh T, McMullen I, Moy M, Moy L, Murphy B, Nelson K, Pfannkoch C, Pratts E, Puri V, Qureshi H, Reardon M, Rodriguez R, Rogers YH, Romblad D, Ruhfel B, Scott R, Sitter C, Smallwood M, Stewart E, Strong R, Suh E, Thomas R, Tint NN, Tse S, Vech C, Wang G, Wetter J, Williams S, Williams M, Windsor S, Winn-Deen E, Wolfe K, Zaveri J, Zaveri K, Abril JF, Guigó R, Campbell MJ, Sjolander KV, Karlak B, Kejariwal A, Mi H, Lazareva B, Hatton T, Narechania A, Diemer K, Muruganujan A, Guo N, Sato S, Bafna V, Istrail S, Lippert R, Schwartz R, Walenz B, Yooseph S, Allen D, Basu A, Baxendale J, Blick L, Caminha M, Carnes-Stine J, Caulk P, Chiang YH, Coyne M, Dahlke C, Mays A, Dombroski M, Donnelly M, Ely D, Esparham S, Fosler C, Gire H, Glanowski S, Glasser K, Glodek A, Gorokhov M, Graham K, Gropman B, Harris M, Heil J, Henderson S, Hoover J, Jennings D, Jordan C, Jordan J, Kasha J, Kagan L, Kraft C, Levitsky A, Lewis M, Liu X, Lopez J, Ma D, Majoros W, McDaniel J, Murphy S, Newman M, Nguyen T, Nguyen N, Nodell M, Pan S, Peck J, Peterson M, Rowe W, Sanders R, Scott J, Simpson M, Smith T, Sprague A, Stockwell T, Turner R, Venter E, Wang M, Wen M, Wu D, Wu M, Xia A, Zandieh A, Zhu X (February 2001). “The sequence of the human genome”. Science 291 (5507): 1304–51. Bibcode: 2001Sci...291.1304V. doi:10.1126/science.1058040. PMID 11181995.
- ^ Thanbichler M, Wang SC, Shapiro L (October 2005). “The bacterial nucleoid: a highly organized and dynamic structure”. Journal of Cellular Biochemistry 96 (3): 506–21. doi:10.1002/jcb.20519. PMID 15988757.
- ^ Wolfsberg TG, McEntyre J, Schuler GD (February 2001). “Guide to the draft human genome”. Nature 409 (6822): 824–26. Bibcode: 2001Natur.409..824W. doi:10.1038/35057000. PMID 11236998.
- ^ Gregory TR (January 2005). “The C-value enigma in plants and animals: a review of parallels and an appeal for partnership”. Annals of Botany 95 (1): 133–46. doi:10.1093/aob/mci009. PMC 4246714. PMID 15596463.
- ^ Birney E, Stamatoyannopoulos JA, Dutta A, Guigó R, Gingeras TR, Margulies EH, Weng Z, Snyder M, Dermitzakis ET, Thurman RE, Kuehn MS, Taylor CM, Neph S, Koch CM, Asthana S, Malhotra A, Adzhubei I, Greenbaum JA, Andrews RM, Flicek P, Boyle PJ, Cao H, Carter NP, Clelland GK, Davis S, Day N, Dhami P, Dillon SC, Dorschner MO, Fiegler H, Giresi PG, Goldy J, Hawrylycz M, Haydock A, Humbert R, James KD, Johnson BE, Johnson EM, Frum TT, Rosenzweig ER, Karnani N, Lee K, Lefebvre GC, Navas PA, Neri F, Parker SC, Sabo PJ, Sandstrom R, Shafer A, Vetrie D, Weaver M, Wilcox S, Yu M, Collins FS, Dekker J, Lieb JD, Tullius TD, Crawford GE, Sunyaev S, Noble WS, Dunham I, Denoeud F, Reymond A, Kapranov P, Rozowsky J, Zheng D, Castelo R, Frankish A, Harrow J, Ghosh S, Sandelin A, Hofacker IL, Baertsch R, Keefe D, Dike S, Cheng J, Hirsch HA, Sekinger EA, Lagarde J, Abril JF, Shahab A, Flamm C, Fried C, Hackermüller J, Hertel J, Lindemeyer M, Missal K, Tanzer A, Washietl S, Korbel J, Emanuelsson O, Pedersen JS, Holroyd N, Taylor R, Swarbreck D, Matthews N, Dickson MC, Thomas DJ, Weirauch MT, Gilbert J, Drenkow J, Bell I, Zhao X, Srinivasan KG, Sung WK, Ooi HS, Chiu KP, Foissac S, Alioto T, Brent M, Pachter L, Tress ML, Valencia A, Choo SW, Choo CY, Ucla C, Manzano C, Wyss C, Cheung E, Clark TG, Brown JB, Ganesh M, Patel S, Tammana H, Chrast J, Henrichsen CN, Kai C, Kawai J, Nagalakshmi U, Wu J, Lian Z, Lian J, Newburger P, Zhang X, Bickel P, Mattick JS, Carninci P, Hayashizaki Y, Weissman S, Hubbard T, Myers RM, Rogers J, Stadler PF, Lowe TM, Wei CL, Ruan Y, Struhl K, Gerstein M, Antonarakis SE, Fu Y, Green ED, Karaöz U, Siepel A, Taylor J, Liefer LA, Wetterstrand KA, Good PJ, Feingold EA, Guyer MS, Cooper GM, Asimenos G, Dewey CN, Hou M, Nikolaev S, Montoya-Burgos JI, Löytynoja A, Whelan S, Pardi F, Massingham T, Huang H, Zhang NR, Holmes I, Mullikin JC, Ureta-Vidal A, Paten B, Seringhaus M, Church D, Rosenbloom K, Kent WJ, Stone EA, Batzoglou S, Goldman N, Hardison RC, Haussler D, Miller W, Sidow A, Trinklein ND, Zhang ZD, Barrera L, Stuart R, King DC, Ameur A, Enroth S, Bieda MC, Kim J, Bhinge AA, Jiang N, Liu J, Yao F, Vega VB, Lee CW, Ng P, Shahab A, Yang A, Moqtaderi Z, Zhu Z, Xu X, Squazzo S, Oberley MJ, Inman D, Singer MA, Richmond TA, Munn KJ, Rada-Iglesias A, Wallerman O, Komorowski J, Fowler JC, Couttet P, Bruce AW, Dovey OM, Ellis PD, Langford CF, Nix DA, Euskirchen G, Hartman S, Urban AE, Kraus P, Van Calcar S, Heintzman N, Kim TH, Wang K, Qu C, Hon G, Luna R, Glass CK, Rosenfeld MG, Aldred SF, Cooper SJ, Halees A, Lin JM, Shulha HP, Zhang X, Xu M, Haidar JN, Yu Y, Ruan Y, Iyer VR, Green RD, Wadelius C, Farnham PJ, Ren B, Harte RA, Hinrichs AS, Trumbower H, Clawson H, Hillman-Jackson J, Zweig AS, Smith K, Thakkapallayil A, Barber G, Kuhn RM, Karolchik D, Armengol L, Bird CP, de Bakker PI, Kern AD, Lopez-Bigas N, Martin JD, Stranger BE, Woodroffe A, Davydov E, Dimas A, Eyras E, Hallgrímsdóttir IB, Huppert J, Zody MC, Abecasis GR, Estivill X, Bouffard GG, Guan X, Hansen NF, Idol JR, Maduro VV, Maskeri B, McDowell JC, Park M, Thomas PJ, Young AC, Blakesley RW, Muzny DM, Sodergren E, Wheeler DA, Worley KC, Jiang H, Weinstock GM, Gibbs RA, Graves T, Fulton R, Mardis ER, Wilson RK, Clamp M, Cuff J, Gnerre S, Jaffe DB, Chang JL, Lindblad-Toh K, Lander ES, Koriabine M, Nefedov M, Osoegawa K, Yoshinaga Y, Zhu B, de Jong PJ (June 2007). “Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project”. Nature 447 (7146): 799–816. Bibcode: 2007Natur.447..799B. doi:10.1038/nature05874. PMC 2212820. PMID 17571346.
- ^ Yin YW, Steitz TA. “RCSB PDB – 1MSW: Structural basis for the transition from initiation to elongation transcription in T7 RNA polymerase” (
英語 ). www.rcsb.org. 2023年 3月 27日 閲覧 。 - ^ Pidoux AL, Allshire RC (March 2005). “The role of heterochromatin in centromere function”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 360 (1455): 569–79. doi:10.1098/rstb.2004.1611. PMC 1569473. PMID 15905142.
- ^ Harrison PM, Hegyi H, Balasubramanian S, Luscombe NM, Bertone P, Echols N, Johnson T, Gerstein M (February 2002). “Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22”. Genome Research 12 (2): 272–80. doi:10.1101/gr.207102. PMC 155275. PMID 11827946.
- ^ Harrison PM, Gerstein M (May 2002). “Studying genomes through the aeons: protein families, pseudogenes and proteome evolution”. Journal of Molecular Biology 318 (5): 1155–74. doi:10.1016/S0022-2836(02)00109-2. PMID 12083509.
- ^ Albà M (2001). “Replicative DNA polymerases”. Genome Biology 2 (1): REVIEWS3002. doi:10.1186/gb-2001-2-1-reviews3002. PMC 150442. PMID 11178285.
- ^ Tani K, Nasu M (2010). “Roles of Extracellular DNA in Bacterial Ecosystems”. Extracellular Nucleic Acids. Springer. pp. 25–38. ISBN 978-3-642-12616-1
- ^ Vlassov VV, Laktionov PP, Rykova EY (July 2007). “Extracellular nucleic acids”. BioEssays 29 (7): 654–67. doi:10.1002/bies.20604. PMID 17563084.
- ^ Finkel SE, Kolter R (November 2001). “DNA as a nutrient: novel role for bacterial competence gene homologs”. Journal of Bacteriology 183 (21): 6288–93. doi:10.1128/JB.183.21.6288-6293.2001. PMC 100116. PMID 11591672.
- ^ Mulcahy H, Charron-Mazenod L, Lewenza S (November 2008). “Extracellular DNA chelates cations and induces antibiotic resistance in Pseudomonas aeruginosa biofilms”. PLOS Pathogens 4 (11): e1000213. doi:10.1371/journal.ppat.1000213. PMC 2581603. PMID 19023416.
- ^ Berne C, Kysela DT, Brun YV (August 2010). “A bacterial extracellular DNA inhibits settling of motile progeny cells within a biofilm”. Molecular Microbiology 77 (4): 815–29. doi:10.1111/j.1365-2958.2010.07267.x. PMC 2962764. PMID 20598083.
- ^ Whitchurch CB, Tolker-Nielsen T, Ragas PC, Mattick JS (February 2002). “Extracellular DNA required for bacterial biofilm formation”. Science 295 (5559): 1487. doi:10.1126/science.295.5559.1487. PMID 11859186.
- ^ Hu W, Li L, Sharma S, Wang J, McHardy I, Lux R, Yang Z, He X, Gimzewski JK, Li Y, Shi W (2012). “DNA builds and strengthens the extracellular matrix in Myxococcus xanthus biofilms by interacting with exopolysaccharides”. PLOS ONE 7 (12): e51905. Bibcode: 2012PLoSO...751905H. doi:10.1371/journal.pone.0051905. PMC 3530553. PMID 23300576.
- ^ Hui L, Bianchi DW (February 2013). “Recent advances in the prenatal interrogation of the human fetal genome”. Trends in Genetics 29 (2): 84–91. doi:10.1016/j.tig.2012.10.013. PMC 4378900. PMID 23158400.
- ^ Foote AD, Thomsen PF, Sveegaard S, Wahlberg M, Kielgast J, Kyhn LA, Salling AB, Galatius A, Orlando L, Gilbert MT (2012). “Investigating the potential use of environmental DNA (eDNA) for genetic monitoring of marine mammals”. PLOS ONE 7 (8): e41781. Bibcode: 2012PLoSO...741781F. doi:10.1371/journal.pone.0041781. PMC 3430683. PMID 22952587.
- ^ “Researchers Detect Land Animals Using DNA in Nearby Water Bodies”. 2020
年 5月 24日 閲覧 。 - ^ Sandman K, Pereira SL, Reeve JN (December 1998). “Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome”. Cellular and Molecular Life Sciences 54 (12): 1350–64. doi:10.1007/s000180050259. PMID 9893710.
- ^ Dame RT (May 2005). “The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin”. Molecular Microbiology 56 (4): 858–70. doi:10.1111/j.1365-2958.2005.04598.x. PMID 15853876.
- ^ Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ (September 1997). “Crystal structure of the nucleosome core particle at 2.8 A resolution”. Nature 389 (6648): 251–60. Bibcode: 1997Natur.389..251L. doi:10.1038/38444. PMID 9305837.
- ^ Jenuwein T, Allis CD (August 2001). “Translating the histone code”. Science 293 (5532): 1074–80. doi:10.1126/science.1063127. PMID 11498575. オリジナルの8 August 2017
時点 におけるアーカイブ。. - ^ Ito T (2003). “Nucleosome Assembly and Remodeling”. Protein Complexes that Modify Chromatin. Current Topics in Microbiology and Immunology. 274. pp. 1–22. doi:10.1007/978-3-642-55747-7_1. ISBN 978-3-540-44208-0. PMID 12596902
- ^ Thomas JO (August 2001). “HMG1 and 2: architectural DNA-binding proteins”. Biochemical Society Transactions 29 (Pt 4): 395–401. doi:10.1042/BST0290395. PMID 11497996.
- ^ Grosschedl R, Giese K, Pagel J (March 1994). “HMG domain proteins: architectural elements in the assembly of nucleoprotein structures”. Trends in Genetics 10 (3): 94–100. doi:10.1016/0168-9525(94)90232-1. PMID 8178371.
- ^ Iftode C, Daniely Y, Borowiec JA (1999). “Replication protein A (RPA): the eukaryotic SSB”. Critical Reviews in Biochemistry and Molecular Biology 34 (3): 141–80. doi:10.1080/10409239991209255. PMID 10473346.
- ^ Beamer LJ, Pabo CO. “RCSB PDB – 1LMB: Refined 1.8 Å crystal structure of the lambda repressor-operator complex” (
英語 ). www.rcsb.org. 2023年 3月 27日 閲覧 。 - ^ Myers LC, Kornberg RD (2000). “Mediator of transcriptional regulation”. Annual Review of Biochemistry 69: 729–49. doi:10.1146/annurev.biochem.69.1.729. PMID 10966474.
- ^ Spiegelman BM, Heinrich R (October 2004). “Biological control through regulated transcriptional coactivators”. Cell 119 (2): 157–67. doi:10.1016/j.cell.2004.09.037. PMID 15479634.
- ^ Li Z, Van Calcar S, Qu C, Cavenee WK, Zhang MQ, Ren B (July 2003). “A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells”. Proceedings of the National Academy of Sciences of the United States of America 100 (14): 8164–69. Bibcode: 2003PNAS..100.8164L. doi:10.1073/pnas.1332764100. PMC 166200. PMID 12808131.
- ^ Kostrewa D, Winkler FK. “RCSB PDB – 1RVA: Mg2+ binding to the active site of EcoRV endonuclease: a crystallographic study of complexes with substrate and product DNA at 2 Å resolution” (
英語 ). www.rcsb.org. 2023年 3月 27日 閲覧 。 - ^ Bickle TA, Krüger DH (June 1993). “Biology of DNA restriction”. Microbiological Reviews 57 (2): 434–50. doi:10.1128/MMBR.57.2.434-450.1993. PMC 372918. PMID 8336674.
- ^ a b Doherty AJ, Suh SW (November 2000). “Structural and mechanistic conservation in DNA ligases”. Nucleic Acids Research 28 (21): 4051–58. doi:10.1093/nar/28.21.4051. PMC 113121. PMID 11058099.
- ^ Schoeffler AJ, Berger JM (December 2005). “Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism”. Biochemical Society Transactions 33 (Pt 6): 1465–70. doi:10.1042/BST20051465. PMID 16246147.
- ^ Tuteja N, Tuteja R (May 2004). “Unraveling DNA helicases. Motif, structure, mechanism and function”. European Journal of Biochemistry 271 (10): 1849–63. doi:10.1111/j.1432-1033.2004.04094.x. PMID 15128295.
- ^ Joyce CM, Steitz TA (November 1995). “Polymerase structures and function: variations on a theme?”. Journal of Bacteriology 177 (22): 6321–29. doi:10.1128/jb.177.22.6321-6329.1995. PMC 177480. PMID 7592405.
- ^ Hubscher U, Maga G, Spadari S (2002). “Eukaryotic DNA polymerases”. Annual Review of Biochemistry 71: 133–63. doi:10.1146/annurev.biochem.71.090501.150041. PMID 12045093. オリジナルの26 January 2021
時点 におけるアーカイブ。. - ^ Johnson A, O'Donnell M (2005). “Cellular DNA replicases: components and dynamics at the replication fork”. Annual Review of Biochemistry 74: 283–315. doi:10.1146/annurev.biochem.73.011303.073859. PMID 15952889.
- ^ a b Tarrago-Litvak L, Andréola ML, Nevinsky GA, Sarih-Cottin L, Litvak S (May 1994). “The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention”. FASEB Journal 8 (8): 497–503. doi:10.1096/fasebj.8.8.7514143. PMID 7514143.
- ^ Martinez E (December 2002). “Multi-protein complexes in eukaryotic gene transcription”. Plant Molecular Biology 50 (6): 925–47. doi:10.1023/A:1021258713850. PMID 12516863.
- ^ Thorpe JH, Gale BC, Teixeira SC, Cardin CJ. “RCSB PDB – 1M6G: Structural Characterisation of the Holliday Junction TCGGTACCGA” (
英語 ). www.rcsb.org. 2023年 3月 27日 閲覧 。 - ^ Cremer T, Cremer C (April 2001). “Chromosome territories, nuclear architecture and gene regulation in mammalian cells”. Nature Reviews Genetics 2 (4): 292–301. doi:10.1038/35066075. PMID 11283701.
- ^ Pál C, Papp B, Lercher MJ (May 2006). “An integrated view of protein evolution”. Nature Reviews Genetics 7 (5): 337–48. doi:10.1038/nrg1838. PMID 16619049.
- ^ O'Driscoll M, Jeggo PA (January 2006). “The role of double-strand break repair – insights from human genetics”. Nature Reviews Genetics 7 (1): 45–54. doi:10.1038/nrg1746. PMID 16369571.
- ^ Vispé S, Defais M (October 1997). “Mammalian Rad51 protein: a RecA homologue with pleiotropic functions”. Biochimie 79 (9–10): 587–92. doi:10.1016/S0300-9084(97)82007-X. PMID 9466696.
- ^ Neale MJ, Keeney S (July 2006). “Clarifying the mechanics of DNA strand exchange in meiotic recombination”. Nature 442 (7099): 153–58. Bibcode: 2006Natur.442..153N. doi:10.1038/nature04885. PMC 5607947. PMID 16838012.
- ^ Dickman MJ, Ingleston SM, Sedelnikova SE, Rafferty JB, Lloyd RG, Grasby JA, Hornby DP (November 2002). “The RuvABC resolvasome”. European Journal of Biochemistry 269 (22): 5492–501. doi:10.1046/j.1432-1033.2002.03250.x. PMID 12423347.
- ^ Joyce GF (July 2002). “The antiquity of RNA-based evolution”. Nature 418 (6894): 214–21. Bibcode: 2002Natur.418..214J. doi:10.1038/418214a. PMID 12110897.
- ^ Orgel LE (2004). “Prebiotic chemistry and the origin of the RNA world”. Critical Reviews in Biochemistry and Molecular Biology 39 (2): 99–123. doi:10.1080/10409230490460765. PMID 15217990.
- ^ Davenport RJ (May 2001). “Ribozymes. Making copies in the RNA world”. Science 292 (5520): 1278a–1278. doi:10.1126/science.292.5520.1278a. PMID 11360970.
- ^ Szathmáry E (April 1992). “What is the optimum size for the genetic alphabet?”. Proceedings of the National Academy of Sciences of the United States of America 89 (7): 2614–18. Bibcode: 1992PNAS...89.2614S. doi:10.1073/pnas.89.7.2614. PMC 48712. PMID 1372984.
- ^ Lindahl T (April 1993). “Instability and decay of the primary structure of DNA”. Nature 362 (6422): 709–15. Bibcode: 1993Natur.362..709L. doi:10.1038/362709a0. PMID 8469282.
- ^ Vreeland RH, Rosenzweig WD, Powers DW (October 2000). “Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal”. Nature 407 (6806): 897–900. Bibcode: 2000Natur.407..897V. doi:10.1038/35038060. PMID 11057666.
- ^ Hebsgaard MB, Phillips MJ, Willerslev E (May 2005). “Geologically ancient DNA: fact or artefact?”. Trends in Microbiology 13 (5): 212–20. doi:10.1016/j.tim.2005.03.010. PMID 15866038.
- ^ Nickle DC, Learn GH, Rain MW, Mullins JI, Mittler JE (January 2002). “Curiously modern DNA for a "250 million-year-old" bacterium”. Journal of Molecular Evolution 54 (1): 134–37. Bibcode: 2002JMolE..54..134N. doi:10.1007/s00239-001-0025-x. PMID 11734907.
- ^ Callahan MP, Smith KE, Cleaves HJ, Ruzicka J, Stern JC, Glavin DP, House CH, Dworkin JP (August 2011). “Carbonaceous meteorites contain a wide range of extraterrestrial nucleobases”. Proceedings of the National Academy of Sciences of the United States of America 108 (34): 13995–98. Bibcode: 2011PNAS..10813995C. doi:10.1073/pnas.1106493108. PMC 3161613. PMID 21836052.
- ^ Steigerwald J (2011
年 8月 8日 ). “NASA Researchers: DNA Building Blocks Can Be Made in Space”. NASA. 2015年 6月 23日 時点 のオリジナルよりアーカイブ。2011年 8月 10日 閲覧 。 - ^ ScienceDaily Staff (2011
年 8月 9日 ). “DNA Building Blocks Can Be Made in Space, NASA Evidence Suggests”. ScienceDaily. 2011年 9月 5日 時点 のオリジナルよりアーカイブ。2011年 8月 9日 閲覧 。 - ^ Marlaire R (2015
年 3月 3日 ). “NASA Ames Reproduces the Building Blocks of Life in Laboratory”. NASA. 2015年 3月 5日 時点 のオリジナルよりアーカイブ。2015年 3月 5日 閲覧 。 - ^ Hunt K (2021
年 2月 17日 ). “World's oldest DNA sequenced from a mammoth that lived more than a million years ago”. CNN News 2021年 2月 17日 閲覧 。 - ^ Callaway E (17 February 2021). “Million-year-old mammoth genomes shatter record for oldest ancient DNA – Permafrost-preserved teeth, up to 1.6 million years old, identify a new kind of mammoth in Siberia.”. Nature 590 (7847): 537–538. Bibcode: 2021Natur.590..537C. doi:10.1038/d41586-021-00436-x. ISSN 0028-0836. PMID 33597786.
- ^ Goff SP, Berg P (December 1976). “Construction of hybrid viruses containing SV40 and lambda phage DNA segments and their propagation in cultured monkey cells”. Cell 9 (4 PT 2): 695–705. doi:10.1016/0092-8674(76)90133-1. PMID 189942.
- ^ Houdebine LM (2007). “Transgenic animal models in biomedical research”. Target Discovery and Validation Reviews and Protocols. Methods in Molecular Biology. 360. pp. 163–202. doi:10.1385/1-59745-165-7:163. ISBN 978-1-59745-165-9. PMID 17172731
- ^ Daniell H, Dhingra A (April 2002). “Multigene engineering: dawn of an exciting new era in biotechnology”. Current Opinion in Biotechnology 13 (2): 136–41. doi:10.1016/S0958-1669(02)00297-5. PMC 3481857. PMID 11950565.
- ^ Job D (November 2002). “Plant biotechnology in agriculture”. Biochimie 84 (11): 1105–10. doi:10.1016/S0300-9084(02)00013-5. PMID 12595138.
- ^ Curtis C, Hereward J (2017
年 8月 29日 ). “From the crime scene to the courtroom: the journey of a DNA sample”. The Conversation. オリジナルの2017年 10月 22日 時点 におけるアーカイブ。 2017年 10月 22日 閲覧 。 - ^ Collins A, Morton NE (June 1994). “Likelihood ratios for DNA identification”. Proceedings of the National Academy of Sciences of the United States of America 91 (13): 6007–11. Bibcode: 1994PNAS...91.6007C. doi:10.1073/pnas.91.13.6007. PMC 44126. PMID 8016106.
- ^ Weir BS, Triggs CM, Starling L, Stowell LI, Walsh KA, Buckleton J (March 1997). “Interpreting DNA mixtures”. Journal of Forensic Sciences 42 (2): 213–22. doi:10.1520/JFS14100J. PMID 9068179.
- ^ Jeffreys AJ, Wilson V, Thein SL (1985). “Individual-specific 'fingerprints' of human DNA”. Nature 316 (6023): 76–79. Bibcode: 1985Natur.316...76J. doi:10.1038/316076a0. PMID 2989708.
- ^ “Colin Pitchfork” (2006
年 12月14日 ). 2006年 12月14日 時点 のオリジナルよりアーカイブ。2023年 3月 27日 閲覧 。 - ^ “DNA Identification in Mass Fatality Incidents”. National Institute of Justice (2006
年 9月 ). 2006年 11月12日 時点 のオリジナルよりアーカイブ。2007年 1月 1日 閲覧 。 - ^ Pollack A (2012
年 6月 19日 ). “Before Birth, Dad's ID” (英語 ). The New York Times. ISSN 0362-4331. オリジナルの2017年 6月 24日 時点 におけるアーカイブ。 2023年 3月 27日 閲覧 。 - ^ a b Breaker RR, Joyce GF (December 1994). “A DNA enzyme that cleaves RNA”. Chemistry & Biology 1 (4): 223–29. doi:10.1016/1074-5521(94)90014-0. PMID 9383394.
- ^ Chandra M, Sachdeva A, Silverman SK (October 2009). “DNA-catalyzed sequence-specific hydrolysis of DNA”. Nature Chemical Biology 5 (10): 718–20. doi:10.1038/nchembio.201. PMC 2746877. PMID 19684594.
- ^ Carmi N, Shultz LA, Breaker RR (December 1996). “In vitro selection of self-cleaving DNAs”. Chemistry & Biology 3 (12): 1039–46. doi:10.1016/S1074-5521(96)90170-2. PMID 9000012.
- ^ Torabi SF, Wu P, McGhee CE, Chen L, Hwang K, Zheng N, Cheng J, Lu Y (May 2015). “In vitro selection of a sodium-specific DNAzyme and its application in intracellular sensing”. Proceedings of the National Academy of Sciences of the United States of America 112 (19): 5903–08. Bibcode: 2015PNAS..112.5903T. doi:10.1073/pnas.1420361112. PMC 4434688. PMID 25918425.
- ^ Baldi P, Brunak S (2001). Bioinformatics: The Machine Learning Approach. MIT Press. ISBN 978-0-262-02506-5. OCLC 45951728
- ^ Gusfield D (15 January 1997). Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology. Cambridge University Press. ISBN 978-0-521-58519-4
- ^ Sjölander K (January 2004). “Phylogenomic inference of protein molecular function: advances and challenges”. Bioinformatics 20 (2): 170–79. doi:10.1093/bioinformatics/bth021. PMID 14734307.
- ^ Mount DM (2004). Bioinformatics: Sequence and Genome Analysis (2nd ed.). Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. ISBN 0-87969-712-1. OCLC 55106399
- ^ Strong M (March 2004). “Protein nanomachines”. PLOS Biology 2 (3): E73. doi:10.1371/journal.pbio.0020073. PMC 368168. PMID 15024422.
- ^ Rothemund PW (March 2006). “Folding DNA to create nanoscale shapes and patterns”. Nature 440 (7082): 297–302. Bibcode: 2006Natur.440..297R. doi:10.1038/nature04586. PMID 16541064.
- ^ Andersen ES, Dong M, Nielsen MM, Jahn K, Subramani R, Mamdouh W, Golas MM, Sander B, Stark H, Oliveira CL, Pedersen JS, Birkedal V, Besenbacher F, Gothelf KV, Kjems J (May 2009). “Self-assembly of a nanoscale DNA box with a controllable lid”. Nature 459 (7243): 73–76. Bibcode: 2009Natur.459...73A. doi:10.1038/nature07971. hdl:11858/00-001M-0000-0010-9362-B. PMID 19424153.
- ^ Ishitsuka Y, Ha T (May 2009). “DNA nanotechnology: a nanomachine goes live”. Nature Nanotechnology 4 (5): 281–82. Bibcode: 2009NatNa...4..281I. doi:10.1038/nnano.2009.101. PMID 19421208.
- ^ Aldaye FA, Palmer AL, Sleiman HF (September 2008). “Assembling materials with DNA as the guide”. Science 321 (5897): 1795–99. Bibcode: 2008Sci...321.1795A. doi:10.1126/science.1154533. PMID 18818351.
- ^ Dunn MR, Jimenez RM, Chaput JC (2017). “Analysis of aptamer discovery and technology”. Nature Reviews Chemistry 1 (10). doi:10.1038/s41570-017-0076 2022
年 6月 30日 閲覧 。. - ^ Wray GA (2002). “Dating branches on the tree of life using DNA”. Genome Biology 3 (1): REVIEWS0001. doi:10.1186/gb-2001-3-1-reviews0001. PMC 150454. PMID 11806830.
- ^ Panda D, Molla KA, Baig MJ, Swain A, Behera D, Dash M (May 2018). “DNA as a digital information storage device: hope or hype?”. 3 Biotech 8 (5): 239. doi:10.1007/s13205-018-1246-7. PMC 5935598. PMID 29744271.
- ^ Akram F, Haq IU, Ali H, Laghari AT (October 2018). “Trends to store digital data in DNA: an overview”. Molecular Biology Reports 45 (5): 1479–1490. doi:10.1007/s11033-018-4280-y. PMID 30073589.
- ^ Miescher F (1871). “Ueber die chemische Zusammensetzung der Eiterzellen [On the chemical composition of pus cells]” (ドイツ
語 ). Medicinisch-chemische Untersuchungen 4: 441–60. "[p. 456] Ich habe mich daher später mit meinen Versuchen an die ganzen Kerne gehalten, die Trennung der Körper, die ich einstweilen ohne weiteres Präjudiz als lösliches und unlösliches Nuclein bezeichnen will, einem günstigeren Material überlassend. (Therefore, in my experiments I subsequently limited myself to the whole nucleus, leaving to a more favorable material the separation of the substances, that for the present, without further prejudice, I will designate as soluble and insoluble nuclear material ("Nuclein"))" - ^ Dahm R (January 2008). “Discovering DNA: Friedrich Miescher and the early years of nucleic acid research”. Human Genetics 122 (6): 565–81. doi:10.1007/s00439-007-0433-0. PMID 17901982.
- ^ See:
- Kossel A (1879). “Ueber Nucleïn der Hefe [On nuclein in yeast]” (ドイツ
語 ). Zeitschrift für physiologische Chemie 3: 284–91. - Kossel A (1880). “Ueber Nucleïn der Hefe II [On nuclein in yeast, Part 2]” (ドイツ
語 ). Zeitschrift für physiologische Chemie 4: 290–95. - Kossel A (1881). “Ueber die Verbreitung des Hypoxanthins im Thier- und Pflanzenreich [On the distribution of hypoxanthins in the animal and plant kingdoms]” (ドイツ
語 ). Zeitschrift für physiologische Chemie 5: 267–71. - Kossel A (1881) (ドイツ
語 ). Untersuchungen über die Nucleine und ihre Spaltungsprodukte [Investigations into nuclein and its cleavage products]. Strassburg, Germany. pp. 19 - Kossel A (1882). “Ueber Xanthin und Hypoxanthin [On xanthin and hypoxanthin]”. Zeitschrift für physiologische Chemie 6: 422–31.
- Albrect Kossel (1883) "Zur Chemie des Zellkerns" Archived 17 November 2017 at the Wayback Machine. (On the chemistry of the cell nucleus), Zeitschrift für physiologische Chemie, 7: 7–22.
- Kossel A (1886). “Weitere Beiträge zur Chemie des Zellkerns [Further contributions to the chemistry of the cell nucleus]” (ドイツ
語 ). Zeitschrift für Physiologische Chemie 10: 248–64. "On p. 264, Kossel remarked presciently: Der Erforschung der quantitativen Verhältnisse der vier stickstoffreichen Basen, der Abhängigkeit ihrer Menge von den physiologischen Zuständen der Zelle, verspricht wichtige Aufschlüsse über die elementaren physiologisch-chemischen Vorgänge. (The study of the quantitative relations of the four nitrogenous bases—[and] of the dependence of their quantity on the physiological states of the cell—promises important insights into the fundamental physiological-chemical processes.)"
- Kossel A (1879). “Ueber Nucleïn der Hefe [On nuclein in yeast]” (ドイツ
- ^ Jones ME (September 1953). “Albrecht Kossel, a biographical sketch”. The Yale Journal of Biology and Medicine 26 (1): 80–97. PMC 2599350. PMID 13103145.
- ^ Levene PA, Jacobs WA (1909). “Über Inosinsäure” (ドイツ
語 ). Berichte der Deutschen Chemischen Gesellschaft 42: 1198–203. doi:10.1002/cber.190904201196. - ^ Levene PA, Jacobs WA (1909). “Über die Hefe-Nucleinsäure” (ドイツ
語 ). Berichte der Deutschen Chemischen Gesellschaft 42 (2): 2474–78. doi:10.1002/cber.190904202148. - ^ Levene P (1919). “The structure of yeast nucleic acid”. J Biol Chem 40 (2): 415–24. doi:10.1016/S0021-9258(18)87254-4.
- ^ Cohen JS, Portugal FH (1974). “The search for the chemical structure of DNA”. Connecticut Medicine 38 (10): 551–52, 554–57. PMID 4609088.
- ^ Koltsov proposed that a cell's genetic information was encoded in a long chain of amino acids. See:
- Koltsov HK (12 December 1927). Физико-химические основы морфологии [The physical-chemical basis of morphology] (Speech). 3rd All-Union Meeting of Zoologist, Anatomists, and Histologists (ロシア
語 ). Leningrad, U.S.S.R. - Reprinted in: Koltsov HK (1928). “Физико-химические основы морфологии [The physical-chemical basis of morphology]” (ロシア
語 ). Успехи экспериментальной биологии (Advances in Experimental Biology) series B 7 (1): ?. - Reprinted in German as: Koltzoff NK (1928). “Physikalisch-chemische Grundlagen der Morphologie [The physical-chemical basis of morphology]” (ドイツ
語 ). Biologisches Zentralblatt 48 (6): 345–69. - In 1934, Koltsov contended that the proteins that contain a cell's genetic information replicate. See: Koltzoff N (October 1934). “The structure of the chromosomes in the salivary glands of Drosophila”. Science 80 (2075): 312–13. Bibcode: 1934Sci....80..312K. doi:10.1126/science.80.2075.312. PMID 17769043. "From page 313: "I think that the size of the chromosomes in the salivary glands [of Drosophila] is determined through the multiplication of genonemes. By this term I designate the axial thread of the chromosome, in which the geneticists locate the linear combination of genes; … In the normal chromosome there is usually only one genoneme; before cell-division this genoneme has become divided into two strands.""
- Koltsov HK (12 December 1927). Физико-химические основы морфологии [The physical-chemical basis of morphology] (Speech). 3rd All-Union Meeting of Zoologist, Anatomists, and Histologists (ロシア
- ^ Soyfer VN (September 2001). “The consequences of political dictatorship for Russian science”. Nature Reviews Genetics 2 (9): 723–29. doi:10.1038/35088598. PMID 11533721.
- ^ Griffith F (January 1928). “The Significance of Pneumococcal Types”. The Journal of Hygiene 27 (2): 113–59. doi:10.1017/S0022172400031879. PMC 2167760. PMID 20474956.
- ^ Lorenz MG, Wackernagel W (September 1994). “Bacterial gene transfer by natural genetic transformation in the environment”. Microbiological Reviews 58 (3): 563–602. doi:10.1128/MMBR.58.3.563-602.1994. PMC 372978. PMID 7968924.
- ^ Brachet J (1933). “Recherches sur la synthese de l'acide thymonucleique pendant le developpement de l'oeuf d'Oursin” (イタリア
語 ). Archives de Biologie 44: 519–76. - ^ Burian R (1994). “Jean Brachet's Cytochemical Embryology: Connections with the Renovation of Biology in France?”. Les sciences biologiques et médicales en France 1920–1950. Cahiers pour I'histoire de la recherche. 2. Paris: CNRS Editions. pp. 207–20
- ^ See:
- Astbury WT, Bell FO (1938). “Some recent developments in the X-ray study of proteins and related structures”. Cold Spring Harbor Symposia on Quantitative Biology 6: 109–21. doi:10.1101/sqb.1938.006.01.013. オリジナルの14 July 2014
時点 におけるアーカイブ。. - Astbury WT (1947). “X-ray studies of nucleic acids”. Symposia of the Society for Experimental Biology (1): 66–76. PMID 20257017. オリジナルの5 July 2014
時点 におけるアーカイブ。.
- Astbury WT, Bell FO (1938). “Some recent developments in the X-ray study of proteins and related structures”. Cold Spring Harbor Symposia on Quantitative Biology 6: 109–21. doi:10.1101/sqb.1938.006.01.013. オリジナルの14 July 2014
- ^ Avery OT, Macleod CM, McCarty M (February 1944). “Studies on the Chemical Nature of the Substance Inducing Transformation of Pneumococcal Types: Induction of Transformation by a Desoxyribonucleic Acid Fraction Isolated from Pneumococcus Type III”. The Journal of Experimental Medicine 79 (2): 137–158. doi:10.1084/jem.79.2.137. PMC 2135445. PMID 19871359.
- ^ Chargaff E (June 1950). “Chemical specificity of nucleic acids and mechanism of their enzymatic degradation”. Experientia 6 (6): 201–209. doi:10.1007/BF02173653. PMID 15421335.
- ^ Kresge N, Simoni RD, Hill RL (June 2005). “Chargaff's Rules: the Work of Erwin Chargaff”. Journal of Biological Chemistry 280 (24): 172–174. doi:10.1016/S0021-9258(20)61522-8.
- ^ Hershey AD, Chase M (May 1952). “Independent functions of viral protein and nucleic acid in growth of bacteriophage”. The Journal of General Physiology 36 (1): 39–56. doi:10.1085/jgp.36.1.39. PMC 2147348. PMID 12981234.
- ^ “Pictures and Illustrations: Crystallographic photo of Sodium Thymonucleate, Type B. "Photo 51." May 1952”. scarc.library.oregonstate.edu. 2023
年 5月 18日 閲覧 。 - ^ Schwartz J (2008). In pursuit of the gene: from Darwin to DNA. Cambridge, Mass.: Harvard University Press. ISBN 978-0-674-02670-4
- ^ Pauling L, Corey RB (February 1953). “A Proposed Structure For The Nucleic Acids”. Proceedings of the National Academy of Sciences of the United States of America 39 (2): 84–97. Bibcode: 1953PNAS...39...84P. doi:10.1073/pnas.39.2.84. PMC 1063734. PMID 16578429.
- ^ Regis E (2009). What Is Life?: investigating the nature of life in the age of synthetic biology. Oxford: Oxford University Press. p. 52. ISBN 978-0-19-538341-6
- ^ “Double Helix of DNA: 50 Years”. Nature Archives. 2015
年 4月 5日 時点 のオリジナルよりアーカイブ。2007年 2月 15日 閲覧 。 - ^ “Original X-ray diffraction image”. Oregon State Library. 2009
年 1月 30日 時点 のオリジナルよりアーカイブ。2011年 2月 6日 閲覧 。 - ^ “The Nobel Prize in Physiology or Medicine 1962”. Nobelprize.org. 2006
年 12月24日 閲覧 。 - ^ Burakoff M (2023
年 4月 25日 ). “Rosalind Franklin's role in DNA discovery gets a new twist”. AP News 2023年 4月 25日 閲覧 。 - ^ Anthes E (2023
年 4月 25日 ). “Untangling Rosalind Franklin's Role in DNA Discovery, 70 Years On – Historians have long debated the role that Dr. Franklin played in identifying the double helix. A new opinion essay argues that she was an "equal contributor."”. The New York Times. オリジナルの2023年 4月 25日 時点 におけるアーカイブ。 2023年 4月 26日 閲覧 。 - ^ Cobb M, Comfort N (25 April 2023). “What Rosalind Franklin truly contributed to the discovery of DNA's structure – Franklin was no victim in how the DNA double helix was solved. An overlooked letter and an unpublished news article, both written in 1953, reveal that she was an equal player.”. Nature 616 (7958): 657–660. doi:10.1038/d41586-023-01313-5. PMID 37100935.
- ^ Maddox B (January 2003). “The double helix and the 'wronged heroine'”. Nature 421 (6921): 407–08. Bibcode: 2003Natur.421..407M. doi:10.1038/nature01399. PMID 12540909. オリジナルの17 October 2016
時点 におけるアーカイブ。. - ^ Crick FH (1955). A Note for the RNA Tie Club (PDF) (Speech). Cambridge, England. 2008
年 10月 1日 時点 のオリジナル (PDF)よりアーカイブ。 - ^ Meselson M, Stahl FW (July 1958). “The Replication of DNA in Escherichia Coli”. Proceedings of the National Academy of Sciences of the United States of America 44 (7): 671–82. Bibcode: 1958PNAS...44..671M. doi:10.1073/pnas.44.7.671. PMC 528642. PMID 16590258.
- ^ “The Nobel Prize in Physiology or Medicine 1968”. Nobelprize.org. 2006
年 12月24日 閲覧 。 - ^ Pray L (2008). “Discovery of DNA structure and function: Watson and Crick.”. Nature Education 1 (1): 100.
- ^ Panneerchelvam S, Norazmi MN (2003). “Forensic DNA Profiling and Database”. The Malaysian Journal of Medical Sciences 10 (2): 20–26. PMC 3561883. PMID 23386793.
推薦 文献
編集
田村 隆明 『基礎 細胞 生物 学 』(第 1版 第 1刷 版 )、東京 化学 同人 、2010年 。ISBN 978-4-8079-0724-3。武村 政春 『DNAを操 る分子 たち』(初版 第 1刷 版 )、技術評論社 、2012年 。ISBN 978-4-7741-4998-1。- Berry A, Watson J (2003
年 ). DNA: the secret of life. New York: Alfred A. Knopf. ISBN 0-375-41546-7。 - Calladine CR, Drew HR, Luisi BF, Travers AA (2003
年 ). Understanding DNA: the molecule & how it works. Amsterdam: Elsevier Academic Press. ISBN 0-12-155089-3。 - Carina D, Clayton J (2003
年 ). 50 years of DNA. Basingstoke: Palgrave Macmillan. ISBN 1-4039-1479-6。 - Judson HF [in
英語 ] (1979年 ). The Eighth Day of Creation: Makers of the Revolution in Biology (2nd ed.). Cold Spring Harbor Laboratory Press. ISBN 0-671-22540-5。 - Olby RC [in
英語 ] (1994年 ). The path to the double helix: the discovery of DNA. New York: Dover Publications. ISBN 0-486-68117-3。 First published in October 1974 by MacMillan, with foreword by Francis Crick; the definitive DNA textbook, revised in 1994 with a nine-page postscript. - Olby R (2003
年 1月 ). "Quiet debut for the double helix". Nature. 421 (6921): 402–05. Bibcode:2003Natur.421..402O. doi:10.1038/nature01397. PMID 12540907。 - Olby RC (2009
年 ). Francis Crick: A Biography. Plainview, N.Y: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-798-3。 - Micklas D (2003
年 ). DNA Science: A First Course. Cold Spring Harbor Press. ISBN 978-0-87969-636-8。 - Ridley M [in
英語 ] (2006年 ). Francis Crick: discoverer of the genetic code. Ashland, OH: Eminent Lives, Atlas Books. ISBN 0-06-082333-X。 - Rosenfeld I (2010
年 ). DNA: A Graphic Guide to the Molecule that Shook the World. Columbia University Press. ISBN 978-0-231-14271-7。 - Schultz M, Cannon Z (2009
年 ). The Stuff of Life: A Graphic Guide to Genetics and DNA. Hill and Wang. ISBN 978-0-8090-8947-5。 - Stent GS, Watson J [in
英語 ] (1980年 ). The Double Helix: A Personal Account of the Discovery of the Structure of DNA. New York: Norton. ISBN 0-393-95075-1。 - Watson J (2004
年 ). DNA: The Secret of Life. Random House. ISBN 978-0-09-945184-6。 - Wilkins M [in
英語 ] (2003年 ). The third man of the double helix the autobiography of Maurice Wilkins. Cambridge, England: University Press. ISBN 0-19-860665-6。
外部 リンク
編集
- DNA - Curlie(
英語 ) - DNA binding site prediction on protein
- DNA the Double Helix Game From the official Nobel Prize web site
- DNA under electron microscope
- Dolan DNA Learning Center
- Double Helix: 50 years of DNA, Nature
- Proteopedia DNA
- Proteopedia Forms_of_DNA
- ENCODE threads explorer ENCODE home page at Nature
- Double Helix 1953–2003 National Centre for Biotechnology Education
- Genetic Education Modules for Teachers – DNA from the Beginning Study Guide
蛋白質 構造 データバンク今月 の分子 23:DNA(DNA)- "Clue to chemistry of heredity found". The New York Times, June 1953. First American newspaper coverage of the discovery of the DNA structure
- DNA from the Beginning Another DNA Learning Center site on DNA, genes, and heredity from Mendel to the human genome project.
- The Register of Francis Crick Personal Papers 1938 – 2007 at Mandeville Special Collections Library, University of California, San Diego
- Seven-page, handwritten letter that Crick sent to his 12-year-old son Michael in 1953 describing the structure of DNA. See Crick's medal goes under the hammer, Nature, 5 April 2013.
- 『デオキシリボ
核酸 』 - コトバンク