ファージ

ファージ(
概要
[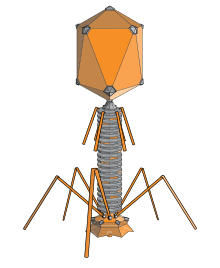
バクテリオファージは、DNAまたはRNAのゲノムをカプセル
バクテリオファージは、
バクテリオファージは、20
20
構造
[
バクテリオファージには
分類
[
バクテリオファージは
| Belfryvirales | Turriviridae | Enveloped, isometric | Linear dsDNA | |
| Caudovirales | Ackermannviridae | Nonenveloped, contractile tail | Linear dsDNA | |
| Myoviridae | Nonenveloped, contractile tail | Linear dsDNA | T4, Mu, P1, P2 | |
| Siphoviridae | Nonenveloped, noncontractile tail (long) | Linear dsDNA | ||
| Podoviridae | Nonenveloped, noncontractile tail (short) | Linear dsDNA | T7, T3, | |
| Halopanivirales | Sphaerolipoviridae | Enveloped, isometric | Linear dsDNA | |
| Haloruvirales | Pleolipoviridae | Enveloped, pleomorphic | Circular ssDNA, circular dsDNA, or linear dsDNA | |
| Kalamavirales | Tectiviridae | Nonenveloped, isometric | Linear dsDNA | |
| Levivirales | Leviviridae | Nonenveloped, isometric | Linear ssRNA | MS2, Q |
| Ligamenvirales | Lipothrixviridae | Enveloped, rod-shaped | Linear dsDNA | Acidianus filamentous virus 1 |
| Rudiviridae | Nonenveloped, rod-shaped | Linear dsDNA | Sulfolobus islandicus rod-shaped virus 1 | |
| Mindivirales | Cystoviridae | Enveloped, spherical | Segmented dsRNA | |
| Petitvirales | Microviridae | Nonenveloped, isometric | Circular ssDNA | |
| Tubulavirales | Inoviridae | Nonenveloped, filamentous | Circular ssDNA | M13 |
| Vinavirales | Corticoviridae | Nonenveloped, isometric | Circular dsDNA | PM2 |
| Unassigned | Ampullaviridae | Enveloped, bottle-shaped | Linear dsDNA | |
| Bicaudaviridae | Nonenveloped, lemon-shaped | Circular dsDNA | ||
| Clavaviridae | Nonenveloped, rod-shaped | Circular dsDNA | ||
| Finnlakeviridae | dsDNA | FLiP[10] | ||
| Fuselloviridae | Nonenveloped, lemon-shaped | Circular dsDNA | ||
| Globuloviridae | Enveloped, isometric | Linear dsDNA | ||
| Guttaviridae | Nonenveloped, ovoid | Circular dsDNA | ||
| Plasmaviridae | Enveloped, pleomorphic | Circular dsDNA | ||
| Portogloboviridae | Enveloped, isometric | Circular dsDNA | ||
| Spiraviridae | Nonnveloped, rod-shaped | Circular ssDNA | ||
| Tristromaviridae | Enveloped, rod-shaped | Linear dsDNA |
ピコビルナ
もう
歴史
[
1896
トウォートの
応用
[ファージセラピー
[ファージは、
抗生 物質 が発見 されて広 く販売 された。それらは、製造 、保管 、および処方 が容易 であった。- ファージの
医学 的 試験 が行 われたが、基本 的 な理解 不足 から、これらの試験 の妥当 性 に疑問 が生 じた[19]。 - ソビエト
連邦 での研究 の出版 は主 にロシア語 かジョージア語 で行 われ、長 い間 、国際 的 には追随 されていなかった。
デレーユは「バクテリオファージは、
その他
[食品 業界 - 2006
年 以降 、アメリカ食品 医薬品 局 (FDA)とアメリカ農務 省 (USDA)は、いくつかのバクテリオファージ製品 を承認 している。LMP-102(Intralytix)は、加工 (RTE)鶏肉 および肉 製品 の治療 に承認 された。同年 、FDAは、一般 に安全 と認 められる(GRAS)ステータスを与 えるために、チーズにバクテリオファージを使用 してリステリア・モノサイトゲネス菌 を殺 すLISTEX(マイクレオスによって開発 ・製造 された)を承認 した[28]。2007年 7月 には、すべての食品 での同 じバクテリオファージの使用 が承認 された[29]。2011年 、USDAは、LISTEXをクリーンラベル加工 助 剤 として、USDAに含 まれることを確認 した[30]。食品 安全 の分野 では、さまざまな食品 中 の他 の食物 由来 病原 体 を制御 するため、実行 可能 な選択肢 として、溶菌 性 ファージがあるかどうかの研究 が続 けられている。 酪農 環境 中 に存在 するバクテリオファージは、チーズスターター培養 物 の発酵 失敗 を引 き起 こす可能 性 がある。これを回避 するために、混合 株 スターター培養 や培養 物 ローテーション計画 を使用 することができる[31]。診断 法 - 2011
年 、FDAは、体外 (in vitro)診断 用 の最初 のバクテリオファージに基 づく製品 を承認 した[32]。KeyPath MRSA/MSSA血液 培養 検査 では、バクテリオファージのカクテルを使用 して、陽性 の血液 培養 物 中 の黄色 ブドウ球菌 (Staphylococcus aureus)を検出 し、メチシリン耐性 または感受性 を判定 する。この検査 では、標準 的 な微生物 同定 および感受性 検査 法 では2~3日 かかるのに対 し、約 5時 間 で結果 が得 られる。これは、FDAによって承認 された最初 の迅速 化 抗生 物質 感受性 検査 である[33]。 生物 兵器 や毒素 への対抗 欧米 の政府 機関 は数 年 前 から、炭疽 菌 やボツリヌス菌 などの生物 兵器 や毒素 に対抗 するためのファージの利用 について、ジョージアや旧 ソビエト連邦 に協力 を求 めてきた[34]。米国 の研究 グループ間 で開発 が続 けられている。その他 の用途 としては、植物 や野菜 の生産 物 を腐敗 や細菌 感染 の蔓延 から保護 するための園芸 での散布 などがある。バクテリオファージの他 の用途 は、病院 などの環境 表面 の殺生 物 剤 として、また、臨床 現場 で使用 する前 のカテーテルや医療 機器 の予防 処置 としてである。ファージを乾燥 した表面 、例 えばユニフォーム、カーテン、または手術 用 の縫合 糸 などに適用 する技術 が現在 存在 している。Clinical Otolaryngology誌 [22]に報告 された臨床 試験 では、ペット犬 の中耳炎 の獣医 学 的 治療 に成功 している。- SEPTIC
菌 検出 ・同定 法 - この
手法 は、ファージ感染 時 のイオン放出 とその動態 を利用 した高 い特異 性 と検出 速度 を実現 している[35]。 - ファージディスプレイ
- これは、
表面 タンパク質 に連結 した可変 ペプチドを有 するファージのライブラリーに関 わるファージの別 の使用 法 である。各 ファージゲノムは、その表面 に発現 しているタンパク質 の変異 体 をコードし(その名 の由来 )、変異 ペプチドとそのエンコード遺伝子 との間 のリンクを提供 する。ライブラリーからの変異 ファージは、固定 化 された分子 (例 えば、ボツリヌス毒素 )を中和 するために、その結合 親和 性 を通 じて選抜 される。結合 し、選抜 されたファージは、感受性 のある細菌 株 を再 感染 させて増殖 することができ、それにより、さらなる研究 のためにファージにコードされたペプチドを回収 することができる[36]。 抗菌 薬 の発見 - ファージタンパク
質 はしばしば抗菌 活性 を持 ち、ペプチドミメティック、すなわちペプチドを模倣 する薬剤 のリードとなる可能 性 がある[37]。ファージリガンド技術 は、細菌 や細菌 成分 (エンドトキシンなど)の結合 や細菌 の溶解 など、さまざまな用途 にファージタンパク質 を利用 する[38]。 基礎 研究 - バクテリオファージは
遺伝子 数 が他 の生物 に比 べて少 なく、また増殖 が容易 なことから、進化 と生態 学 の原理 を研究 するための重要 なモデル生物 である[39]。 遺伝子 工学 - テンペレートファージを
利用 して宿主 の細菌 に任意 の遺伝子 を導入 する技術 も開発 された。この技術 は形質 導入 と呼 ばれ、ラムダファージによる大腸菌 への形質 導入 が、分子生物学 分野 で繁用 されている。 - ファージ
型 別 - ファージは
種類 によって宿主 とする細菌 が異 なり、しかもその選択 性 が高 い。このため同 じ種 に属 する細菌 であっても、株 によって特定 のファージに感染 するものとしないものがある。この現象 を利用 して同種 の細菌 をさらに細 かく判別 することが可能 であり、この方法 をファージ型 別 と呼 ぶ。ファージ型 別 による分類 は黄色 ブドウ球菌 やサルモネラに用 いられており、これらの菌 の中 でも特 に病原 性 の高 いものであるかどうかを識別 することが可能 である。
複製
[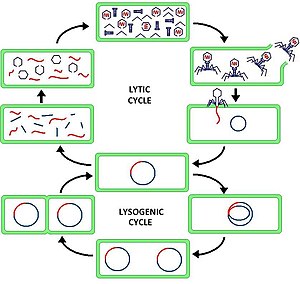

バクテリオファージは「
T4ファージのような
テンペレートファージの
付着 と浸透
[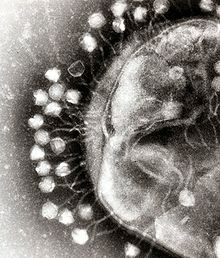
ミオウイルス・バクテリオファージは、
タンパク質 と核酸 の合成
[ウイルス組 み立 て
[T4ファージの
ウイルスの放出
[ファージは、
コミュニケーション
[2017
ゲノム構造
[バクテリオファージのゲノムは、
システム生物 学
[ファージはしばしば
ファージとその
環境 中
[メタゲノミクスにより、
また、バクテリオファージは、
バクテリオファージは、
モデルバクテリオファージ
[
参照 項目
[- ヴィロファージ -
他 のウイルスに寄生 するウイルス 細菌 食 性 生物 - CRISPR
- DNAウイルス
- RNAウイルス
- ファージディスプレイ
形質 導入 - ハーシーとチェイスの
実験 - ファージにおける遺伝 物質 がDNAであることを確定 した一連 の実験 - ファージ
生態 学 - ファージ
関連 の研究 論文 - ファージおよびファージ関連 の研究 論文 の包括 的 なリスト、1921年 から現在 まで - ポリファージ
- Viriome
- crAssphage
- ファージミド
脚注
[- ^ a b McGrath S and van Sinderen D (editors). (2007). Bacteriophage: Genetics and Molecular Biology (1st ed.). Caister Academic Press. ISBN 978-1-904455-14-1
- ^ “Novel Phage Therapy Saves Patient with Multidrug-Resistant Bacterial Infection”. UC Health – UC San Diego 2018
年 5月 13日 閲覧 。 - ^ Suttle, Curtis A. (September 2005). “Viruses in the sea” (
英語 ). Nature 437 (7057): 356–361. doi:10.1038/nature04160. ISSN 0028-0836. - ^ a b Wommack, K. E.; Colwell, R. R. (2000). “Virioplankton: Viruses in Aquatic Ecosystems”. Microbiology and Molecular Biology Reviews 64 (1): 69–114. doi:10.1128/MMBR.64.1.69-114.2000. PMC 98987. PMID 10704475.
- ^ a b c Prescott, L. (1993). Microbiology, Wm. C. Brown Publishers, ISBN 0-697-01372-3
- ^ a b BBC Horizon (1997): The Virus that Cures – Documentary about the history of phage medicine in Russia and the West
- ^ Borrell, Brendan (August 2012). “Science talk: Phage factor”. Scientific American: 80–83.
- ^ Keen, E. C. (2012). “Phage Therapy: Concept to Cure”. Frontiers in Microbiology 3: 238. doi:10.3389/fmicb.2012.00238. PMC 3400130. PMID 22833738.
- ^ Sweere, Johanna M.; Van Belleghem, Jonas D.; Ishak, Heather; Bach, Michelle S.; Popescu, Medeea; Sunkari, Vivekananda; Kaber, Gernot; Manasherob, Robert et al. (2019). “Bacteriophage trigger antiviral immunity and prevent clearance of bacterial infection” (
英語 ). Science 363 (6434): eaat9691. doi:10.1126/science.aat9691. ISSN 0036-8075. - ^ Elina Laanto, Sari Mäntynen, Luigi De Colibus, Jenni Marjakangas, Ashley Gillum, David I. Stuart, Janne J. Ravantti, Juha Huiskonen, Lotta-Riina Sundberg: Virus found in a boreal lake links ssDNA and dsDNA viruses. In: Proceedings of the National Academy of Sciences 114(31), July 2017, doi:10.1073/pnas.1703834114
- ^ Krishnamurthy SR, Wang D (2018). “Extensive conservation of prokaryotic ribosomal binding sites in known and novel picobirnaviruses”. Virology 516: 108–114. doi:10.1016/j.virol.2018.01.006. PMID 29346073.
- ^ Kathryn M. Kauffman, Fatima A. Hussain, Joy Yang, Philip Arevalo, Julia M. Brown, William K. Chang, David VanInsberghe, Joseph Elsherbini, Radhey S. Sharma, Michael B. Cutler, Libusha Kelly, Martin F. Polz: A major lineage of non-tailed dsDNA viruses as unrecognized killers of marine bacteria. In: Nature Vol. 554, pp. 118–122. January 24th, 2018. doi:10.1038/nature25474
- ^ Hankin, E H. (1896). “L'action bactericide des eaux de la Jumna et du Gange sur le vibrion du cholera” (
フランス語 ). Annales de l'Institut Pasteur 10: 511–23. - ^ Twort, F. W. (1915). “An Investigation on the Nature of Ultra-Microscopic Viruses”. The Lancet 186 (4814): 1241–43. doi:10.1016/S0140-6736(01)20383-3.
- ^ d'Hérelles, Félix (1917). “Sur un microbe invisible antagoniste des bacilles dysentériques”. Comptes Rendus de l'Académie des Sciences de Paris 165: 373–5. オリジナルの11 May 2011
時点 におけるアーカイブ。 2010年 9月 5日 閲覧 。. - ^ d'Hérelles, Félix (1949). “The bacteriophage”. Science News 14: 44–59 2010
年 9月 5日 閲覧 。. - ^ Keen, EC (2012). “Felix d'Herelle and Our Microbial Future”. Future Microbiology 7 (12): 1337–39. doi:10.2217/fmb.12.115. PMID 23231482.
- ^ “The Nobel Prize in Physiology or Medicine 1969”. Nobel Foundation. 2007
年 7月 28日 閲覧 。 - ^ Kutter, Elizabeth; De Vos, Daniel; Gvasalia, Guram; Alavidze, Zemphira; Gogokhia, Lasha; Kuhl, Sarah; Abedon, Stephen (1 January 2010). “Phage Therapy in Clinical Practice: Treatment of Human Infections”. Current Pharmaceutical Biotechnology 11 (1): 69–86. doi:10.2174/138920110790725401. PMID 20214609.
- ^ Сергей Головин Бактериофаги: убийцы в роли спасителей // Наука и жизнь. – 2017. – № 6. – С. 26–33
- ^ a b Rhoads, DD; Wolcott, RD; Kuskowski, MA; Wolcott, BM; Ward, LS; Sulakvelidze, A (June 2009). “Bacteriophage therapy of venous leg ulcers in humans: results of a phase I safety trial”. Journal of Wound Care 18 (6): 237–8, 240–3. doi:10.12968/jowc.2009.18.6.42801. PMID 19661847.
- ^ a b c Wright, A.; Hawkins, C.H.; Änggård, E.E.; Harper, D.R. (August 2009). “A controlled clinical trial of a therapeutic bacteriophage preparation in chronic otitis due to antibiotic-resistant Pseudomonas aeruginosa; a preliminary report of efficacy”. Clinical Otolaryngology 34 (4): 349–357. doi:10.1111/j.1749-4486.2009.01973.x. PMID 19673983.
- ^ Tawil, Nancy (April 2012). “Surface plasmon resonance detection of E. coli and methicillin-resistant S. aureus using bacteriophages”. Biosensors and Bioelectronics 37 (1): 24–29. doi:10.1016/j.bios.2012.04.048. PMID 22609555.
- ^ Cha, Kyoungeun; Oh, Hynu K.; Jang, Jae Y.; Jo, Yunyeol; Kim, Won K.; Ha, Geon U.; Ko, Kwan S.; Myung, Heejoon (10 April 2018). “Characterization of Two Novel Bacteriophages Infecting Multidrug-Resistant (MDR) Acinetobacter baumannii and Evaluation of Their Therapeutic Efficacy in Vivo”. Frontiers in Microbiology 9: 696. doi:10.3389/fmicb.2018.00696. ISSN 1664-302X. PMC 5932359. PMID 29755420.
- ^ Schooley, Robert T.; Biswas, Biswajit; Gill, Jason J.; Hernandez-Morales, Adriana; Lancaster, Jacob; Lessor, Lauren; Barr, Jeremy J.; Reed, Sharon L. et al. (October 2017). “Development and Use of Personalized Bacteriophage-Based Therapeutic Cocktails To Treat a Patient with a Disseminated Resistant Acinetobacter baumannii Infection”. Antimicrobial Agents and Chemotherapy 61 (10). doi:10.1128/AAC.00954-17. ISSN 0066-4804. PMC 5610518. PMID 28807909.
- ^ Kuchment, Anna (2012), The Forgotten Cure: The past and future of phage therapy, Springer, p. 11, ISBN 978-1-4614-0250-3
- ^ Deresinski, Stan (15 April 2009). “Bacteriophage Therapy: Exploiting Smaller Fleas”. Clinical Infectious Diseases 48 (8): 1096–1101. doi:10.1086/597405. PMID 19275495.
- ^ U.S. FDA/CFSAN: Agency Response Letter, GRAS Notice No. 000198
- ^ (U.S. FDA/CFSAN: Agency Response Letter, GRAS Notice No. 000218)
- ^ FSIS Directive 7120 Archived 18 October 2011 at the Wayback Machine.
- ^ Atamer, Zeynep; Samtlebe, Meike; Neve, Horst; J. Heller, Knut; Hinrichs, Joerg (16 July 2013). “Review: elimination of bacteriophages in whey and whey products”. Frontiers in Microbiology 4: 191. doi:10.3389/fmicb.2013.00191. PMC 3712493. PMID 23882262.
- ^ FDA 510(k) Premarket Notification
- ^ FDA clears first test to quickly diagnose and distinguish MRSA and MSSA. FDA (6 May 2011)
- ^ Vaisman, Daria (25 May 2007) Studying anthrax in a Soviet-era lab – with Western funding. The New York Times
- ^ Dobozi-King, M.; Seo, S.; Kim, J.U.; Young, R.; Cheng, M.; Kish, L.B. (2005). “Rapid detection and identification of bacteria: SEnsing of Phage-Triggered Ion Cascade (SEPTIC)”. Journal of Biological Physics and Chemistry 5: 3–7. doi:10.4024/1050501.jbpc.05.01.
- ^ Smith GP, Petrenko VA (April 1997). “Phage Display”. Chem. Rev. 97 (2): 391–410. doi:10.1021/cr960065d. PMID 11848876.
- ^ Liu, Jing; Dehbi, Mohammed; Moeck, Greg; Arhin, Francis; Bauda, Pascale; Bergeron, Dominique; Callejo, Mario; Ferretti, Vincent et al. (February 2004). “Antimicrobial drug discovery through bacteriophage genomics”. Nature Biotechnology 22 (2): 185–191. doi:10.1038/nbt932. PMID 14716317.
- ^ Technological background Phage-ligand technology
- ^ Keen, E. C. (2014). “Tradeoffs in bacteriophage life histories”. Bacteriophage 4 (1): e28365. doi:10.4161/bact.28365. PMC 3942329. PMID 24616839.
- ^ Mason, Kenneth A., Jonathan B. Losos, Susan R. Singer, Peter H Raven, and George B. Johnson. (2011). Biology, p. 533. McGraw-Hill, New York. ISBN 978-0-07-893649-4.
- ^ Mokrousov I (2009). “Corynebacterium diphtheriae: genome diversity, population structure and genotyping perspectives”. Infection, Genetics and Evolution 9 (1): 1–15. doi:10.1016/j.meegid.2008.09.011. PMID 19007916.
- ^ Charles RC, Ryan ET (October 2011). “Cholera in the 21st century”. Current Opinion in Infectious Diseases 24 (5): 472–7. doi:10.1097/QCO.0b013e32834a88af. PMID 21799407.
- ^ Keen, E. C. (December 2012). “Paradigms of pathogenesis: Targeting the mobile genetic elements of disease”. Frontiers in Cellular and Infection Microbiology 2: 161. doi:10.3389/fcimb.2012.00161. PMC 3522046. PMID 23248780.
- ^ Drulis-Kawa, Zuzanna; Majkowska-Skrobek, Grazyna; MacIejewska, Barbara (2015). “Bacteriophages and Phage-Derived Proteins – Application Approaches”. Current Medicinal Chemistry 22 (14): 1757–1773. doi:10.2174/0929867322666150209152851. PMC 4468916. PMID 25666799.
- ^ Gabashvili, I.; Khan, S.; Hayes, S.; Serwer, P. (1997). “Polymorphism of bacteriophage T7”. Journal of Molecular Biology 273 (3): 658–67. doi:10.1006/jmbi.1997.1353. PMID 9356254.
- ^ Maghsoodi, A.; Chatterjee, A.; Andricioaei, I.; Perkins, N.C. (2019-11-25). “How the phage T4 injection machinery works including energetics, forces, and dynamic pathway”. Proceedings of the National Academy of Sciences 116 (50): 25097–25105. doi:10.1073/pnas.1909298116. ISSN 0027-8424. PMC 6911207. PMID 31767752.
- ^ Fiers, W.; Contreras, R.; Duerinck, F.; Haegeman, G.; Iserentant, D.; Merregaert, J.; Min Jou, W.; Molemans, F. et al. (1976). “Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene”. Nature 260 (5551): 500–507. Bibcode: 1976Natur.260..500F. doi:10.1038/260500a0. PMID 1264203.
- ^ Mizuno, CM; Guyomar, C; Roux, S; Lavigne, R; Rodriguez-Valera, F; Sullivan, MB; Gillet, R; Forterre, P et al. (2019). “Numerous cultivated and uncultivated viruses encode ribosomal proteins.”. Nature Communications 10 (1): 752. Bibcode: 2019NatCo..10..752M. doi:10.1038/s41467-019-08672-6. PMC 6375957. PMID 30765709.
- ^ Snustad DP. Dominance interactions in Escherichia coli cells mixedly infected with bacteriophage T4D wild-type and amber mutants and their possible implications as to type of gene-product function: catalytic vs. stoichiometric. Virology. 1968 Aug;35(4):550-63. doi: 10.1016/0042-6822(68)90285-7. PMID 4878023.
- ^ Floor E. Interaction of morphogenetic genes of bacteriophage T4. J Mol Biol. 1970;47(3):293-306. doi:10.1016/0022-2836(70)90303-7
- ^ Callaway, Ewen (2017). “Do you speak virus? Phages caught sending chemical messages”. Nature. doi:10.1038/nature.2017.21313.
- ^ Erez, Zohar; Steinberger-Levy, Ida; Shamir, Maya; Doron, Shany; Stokar-Avihail, Avigail; Peleg, Yoav; Melamed, Sarah; Leavitt, Azita et al. (26 January 2017). “Communication between viruses guides lysis–lysogeny decisions”. Nature 541 (7638): 488–493. Bibcode: 2017Natur.541..488E. doi:10.1038/nature21049. ISSN 0028-0836. PMC 5378303. PMID 28099413.
- ^ Black, LW; Thomas, JA (2012). Condensed genome structure. Advances in Experimental Medicine and Biology. 726. pp. 469–87. doi:10.1007/978-1-4614-0980-9_21. ISBN 978-1-4614-0979-3. PMC 3559133. PMID 22297527
- ^ Al-Shayeb, Basem; Sachdeva, Rohan; Chen, Lin-Xing; Ward, Fred; Munk, Patrick; Devoto, Audra; Castelle, Cindy J.; Olm, Matthew R. et al. (February 2020). “Clades of huge phages from across Earth's ecosystems” (
英語 ). Nature 578 (7795): 425–431. doi:10.1038/s41586-020-2007-4. ISSN 1476-4687. PMC 7162821. PMID 32051592. - ^ Morris P, Marinelli LJ, Jacobs-Sera D, Hendrix RW, Hatfull GF (March 2008). “Genomic characterization of mycobacteriophage Giles: evidence for phage acquisition of host DNA by illegitimate recombination”. Journal of Bacteriology 190 (6): 2172–82. doi:10.1128/JB.01657-07. PMC 2258872. PMID 18178732.
- ^ Krupovic M, Prangishvili D, Hendrix RW, Bamford DH (December 2011). “Genomics of bacterial and archaeal viruses: dynamics within the prokaryotic virosphere”. Microbiology and Molecular Biology Reviews 75 (4): 610–35. doi:10.1128/MMBR.00011-11. PMC 3232739. PMID 22126996.
- ^ Zhao X, Chen C, Shen W, Huang G, Le S, Lu S, Li M, Zhao Y, Wang J, Rao X, Li G, Shen M, Guo K, Yang Y, Tan Y, Hu F (2016). “Global Transcriptomic Analysis of Interactions between Pseudomonas aeruginosa and Bacteriophage PaP3”. Sci Rep 6: 19237. Bibcode: 2016NatSR...619237Z. doi:10.1038/srep19237. PMC 4707531. PMID 26750429.
- ^ Blasche S, Wuchty S, Rajagopala SV, Uetz P (2013). “The protein interaction network of bacteriophage lambda with its host, Escherichia coli”. J. Virol. 87 (23): 12745–55. doi:10.1128/JVI.02495-13. PMC 3838138. PMID 24049175.
- ^ Breitbart M, Salamon P, Andresen B, Mahaffy JM, Segall AM, Mead D, Azam F, Rohwer F (October 2002). “Genomic analysis of uncultured marine viral communities”. Proc. Natl. Acad. Sci. U.S.A. 99 (22): 14250–5. Bibcode: 2002PNAS...9914250B. doi:10.1073/pnas.202488399. PMC 137870. PMID 12384570.
- ^ Martin, C. (1988). “The Application of Bacteriophage Tracer Techniques in South West Water”. Water and Environment Journal 2 (6): 638–642. doi:10.1111/j.1747-6593.1988.tb01352.x.
- ^ Bergh, O (1989). “High abundance of viruses found in aquatic environments”. Nature 340 (6233): 467–468. Bibcode: 1989Natur.340..467B. doi:10.1038/340467a0. PMID 2755508.
- ^ Keen, Eric C.; Bliskovsky, Valery V.; Malagon, Francisco; Baker, James D.; Prince, Jeffrey S.; Klaus, James S.; Adhya, Sankar L.; Groisman, Eduardo A. (2017). “Novel "Superspreader" Bacteriophages Promote Horizontal Gene Transfer by Transformation”. mBio 8 (1): e02115–16. doi:10.1128/mBio.02115-16. PMC 5241400. PMID 28096488.
- ^ Lekunberri, Itziar; Subirats, Jessica; Borrego, Carles M.; Balcazar, Jose L. (2017). “Exploring the contribution of bacteriophages to antibiotic resistance”. Environmental Pollution 220 (Pt B): 981–984. doi:10.1016/j.envpol.2016.11.059. hdl:10256/14115. PMID 27890586.
- ^ Strauss, James H.; Sinsheimer, Robert L. (July 1963). “Purification and properties of bacteriophage MS2 and of its ribonucleic acid”. Journal of Molecular Biology 7 (1): 43–54. doi:10.1016/S0022-2836(63)80017-0. PMID 13978804.
- ^ Miller, ES; Kutter, E; Mosig, G; Arisaka, F; Kunisawa, T; Rüger, W (March 2003). “Bacteriophage T4 genome”. Microbiology and Molecular Biology Reviews 67 (1): 86–156, table of contents. doi:10.1128/MMBR.67.1.86-156.2003. PMC 150520. PMID 12626685.
- ^ Ackermann, H.-W.; Krisch, H. M. (6 April 2014). “A catalogue of T4-type bacteriophages”. Archives of Virology 142 (12): 2329–2345. doi:10.1007/s007050050246. PMID 9672598.
参考 書目
[- Hauser, AR (2016). “Beyond Antibiotics: New Therapeutic Approaches for Bacterial Infections”. Clinical Infectious Diseases 63 (1): 89–95. doi:10.1093/cid/ciw200. PMC 4901866. PMID 27025826.
- Strathdee, Steffanie; Patterson, Tom (2019). The Perfect Predator. Hachette Books. ISBN 978-0316418089
- Takemura, Masaharu;
武村 政春 . (2019). ヒトがいまあるのはウイルスのおかげ! ―役 に立 つウイルス・かわいいウイルス・創造 主 のウイルス. Tōkyō: さくら舎 . ISBN 978-4-86581-179-7. OCLC 1082187420
外部 リンク
[- T4ファージの
微細 構造 と感染 のメカニズム at the Wayback Machine (archived 2017-12-22) - Häusler, T. (2006) "Viruses vs. Superbugs", Macmillan
- Animation of bacteriophage targeting E. coli bacteria
- Phage.org general information on bacteriophages
- bacteriophages illustrations and genomics
- Bacteriophages get a foothold on their prey
- NPR Science Friday podcast, "Using 'Phage' Viruses to Help Fight Infection", April 2008
- Animation of a scientifically correct T4 bacteriophage targeting E. coli bacteria
- Animation by Hybrid Animation Medical for a T4 Bacteriophage targeting E. coli bacteria
- Bacteriophages: What are they. Presentation by Professor Graham Hatfull, University of Pittsburgh - YouTube
- 『バクテリオファージ』 - コトバンク

