細菌
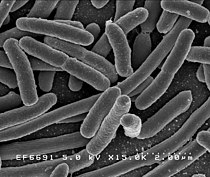 | |||
| |||
| Bacteria Woese et al. 2024 | |||
| シノニム | |||
| |||
|
しかし
また、
呼称
なお、「
このほかの
起源 と初期 の進化
生育 環境
また
バイオマスの
形状 ・大 きさ
細胞 構造
エネルギー
細胞 膜 外 構造
グラム
グリコカリックスは
このような
細胞 膜 内 構造
内 生 胞子
バチルス、クロストリジウム、Sporohalobacter、Anaerobacter、Heliobacteriumなどのグラム
代謝 と物質 循環
バクテリアは
- エネルギー
源 炭素 源
これらの、エネルギー
| エネルギー |
|||
|---|---|---|---|
| シアノバクテリア、 | |||
| リソトロフ | サーモデスルフォバクテリア、ヒドロゲノフィラス、ニトロスピラ | ||
| Bacillus、Clostridium、Enterobacteriaceaeなど |
硫黄 循環
成長 と増殖
ゲノムと遺伝子
ほとんどの
しかし、
- 1つ
目 は形質 転換 と呼 ばれるプロセスで、細胞 外 の外因 性 DNAを取 り込 む仕組 みである[131]。多 くの細菌 はこの取 り込 み機能 を持 っているが、DNAを取 り込 むためには化学 的 な誘導 が必要 となる細菌 もいる[132]。自然 界 でのDNA取 り込 み能力 の発達 は、環境 からのストレスの多 さと関連 しており、細胞 のDNA損傷 の修復 を促進 するための適応 であると考 えられている[133]。 - 2
番 めは形質 導入 と呼 ばれるプロセスであり、これはバクテリオファージの感染 によって外来 DNAの遺伝 物質 が細胞 内 の染色 体 に導入 されるものである。非常 に多様 なバクテリオファージが存在 することが知 られており、それらには宿主 細菌 に感染 して溶菌 してしまうものもあれば、プロファージとして細菌 の染色 体 に挿入 されるものもある[134]。バクテリアは、外来 DNAを分解 する制限 修飾 システム[135]や、バクテリアが過去 に接触 したファージのゲノムの断片 を保持 するためにCRISPR-Casシステムを通 じて、ファージ感染 に抵抗 する[136][137]。 遺伝子 導入 の3番目 の方法 は接合 とよばれるプロセスであり、DNAは細胞 接触 によって他 の細菌 細胞 から直接 導入 される。通常 の状況 では、形質 導入 、接合 、および形質 転換 には、同 じ種 間 でのDNA移動 が含 まれるほか、異 なる細菌 種 の個体 間 での移動 も発生 する場合 があり、これは抗生 物質 耐 性 の移動 などの重大 な結果 をもたらす可能 性 がある[138][139]。このような場合 、他 の細菌 や環境 からの遺伝子 獲得 は遺伝子 水平 伝播 と呼 ばれ、自然 条件下 で広範 に発生 していると考 えられている[140]。
運動 性
リステリア
細胞 間 コミュニケーション
系統 分類 と同定
※
このような
1884
細菌 ドメインの主 な分類
これらの
他 の生物 との相互 作用
共生
捕食
相 利 共生 者
病原 性 細菌
ヒトや
それぞれの
産業 技術 への応用
ラクトバチルスやラクトコッカスといった
また、
歴史
1828
1859
フェルディナント・コーンは
19
しかし、1977
カール・ウーズにより
脚注
出典
- ^ Jean P. Euzéby, Aidan C. Parte. “Domain Bacteria”. List of Prokaryotic names with Standing in Nomenclature. 2024
年 7月 17日 閲覧 。 - ^ bacteria (n.)Online Etymology Dictionary
- ^ Krasner 2014, p. 38.
- ^ “bacteria | Origin and meaning of bacteria by Online Etymology Dictionary”. Online Etymology Dictionary. 2020
年 4月 18日 閲覧 。 - ^ βακτηρία in Liddell and Scott.
- ^ Harper, Douglas. "bacteria". Online Etymology Dictionary.
- ^ C R Woese, O Kandler, M L Wheelis (June 1990). “Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya”. Proceedings of the National Academy of Sciences of the United States of America 87 (12): 4576–79. Bibcode: 1990PNAS...87.4576W. doi:10.1073/pnas.87.12.4576. PMC 54159. PMID 2112744.
- ^ Filipa Godoy-Vitorino (July 2019). “Human microbial ecology and the rising new medicine”. Annals of Translational Medicine 7 (14): 342. doi:10.21037/atm.2019.06.56. PMC 6694241. PMID 31475212.
- ^ J W Schopf (July 1994). “Disparate rates, differing fates: tempo and mode of evolution changed from the Precambrian to the Phanerozoic”. Proceedings of the National Academy of Sciences of the United States of America 91 (15): 6735–42. Bibcode: 1994PNAS...91.6735S. doi:10.1073/pnas.91.15.6735. PMC 44277. PMID 8041691.
- ^ Edward F. DeLong, Norman R. Pace (August 2001). “Environmental diversity of bacteria and archaea”. Systematic Biology 50 (4): 470-478. doi:10.1080/10635150118513.
- ^ J R Brown, W F Doolittle (December 1997). “Archaea and the prokaryote-to-eukaryote transition”. Microbiology and Molecular Biology Reviews 61 (4): 456-502. doi:10.1128/mmbr.61.4.456-502.1997.
- ^ Bertram Daum, Vicki Gold (June 2018). “Twitch or swim: towards the understanding of prokaryotic motion based on the type IV pilus blueprint”. Biological Chemistry 399 (7): 799-808. doi:10.1515/hsz-2018-0157. PMID 29894297.
- ^ “The universal ancestor and the ancestor of bacteria were hyperthermophiles”. Journal of Molecular Evolution 57 (6): 721–30. (December 2003). Bibcode: 2003JMolE..57..721D. doi:10.1007/s00239-003-2522-6. PMID 14745541.
- ^ Battistuzzi, Fabia U.; Feijao, Andreia; Hedges, S. Blair (November 2004). “A genomic timescale of prokaryote evolution: insights into the origin of methanogenesis, phototrophy, and the colonization of land”. BMC Evolutionary Biology 4: 44. doi:10.1186/1471-2148-4-44. PMC 533871. PMID 15535883.
- ^ Homann, Martin (23 July 2018). “Microbial life and biogeochemical cycling on land 3,220 million years ago”. Nature Geoscience 11 (9): 665–671. Bibcode: 2018NatGe..11..665H. doi:10.1038/s41561-018-0190-9et al.
- ^ Van Kranendonk, Martin J.; Baumgartner, Raphael; Djokic, Tara; Ota, Tsutomu; Steller, Luke; Garbe, Ulf; Nakamura, Eizo (2021-01-01). “Elements for the Origin of Life on Land: A Deep-Time Perspective from the Pilbara Craton of Western Australia” (
英語 ). Astrobiology 21 (1): 39–59. doi:10.1089/ast.2019.2107. ISSN 1531-1074. - ^ López-García, Purificación; Moreira, David (2020-05). “The Syntrophy hypothesis for the origin of eukaryotes revisited” (
英語 ). Nature Microbiology 5 (5): 655–667. doi:10.1038/s41564-020-0710-4. ISSN 2058-5276. - ^ “Why is primary endosymbiosis so rare?”. The New Phytologist 231 (5): 1693–1699. (May 2021). doi:10.1111/nph.17478. PMID 34018613.
- ^ Stephens, Timothy G. and Gabr, Arwa and Calatrava, Victoria and Grossman, Arthur R. and Bhattacharya, Debashish (October 2020). “Extremophilic Microorganisms for the Treatment of Toxic Pollutants in the Environment”. Molecules (Basel, Switzerland) 25 (21): 4916. doi:10.3390/molecules25214916. PMC 7660605. PMID 33114255.
- ^ “Life in acid: pH homeostasis in acidophiles”. Trends in Microbiology 15 (4): 165–71. (April 2007). doi:10.1016/j.tim.2007.02.005. PMID 17331729.
- ^ Flemming, Hans-Curt; Wuertz, Stefan (April 2019). “Bacteria and archaea on Earth and their abundance in biofilms”. Nature Reviews. Microbiology 17 (4): 247–260. doi:10.1038/s41579-019-0158-9. PMID 30760902.
- ^ Wheelis 2008, p. 362.
- ^ Kushkevych, Ivan and Procházka, Jiří and Gajdács, Márió and Rittmann, Simon K.-M. R. and Vítězová, Monika (June 2021). “Molecular Physiology of Anaerobic Phototrophic Purple and Green Sulfur Bacteria”. International Journal of Molecular Sciences 22 (12): 6398. doi:10.3390/ijms22126398. PMC 8232776. PMID 34203823.
- ^ a b c Pommerville 2014, p. 3–6.
- ^ a b c
平松 啓一 ・中込 治 編集 「第 III章 細菌 学 総論 」『標準 微生物 学 』(10版 )、2009年 。ISBN 978-4-260-00638-5。 - ^ Yinon M. Bar-On, Rob Phillips, Ron Milo (June 2018). “The biomass distribution on Earth”. Proceedings of the National Academy of Sciences of the United States of America 115 (25): 6506–11. doi:10.1073/pnas.1711842115. PMC 6016768. PMID 29784790.
- ^ Krasner 2014, p. 74.
- ^ Heide N. Schulz; Bo Barker Jørgensen (2001). “Big bacteria”. Annual Review of Microbiology 55: 105–37. doi:10.1146/annurev.micro.55.1.105. PMID 11544351.
- ^ Williams, Caroline (2011). “Who are you calling simple?”. New Scientist 211 (2821): 38–41. doi:10.1016/S0262-4079(11)61709-0.
- ^ Jean-Marie Volland and Silvina Gonzalez-Rizzo and Olivier Gros and Tomáš Tyml and Natalia Ivanova and Frederik Schulz and Danielle Goudeau and Nathalie H. Elisabeth and Nandita Nath and Daniel Udwary and Rex R. Malmstrom and Chantal Guidi-Rontani and Susanne Bolte-Kluge and Karen M. Davies and Maïtena R. Jean and Jean-Louis Mansot and Nigel J. Mouncey and Esther R. Angert and Tanja Woyke and Shailesh V. Date (2022). “A centimeter-long bacterium with DNA contained in metabolically active, membrane-bound organelles”. Science 376 (6600): 1453-1458. doi:10.1126/science.abb3634.
- ^ J Robertson, M Gomersall, P Gill (November 1975). “Mycoplasma hominis: growth, reproduction, and isolation of small viable cells”. Journal of Bacteriology 124 (2): 1007–18. doi:10.1128/JB.124.2.1007-1018.1975. PMC 235991. PMID 1102522.
- ^ Branko Velimirov (2001). “Nanobacteria, Ultramicrobacteria and Starvation Forms: A Search for the Smallest Metabolizing Bacterium”. Microbes and Environments 16 (2): 67–77. doi:10.1264/jsme2.2001.67.
- ^ Dusenbery, David B (2009).
- ^ Desirée C. Yang, Kris M. Blair, Nina R. Salama (March 2016). “Staying in Shape: the Impact of Cell Shape on Bacterial Survival in Diverse Environments”. Microbiology and Molecular Biology Reviews 80 (1): 187–203. doi:10.1128/MMBR.00031-15. PMC 4771367. PMID 26864431.
- ^ “Bacterial cell shape”. Nature Reviews. Microbiology 3 (8): 601–10. (August 2005). doi:10.1038/nrmicro1205. PMID 16012516.
- ^ Kevin D. Young (September 2006). “The selective value of bacterial shape”. Microbiology and Molecular Biology Reviews 70 (3): 660–703. doi:10.1128/MMBR.00001-06. PMC 1594593. PMID 16959965.
- ^ Crawford 2007, p. xi.
- ^ Claessen, Dennis; Rozen, Daniel E.; Kuipers, Oscar P.; Søgaard-Andersen, Lotte; van Wezel, Gilles P. (February 2014). “Bacterial solutions to multicellularity: a tale of biofilms, filaments and fruiting bodies”. Nature Reviews. Microbiology 12 (2): 115–24. doi:10.1038/nrmicro3178. PMID 24384602.
- ^ Lawrence J. Shimkets (1999). “Intercellular signaling during fruiting-body development of Myxococcus xanthus”. Annual Review of Microbiology 53: 525–49. doi:10.1146/annurev.micro.53.1.525. PMID 10547700.
- ^ Dale Kaiser (2004). “Signaling in myxobacteria”. Annual Review of Microbiology 58: 75–98. doi:10.1146/annurev.micro.58.030603.123620. PMID 15487930.
- ^ Wheelis 2008, p. 75.
- ^ Abhishek Mandal, Ahana Dutta, Reshmi Das, Joydeep Mukherjee (June 2021). “Role of intertidal microbial communities in carbon dioxide sequestration and pollutant removal: A review”. Marine Pollution Bulletin 170: 112626. doi:10.1016/j.marpolbul.2021.112626. PMID 34153859.
- ^ Rodney M. Donlan (September 2002). “Biofilms: microbial life on surfaces”. Emerging Infectious Diseases 8 (9): 881–90. doi:10.3201/eid0809.020063. PMC 2732559. PMID 12194761.
- ^ Steven S. Branda, Åshild Vik, Lisa Friedman, Roberto Kolter (January 2005). “Biofilms: the matrix revisited”. Trends in Microbiology 13 (1): 20–26. doi:10.1016/j.tim.2004.11.006. PMID 15639628.
- ^ Mary Ellen Davey, George A. O'toole (December 2000). “Microbial biofilms: from ecology to molecular genetics”. Microbiology and Molecular Biology Reviews 64 (4): 847–67. doi:10.1128/MMBR.64.4.847-867.2000. PMC 99016. PMID 11104821.
- ^ Rodney M. Donlan and J. William Costerton (April 2002). “Biofilms: survival mechanisms of clinically relevant microorganisms”. Clinical Microbiology Reviews 15 (2): 167–93. doi:10.1128/CMR.15.2.167-193.2002. PMC 118068. PMID 11932229.
- ^ Microbiology : an Evolving Science (Third ed.). New York: W W Norton. (2013). p. 82. ISBN 978-0393123678
- ^ Feijoo-Siota, Lucía; Rama, José Luis R.; Sánchez-Pérez, Angeles; Villa, Tomás G. (July 2017). “Considerations on bacterial nucleoids”. Applied Microbiology and Biotechnology 101 (14): 5591–602. doi:10.1007/s00253-017-8381-7. PMID 28664324.
- ^ Thomas A. Bobik (May 2006). “Polyhedral organelles compartmenting bacterial metabolic processes”. Applied Microbiology and Biotechnology 70 (5): 517–25. doi:10.1007/s00253-005-0295-0. PMID 16525780.
- ^ Yeates, Todd O.; Kerfeld, Cheryl A.; Heinhorst, Sabine; Cannon, Gordon C.; Shively, Jessup M. (September 2008). “Protein-based organelles in bacteria: carboxysomes and related microcompartments”. Nature Reviews. Microbiology 6 (9): 681–91. doi:10.1038/nrmicro1913. PMID 18679172.
- ^ Kerfeld, Cheryl A and Sawaya, Michael R and Tanaka, Shiho and Nguyen, Chau V and Phillips, Martin and Beeby, Morgan and Yeates, Todd O (August 2005). “Protein structures forming the shell of primitive bacterial organelles”. Science 309 (5736): 936–38. Bibcode: 2005Sci...309..936K. doi:10.1126/science.1113397. PMID 16081736.
- ^ Gitai, Zemer (March 2005). “The new bacterial cell biology: moving parts and subcellular architecture”. Cell 120 (5): 577–86. doi:10.1016/j.cell.2005.02.026. PMID 15766522.
- ^ Yu-Ling Shih, Lawrence Rothfield (September 2006). “The bacterial cytoskeleton”. Microbiology and Molecular Biology Reviews 70 (3): 729–54. doi:10.1128/MMBR.00017-06. PMC 1594594. PMID 16959967.
- ^ Norris, Vic and Den Blaauwen, Tanneke and Cabin-Flaman, Armelle and Doi, Roy H and Harshey, Rasika and Janniere, Laurent and Jimenez-Sanchez, Alfonso and Jin, Ding Jun and Levin, Petra Anne and Mileykovskaya, Eugenia and others (March 2007). “Functional taxonomy of bacterial hyperstructures”. Microbiology and Molecular Biology Reviews 71 (1): 230–53. doi:10.1128/MMBR.00035-06. PMC 1847379. PMID 17347523.
- ^ Pommerville 2014, pp. 120–121.
- ^ Bryant, Donald A and Frigaard, Niels-Ulrik (November 2006). “Prokaryotic photosynthesis and phototrophy illuminated”. Trends in Microbiology 14 (11): 488–96. doi:10.1016/j.tim.2006.09.001. PMID 16997562.
- ^ Pšenčík, J., et al. (August 2004). “Lamellar organization of pigments in chlorosomes, the light harvesting complexes of green photosynthetic bacteria”. Biophysical Journal 87 (2): 1165–72. Bibcode: 2004BpJ....87.1165P. doi:10.1529/biophysj.104.040956. PMC 1304455. PMID 15298919.
- ^ Martin Thanbichler, Sherry C. Wang, Lucy Shapiro (October 2005). “The bacterial nucleoid: a highly organized and dynamic structure”. Journal of Cellular Biochemistry 96 (3): 506–21. doi:10.1002/jcb.20519. PMID 15988757.
- ^ Poehlsgaard, Jacob and Douthwaite, Stephen (November 2005). “The bacterial ribosome as a target for antibiotics”. Nature Reviews. Microbiology 3 (11): 870–81. doi:10.1038/nrmicro1265. PMID 16261170.
- ^ Yeo, Marcus and Chater, Keith (March 2005). “The interplay of glycogen metabolism and differentiation provides an insight into the developmental biology of Streptomyces coelicolor”. Microbiology 151 (Pt 3): 855–61. doi:10.1099/mic.0.27428-0. PMID 15758231.
- ^ Shiba, T and Tsutsumi, K and Ishige, K and Noguchi, T (March 2000). “Inorganic polyphosphate and polyphosphate kinase: their novel biological functions and applications”. Biochemistry. Biokhimiia 65 (3): 315–23. PMID 10739474.
- ^ Brune, Daniel C (June 1995). “Isolation and characterization of sulfur globule proteins from Chromatium vinosum and Thiocapsa roseopersicina”. Archives of Microbiology 163 (6): 391–99. doi:10.1007/BF00272127. PMID 7575095.
- ^ Kadouri, Daniel; Jurkevitch, Edouard; Okon, Yaacov; Castro-Sowinski, Susana (2005). “Ecological and agricultural significance of bacterial polyhydroxyalkanoates”. Critical Reviews in Microbiology 31 (2): 55–67. doi:10.1080/10408410590899228. PMID 15986831.
- ^ A E Walsby (March 1994). “Gas vesicles”. Microbiological Reviews 58 (1): 94–144. doi:10.1128/MMBR.58.1.94-144.1994. PMC 372955. PMID 8177173.
- ^ Heijenoort, Jean van (March 2001). “Formation of the glycan chains in the synthesis of bacterial peptidoglycan”. Glycobiology 11 (3): 25R–36R. doi:10.1093/glycob/11.3.25R. PMID 11320055.
- ^ a b Arthur L. Koch (October 2003). “Bacterial wall as target for attack: past, present, and future research”. Clinical Microbiology Reviews 16 (4): 673–87. doi:10.1128/CMR.16.4.673-687.2003. PMC 207114. PMID 14557293.
- ^ Gram, H Christian (1884). “Über die isolierte Färbung der Schizomyceten in Schnitt- und Trockenpräparaten”. Fortschr. Med. 2: 185–89.
- ^ Hugenholtz, Philip (2002). “Exploring prokaryotic diversity in the genomic era”. Genome Biology (Springer) 3 (2): 1-8. doi:10.1186/gb-2002-3-2-reviews0003. PMC 139013. PMID 11864374.
- ^ Fiona M Walsh and Sebastian GB Amyes (October 2004). “Microbiology and drug resistance mechanisms of fully resistant pathogens”. Current Opinion in Microbiology 7 (5): 439–44. doi:10.1016/j.mib.2004.08.007. PMID 15451497.
- ^ Alderwick, Luke J and Harrison, James and Lloyd, Georgina S and Birch, Helen L (March 2015). “The Mycobacterial Cell Wall – Peptidoglycan and Arabinogalactan”. Cold Spring Harbor Perspectives in Medicine 5 (8): a021113. doi:10.1101/cshperspect.a021113. PMC 4526729. PMID 25818664.
- ^ Alderwick, Luke J and Harrison, James and Lloyd, Georgina S and Birch, Helen L (March 2014). “Biogenesis and functions of bacterial S-layers”. Nature Reviews. Microbiology 12 (3): 211–22. doi:10.1038/nrmicro3213. PMID 24509785.
- ^ Thompson, Stuart A (December 2002). “Campylobacter surface-layers (S-layers) and immune evasion”. Annals of Periodontology 7 (1): 43-53. doi:10.1902/annals.2002.7.1.43. PMC 2763180. PMID 16013216.
- ^ Beveridge TJ, Pouwels PH, Sára M, et al. (June 1997). “Functions of S-layers”. FEMS Microbiology Reviews 20 (1–2): 99-149. doi:10.1111/j.1574-6976.1997.tb00305.x. PMID 9276929.
- ^ Seiji Kojima, David F Blair (2004). The bacterial flagellar motor: structure and function of a complex molecular machine. International Review of Cytology. 233. pp. 93–134. doi:10.1016/S0074-7696(04)33003-2. ISBN 978-0-12-364637-8. PMID 15037363
- ^ Wheelis 2008, p. 76.
- ^ Cheng, Rachel A and Wiedmann, Martin (2020). “Recent Advances in Our Understanding of the Diversity and Roles of Chaperone-Usher Fimbriae in Facilitating Salmonella Host and Tissue Tropism”. Frontiers in Cellular and Infection Microbiology 10: 628043. doi:10.3389/fcimb.2020.628043. PMC 7886704. PMID 33614531.
- ^ “Towards a structural biology of bacterial conjugation”. Molecular Microbiology 23 (3): 423–29. (February 1997). doi:10.1046/j.1365-2958.1997.2411604.x. PMID 9044277.
- ^ “Secretion systems in Gram-negative bacteria: structural and mechanistic insights”. Nature Reviews. Microbiology 13 (6): 343–59. (June 2015). doi:10.1038/nrmicro3456. PMID 25978706.
- ^ “Dismantling the bacterial glycocalyx: Chemical tools to probe, perturb, and image bacterial glycans”. Bioorganic & Medicinal Chemistry 42: 116268. (July 2021). doi:10.1016/j.bmc.2021.116268. ISSN 0968-0896. PMC 8276522. PMID 34130219.
- ^ “The glycan-rich outer layer of the cell wall of Mycobacterium tuberculosis acts as an antiphagocytic capsule limiting the association of the bacterium with macrophages”. Infection and Immunity 72 (10): 5676–86. (October 2004). doi:10.1128/IAI.72.10.5676-5686.2004. PMC 517526. PMID 15385466.
- ^ “The Mycobacterium tuberculosis capsule: a cell structure with key implications in pathogenesis”. The Biochemical Journal 476 (14): 1995–2016. (July 2019). doi:10.1042/BCJ20190324. PMC 6698057. PMID 31320388.
- ^ Kalscheuer, Rainer, et al. (July 2019). “The Mycobacterium tuberculosis capsule: a cell structure with key implications in pathogenesis”. The Biochemical Journal 476 (14): 1995–2016. doi:10.1042/BCJ20190324. PMC 6698057. PMID 31320388.
- ^ Koga, Yosuke (2011-03-01). “Early Evolution of Membrane Lipids: How did the Lipid Divide Occur?” (
英語 ). Journal of Molecular Evolution 72 (3): 274–282. doi:10.1007/s00239-011-9428-5. ISSN 1432-1432. - ^ Fuerst, John A.; Sagulenko, Evgeny (2011-06). “Beyond the bacterium: planctomycetes challenge our concepts of microbial structure and function” (
英語 ). Nature Reviews Microbiology 9 (6): 403–413. doi:10.1038/nrmicro2578. ISSN 1740-1526. - ^ Azam, Talukder Ali; Ishihama, Akira (1999-11-12). “Twelve Species of the Nucleoid-associated Protein from Escherichia coli: SEQUENCE RECOGNITION SPECIFICITY AND DNA BINDING AFFINITY *” (English). Journal of Biological Chemistry 274 (46): 33105–33113. doi:10.1074/jbc.274.46.33105. ISSN 0021-9258. PMID 10551881.
- ^ Jernigan, John A., et al. (2001). “Bioterrorism-Related Inhalational Anthrax: The First 10 Cases Reported in the United States”. Emerging Infectious Diseases 7 (6): 933–44. doi:10.3201/eid0706.010604. PMC 2631903. PMID 11747719.
- ^ Nicholson, Wayne L and Munakata, Nobuo and Horneck, Gerda and Melosh, Henry J and Setlow, Peter (September 2000). “Resistance of Bacillus endospores to extreme terrestrial and extraterrestrial environments”. Microbiology and Molecular Biology Reviews 64 (3): 548–72. doi:10.1128/MMBR.64.3.548-572.2000. PMC 99004. PMID 10974126.
- ^ a b “The Bacillus subtilis endospore: assembly and functions of the multilayered coat”. Nature Reviews. Microbiology 11 (1): 33–44. (January 2013). doi:10.1038/nrmicro2921. PMID 23202530.
- ^ “Bacterial endospores and their significance in stress resistance”. Antonie van Leeuwenhoek 81 (1–4): 27–32. (August 2002). doi:10.1023/A:1020561122764. PMID 12448702.
- ^ “Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal”. Nature 407 (6806): 897–900. (October 2000). Bibcode: 2000Natur.407..897V. doi:10.1038/35038060. PMID 11057666.
- ^ “Revival and identification of bacterial spores in 25- to 40-million-year-old Dominican amber”. Science 268 (5213): 1060–64. (May 1995). Bibcode: 1995Sci...268.1060C. doi:10.1126/science.7538699. PMID 7538699.
- ^ “Row over ancient bacteria” (
英語 ). BBC News. (2001年 6月 7日 ) 2020年 4月 26日 閲覧 。 - ^ “The solar UV environment and bacterial spore UV resistance: considerations for Earth-to-Mars transport by natural processes and human spaceflight”. Mutation Research 571 (1–2): 249–64. (April 2005). doi:10.1016/j.mrfmmm.2004.10.012. PMID 15748651.
- ^ “Colonising the galaxy is hard. Why not send bacteria instead?”. The Economist. (2018
年 4月 12日 ). ISSN 0013-0613 2020年 4月 26日 閲覧 。 - ^ “Virulence Plasmids of the Pathogenic Clostridia”. Microbiology Spectrum 7 (3). (May 2019). doi:10.1128/microbiolspec.GPP3-0034-2018. PMID 31111816.
- ^ “How to: prophylactic interventions for prevention of Clostridioides difficile infection”. Clinical Microbiology and Infection 27 (12): 1777–1783. (July 2021). doi:10.1016/j.cmi.2021.06.037. PMID 34245901.
- ^ “Post-Viking microbiology: new approaches, new data, new insights”. Origins of Life and Evolution of the Biosphere 29 (1): 73–93. (January 1999). Bibcode: 1999OLEB...29...73N. doi:10.1023/A:1006515817767. PMID 11536899.
- ^ “Microbial ecology in the age of genomics and metagenomics: concepts, tools, and recent advances”. Molecular Ecology 15 (7): 1713–31. (June 2006). doi:10.1111/j.1365-294X.2006.02882.x. PMID 16689892.
- ^ “Comparative biochemistry of Archaea and Bacteria”. Current Opinion in Genetics & Development 1 (4): 544–51. (December 1991). doi:10.1016/S0959-437X(05)80206-0. PMID 1822288.
- ^ a b c Microbiology: An Evolving Science (3 ed.). WW Norton & Company. pp. 491–44
- ^ “Photobiology of bacteria”. Antonie van Leeuwenhoek 65 (4): 331–47. (1994). doi:10.1007/BF00872217. PMID 7832590.
- ^ “The Leeuwenhoek Lecture 2000 the natural and unnatural history of methane-oxidizing bacteria”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 360 (1458): 1207–22. (June 2005). doi:10.1098/rstb.2005.1657. PMC 1569495. PMID 16147517.
- ^ “Diazotrophs for Lowering Nitrogen Pollution Crises: Looking Deep Into the Roots”. Frontiers in Microbiology 12: 637815. (2021). doi:10.3389/fmicb.2021.637815. PMC 8180554. PMID 34108945.
- ^ “Nitrogenase gene diversity and microbial community structure: a cross-system comparison”. Environmental Microbiology 5 (7): 539–54. (July 2003). doi:10.1046/j.1462-2920.2003.00451.x. PMID 12823187.
- ^ “Wastewater Treatment using the "Sulfate Reduction, DenitrificationAnammox and Partial Nitrification (SRDAPN)" Process”. Chemosphere 256: 127092. (October 2020). Bibcode: 2020Chmsp.256l7092K. doi:10.1016/j.chemosphere.2020.127092. PMID 32559887.
- ^ “The chemical cycle and bioaccumulation of mercury”. Annual Review of Ecology and Systematics 29: 543–66. (1998). doi:10.1146/annurev.ecolsys.29.1.543.
- ^ “How to define obligatory anaerobiosis? An evolutionary view on the antioxidant response system and the early stages of the evolution of life on Earth”. Free Radical Biology & Medicine 140: 61–73. (August 2019). doi:10.1016/j.freeradbiomed.2019.03.004. PMID 30862543.
- ^ “Control of the bacterial cell cycle by cytoplasmic growth”. Critical Reviews in Microbiology 28 (1): 61–77. (2002). doi:10.1080/1040-840291046696. PMID 12003041.
- ^ Pommerville 2014, p. 138.
- ^ Pommerville 2014, p. 557.
- ^ a b Wheelis 2008, p. 42.
- ^ “Laboratory diagnosis of central nervous system infections”. Infectious Disease Clinics of North America 15 (4): 1047–71. (December 2001). doi:10.1016/S0891-5520(05)70186-0. PMID 11780267.
- ^ “Harmful freshwater algal blooms, with an emphasis on cyanobacteria”. TheScientificWorldJournal 1: 76–113. (April 2001). doi:10.1100/tsw.2001.16. PMC 6083932. PMID 12805693.
- ^ “Synergy and contingency as driving forces for the evolution of multiple secondary metabolite production by Streptomyces species”. Proceedings of the National Academy of Sciences of the United States of America 100 Suppl 2 (90002): 14555–61. (November 2003). Bibcode: 2003PNAS..10014555C. doi:10.1073/pnas.1934677100. PMC 304118. PMID 12970466.
- ^ “Microbial biofilms: from ecology to molecular genetics”. Microbiology and Molecular Biology Reviews 64 (4): 847–67. (December 2000). doi:10.1128/MMBR.64.4.847-867.2000. PMC 99016. PMID 11104821.
- ^ “Quantitative steps in symbiogenesis and the evolution of homeostasis”. Biological Reviews of the Cambridge Philosophical Society 78 (3): 435–63. (August 2003). doi:10.1017/S1464793102006127. PMID 14558592.
- ^ “Lag Phase is a Dynamic, Organized, Adaptive, and Evolvable Period that Prepares Bacteria for Cell Division”. Journal of Bacteriology 201 (7): e00697-18. (2019). doi:10.1128/JB.00697-18. PMC 6416914. PMID 30642990.
- ^ “Individual-based modelling of bacterial cultures to study the microscopic causes of the lag phase”. Journal of Theoretical Biology 241 (4): 939–53. (August 2006). Bibcode: 2006JThBi.241..939P. doi:10.1016/j.jtbi.2006.01.029. PMID 16524598.
- ^ General stress response of Bacillus subtilis and other bacteria. Advances in Microbial Physiology. 44. (2001). pp. 35–91. doi:10.1016/S0065-2911(01)44011-2. ISBN 978-0-12-027744-5. PMID 11407115
- ^ Microbiology: An Evolving Science (3 ed.). WW Norton & Company. p. 143
- ^ Leppänen, Miika; Sundberg, Lotta-Riina; Laanto, Elina; De Freitas Almeida, Gabriel Magno; Papponen, Petri; Maasilta, Ilari J. (2017). “Imaging Bacterial Colonies and Phage-Bacterium Interaction at Sub-Nanometer Resolution Using Helium-Ion Microscopy”. Advanced Biosystems 1 (8): e1700070. doi:10.1002/adbi.201700070. PMID 32646179.
- ^ “The 160-kilobase genome of the bacterial endosymbiont Carsonella”. Science 314 (5797): 267. (October 2006). doi:10.1126/science.1134196. PMID 17038615.
- ^ “Characterisation, genome size and genetic manipulation of the myxobacterium Sorangium cellulosum So ce56”. Archives of Microbiology 178 (6): 484–92. (December 2002). doi:10.1007/s00203-002-0479-2. PMID 12420170.
- ^ “Linear plasmids and chromosomes in bacteria”. Molecular Microbiology 10 (5): 917–22. (December 1993). doi:10.1111/j.1365-2958.1993.tb00963.x. PMID 7934868.
- ^ “The chromosomal DNA of Streptomyces lividans 66 is linear”. Molecular Microbiology 10 (5): 923–33. (December 1993). doi:10.1111/j.1365-2958.1993.tb00964.x. PMID 7934869.
- ^ “Management of multipartite genomes: the Vibrio cholerae model”. Current Opinion in Microbiology 22: 120–26. (December 2014). doi:10.1016/j.mib.2014.10.003. PMID 25460805.
- ^ “Historical events that spawned the field of plasmid biology”. Microbiology Spectrum 2 (5): 3. (October 2014). doi:10.1128/microbiolspec.PLAS-0019-2013. PMID 26104369.
- ^ “Prokaryotic introns and inteins: a panoply of form and function”. Journal of Bacteriology 177 (14): 3897–903. (July 1995). doi:10.1128/jb.177.14.3897-3903.1995. PMC 177115. PMID 7608058.
- ^ “Evolution of mutation rates in bacteria”. Molecular Microbiology 60 (4): 820–27. (May 2006). doi:10.1111/j.1365-2958.2006.05150.x. PMID 16677295.
- ^ “Stress-directed adaptive mutations and evolution”. Molecular Microbiology 52 (3): 643–50. (May 2004). doi:10.1111/j.1365-2958.2004.04012.x. PMID 15101972.
- ^ “DNA uptake during bacterial transformation”. Nature Reviews. Microbiology 2 (3): 241–49. (March 2004). doi:10.1038/nrmicro844. PMID 15083159.
- ^ “Natural genetic transformation: prevalence, mechanisms and function”. Research in Microbiology 158 (10): 767–78. (December 2007). doi:10.1016/j.resmic.2007.09.004. PMID 17997281.
- ^ Bernstein H, Bernstein C, Michod RE (2012).
- ^ “Phages and the evolution of bacterial pathogens: from genomic rearrangements to lysogenic conversion”. Microbiology and Molecular Biology Reviews 68 (3): 560–602, table of contents. (September 2004). doi:10.1128/MMBR.68.3.560-602.2004. PMC 515249. PMID 15353570.
- ^ “Biology of DNA restriction”. Microbiological Reviews 57 (2): 434–50. (June 1993). doi:10.1128/MMBR.57.2.434-450.1993. PMC 372918. PMID 8336674.
- ^ “CRISPR provides acquired resistance against viruses in prokaryotes”. Science 315 (5819): 1709–12. (March 2007). Bibcode: 2007Sci...315.1709B. doi:10.1126/science.1138140. PMID 17379808.
- ^ “Small CRISPR RNAs guide antiviral defense in prokaryotes”. Science 321 (5891): 960–64. (August 2008). Bibcode: 2008Sci...321..960B. doi:10.1126/science.1159689. PMC 5898235. PMID 18703739.
- ^ “Adaptive value of sex in microbial pathogens”. Infection, Genetics and Evolution 8 (3): 267–85. (May 2008). doi:10.1016/j.meegid.2008.01.002. PMID 18295550.
- ^ “Antibiotic-induced lateral transfer of antibiotic resistance”. Trends in Microbiology 12 (9): 401–14. (September 2004). doi:10.1016/j.tim.2004.07.003. PMID 15337159.
- ^ “Genetic exchange between bacteria in the environment”. Plasmid 42 (2): 73–91. (September 1999). doi:10.1006/plas.1999.1421. PMID 10489325.
- ^ a b “Electron microscopic observations of prokaryotic surface appendages”. Journal of Microbiology (Seoul, Korea) 55 (12): 919–26. (December 2017). doi:10.1007/s12275-017-7369-4. PMID 29214488.
- ^ “The bacterial flagellum: reversible rotary propellor and type III export apparatus”. Journal of Bacteriology 181 (23): 7149–53. (December 1999). doi:10.1128/JB.181.23.7149-7153.1999. PMC 103673. PMID 10572114.
- ^ “Collective bacterial dynamics revealed using a three-dimensional population-scale defocused particle tracking technique”. Applied and Environmental Microbiology 72 (7): 4987–94. (July 2006). Bibcode: 2006ApEnM..72.4987W. doi:10.1128/AEM.00158-06. PMC 1489374. PMID 16820497.
- ^ “Electron microscopic observations of prokaryotic surface appendages”. Journal of Microbiology (Seoul, Korea) 55 (12): 919–26. (December 2017). doi:10.1007/s12275-017-7369-4. PMID 29214488.
- ^ Mattick, John S (2002). “Type IV Pili and Twitching Motility”. Annual Review of Microbiology 56: 289–314. doi:10.1146/annurev.micro.56.012302.160938. PMID 12142488.
- ^ “Pilus retraction powers bacterial twitching motility”. Nature 407 (6800): 98–102. (September 2000). Bibcode: 2000Natur.407...98M. doi:10.1038/35024105. PMID 10993081.
- ^ “Chemotaxis-guided movements in bacteria”. Critical Reviews in Oral Biology and Medicine 15 (4): 207–20. (July 2004). doi:10.1177/154411130401500404. PMID 15284186.
- ^ “Bacterial energy taxis: a global strategy?”. Archives of Microbiology 192 (7): 507–20. (July 2010). doi:10.1007/s00203-010-0575-7. PMC 2886117. PMID 20411245.
- ^ “Magneto-aerotaxis in marine coccoid bacteria”. Biophysical Journal 73 (2): 994–1000. (August 1997). Bibcode: 1997BpJ....73..994F. doi:10.1016/S0006-3495(97)78132-3. PMC 1180996. PMID 9251816.
- ^ “Signaling in myxobacteria”. Annual Review of Microbiology 58: 75–98. (2004). doi:10.1146/annurev.micro.58.030603.123620. PMID 15487930.
- ^ “Uncovering the mystery of gliding motility in the myxobacteria”. Annual Review of Genetics 45: 21–39. (2011). doi:10.1146/annurev-genet-110410-132547. PMC 3397683. PMID 21910630.
- ^ “Actin-based motility of intracellular microbial pathogens”. Microbiology and Molecular Biology Reviews 65 (4): 595–626, table of contents. (December 2001). doi:10.1128/MMBR.65.4.595-626.2001. PMC 99042. PMID 11729265.
- ^ “Bacterial Semiochemicals and Transkingdom Interactions with Insects and Plants”. Insects 10 (12): 441. (December 2019). doi:10.3390/insects10120441. PMC 6955855. PMID 31817999.
- ^ “Thinking about bacterial populations as multicellular organisms”. Annual Review of Microbiology 52: 81–104. (1998). doi:10.1146/annurev.micro.52.1.81. PMID 9891794.
- ^ “Microbial biofilms”. Annual Review of Microbiology 49: 711–45. (1995). doi:10.1146/annurev.mi.49.100195.003431. PMID 8561477.
- ^ “Thinking about bacterial populations as multicellular organisms”. Annual Review of Microbiology 52: 81–104. (1998). doi:10.1146/annurev.micro.52.1.81. PMID 9891794.
- ^ “Microbial biofilms”. Annual Review of Microbiology 49: 711–45. (1995). doi:10.1146/annurev.mi.49.100195.003431. PMID 8561477.
- ^ “Signaling systems in oral bacteria”. Advances in Experimental Medicine and Biology 1197: 27–43. (2019). doi:10.1007/978-3-030-28524-1_3. ISBN 978-3-030-28523-4. PMID 31732932.
- ^ “Bacterial Quorum sensing and microbial community interactions”. mBio 9 (3). (May 2018). doi:10.1128/mBio.02331-17. PMC 5964356. PMID 29789364.
- ^ “Quorum sensing in bacteria”. Annual Review of Microbiology 55: 165–99. (2001). doi:10.1146/annurev.micro.55.1.165. PMID 11544353.
- ^ Zhu, Qiyun; Mai, Uyen; Pfeiffer, Wayne; Janssen, Stefan; Asnicar, Francesco; Sanders, Jon G.; Belda-Ferre, Pedro; Al-Ghalith, Gabriel A. et al. (2019). “Phylogenomics of 10,575 genomes reveals evolutionary proximity between domains Bacteria and Archaea”. Nature Communications 10 (1): 5477. Bibcode: 2019NatCo..10.5477Z. doi:10.1038/s41467-019-13443-4. PMC 6889312. PMID 31792218.
- ^ “Laboratory diagnosis of central nervous system infections”. Infectious Disease Clinics of North America 15 (4): 1047–71. (December 2001). doi:10.1016/S0891-5520(05)70186-0. PMID 11780267.
- ^ “Lateral gene transfer and the origins of prokaryotic groups”. Annual Review of Genetics 37: 283–328. (2003). doi:10.1146/annurev.genet.37.050503.084247. PMID 14616063.
- ^ “The winds of (evolutionary) change: breathing new life into microbiology”. Journal of Bacteriology 176 (1): 1–6. (January 1994). doi:10.2172/205047. PMC 205007. PMID 8282683.
- ^ “IJSEM Home”. Ijs.sgmjournals.org (2011
年 10月 28日 ). 2011年 10月 19日 時点 のオリジナルよりアーカイブ。2011年 11月4日 閲覧 。 - ^ “Bergey's Manual Trust”. Bergeys.org. 2011
年 11月7日 時点 のオリジナルよりアーカイブ。2011年 11月4日 閲覧 。 - ^ “The changing landscape of microbial biodiversity exploration and its implications for systematics”. Systematic and Applied Microbiology 38 (4): 231–36. (June 2015). doi:10.1016/j.syapm.2015.03.003. PMID 25921438.
- ^ "Schizomycetes.”
- ^ “Staphylococcus epidermidis-Skin friend or foe?”. PLOS Pathogens 16 (11): e1009026. (November 2020). doi:10.1371/journal.ppat.1009026. PMC 7660545. PMID 33180890.
- ^ a b Hall 2008, p. 145.
- ^ “The natural evolutionary relationships among prokaryotes”. Critical Reviews in Microbiology 26 (2): 111–31. (2000). doi:10.1080/10408410091154219. PMID 10890353.
- ^ “The uncultured microbial majority”. Annual Review of Microbiology 57: 369–94. (2003). doi:10.1146/annurev.micro.57.030502.090759. PMID 14527284.
- ^ “Evolutionary aspects of whole-genome biology”. Current Opinion in Structural Biology 15 (3): 248–53. (June 2005). doi:10.1016/j.sbi.2005.04.001. PMID 15963888.
- ^ Pommerville 2014, p. 15−31.
- ^ Castelle, C.J., Banfield, J.F. (2018-03-08). “Major New Microbial Groups Expand Diversity and Alter our Understanding of the Tree of Life”. Cell 172 (6): 1181-1197. doi:10.1016/j.cell.2018.02.016. PMID 29522741.
- ^ a b Krasner 2014, p. 77.
- ^ Gram, HC (1884). “Über die isolierte Färbung der Schizomyceten in Schnitt- und Trockenpräparaten”. Fortschr. Med. 2: 185–89.
- ^ “Detection of infection or infectious agents by use of cytologic and histologic stains”. Clinical Microbiology Reviews 9 (3): 382–404. (July 1996). doi:10.1128/CMR.9.3.382. PMC 172900. PMID 8809467.
- ^ “Assessment and Comparison of Molecular Subtyping and Characterization Methods for Salmonella”. Frontiers in Microbiology 10: 1591. (2019). doi:10.3389/fmicb.2019.01591. PMC 6639432. PMID 31354679.
- ^ Krasner 2014, p. 87–89.
- ^ “Laboratory diagnosis of central nervous system infections”. Infectious Disease Clinics of North America 15 (4): 1047–71. (December 2001). doi:10.1016/S0891-5520(05)70186-0. PMID 11780267.
- ^ “Clinical importance of blood cultures”. Clinics in Laboratory Medicine 14 (1): 9–16. (March 1994). doi:10.1016/S0272-2712(18)30390-1. PMID 8181237.
- ^ “Laboratory Methods in Molecular Epidemiology: Bacterial Infections”. Microbiology Spectrum 6 (6). (November 2018). doi:10.1128/microbiolspec.AME-0004-2018. PMID 30387415.
- ^ “The Loop-Mediated Isothermal Amplification Technique in Periodontal Diagnostics: A Systematic Review”. Journal of Clinical Medicine 10 (6): 1189. (March 2021). doi:10.3390/jcm10061189. PMC 8000232. PMID 33809163.
- ^ “MALDI-TOF Mass Spectroscopy Applications in Clinical Microbiology”. Advances in Pharmacological and Pharmaceutical Sciences 2021: 9928238. (2021). doi:10.1155/2021/9928238. PMC 8121603. PMID 34041492.
- ^ “Novel Microbial Diversity and Functional Potential in the Marine Mammal Oral Microbiome”. Current Biology 27 (24): 3752–62. (2017). doi:10.1016/j.cub.2017.10.040. PMID 29153320.
- ^ “The role of DNA amplification technology in the diagnosis of infectious diseases”. CMAJ 163 (3): 301–09. (August 2000). doi:10.1016/s1381-1169(00)00220-x. PMC 80298. PMID 10951731.
- ^ “The viable but nonculturable state in bacteria”. Journal of Microbiology 43 Spec No: 93–100. (February 2005). PMID 15765062.
- ^ “Number of published names”. List of Prokaryotic names with Standing in Nomenclature (2011
年 12月8日 ). 2012年 1月 19日 時点 のオリジナルよりアーカイブ。2011年 12月 10日 閲覧 。 - ^ “Estimating prokaryotic diversity and its limits”. Proceedings of the National Academy of Sciences of the United States of America 99 (16): 10494–99. (August 2002). Bibcode: 2002PNAS...9910494C. doi:10.1073/pnas.142680199. PMC 124953. PMID 12097644.
- ^ “Status of the microbial census”. Microbiology and Molecular Biology Reviews 68 (4): 686–91. (December 2004). doi:10.1128/MMBR.68.4.686-691.2004. PMC 539005. PMID 15590780.
- ^ a b Hug, Laura A.; Baker, Brett J.; Anantharaman, Karthik; Brown, Christopher T.; Probst, Alexander J.; Castelle, Cindy J.; Butterfield, Cristina N.; Hernsdorf, Alex W. et al. (2016-05). “A new view of the tree of life” (
英語 ). Nature Microbiology 1 (5): 16048. doi:10.1038/nmicrobiol.2016.48. ISSN 2058-5276. - ^ Fisher, Bruce; Harvey, Richard P; Champe, Pamela C (2007). “Chapter 33”. Lippincott's Illustrated Reviews: Microbiology (Lippincott's Illustrated Reviews Series). Hagerstwon, MD: Lippincott Williams & Wilkins. pp. 367–92. ISBN 978-0-7817-8215-9
- ^ “Rethinking "mutualism" in diverse host-symbiont communities”. BioEssays 38 (1): 100–8. (January 2016). doi:10.1002/bies.201500074. PMID 26568407.
- ^ Harper, Douglas. "commensalism". Online Etymology Dictionary.
- ^ “A dynamic partnership: celebrating our gut flora”. Anaerobe 11 (5): 247–51. (October 2005). doi:10.1016/j.anaerobe.2005.05.001. PMID 16701579.
- ^ “Commensal Bacteria: An Emerging Player in Defense Against Respiratory Pathogens”. Frontiers in Immunology 10: 1203. (2019). doi:10.3389/fimmu.2019.01203. PMC 6554327. PMID 31214175.
- ^ “Normal flora and mucosal immunity of the head and neck”. Infectious Disease Clinics of North America 2 (1): 1–19. (March 1988). doi:10.1016/S0891-5520(20)30163-X. PMID 3074102.
- ^ “Commensal bacteria and fungi differentially regulate tumor responses to radiation therapy”. Cancer Cell 39 (9): 1202–1213.e6. (July 2021). doi:10.1016/j.ccell.2021.07.002. PMID 34329585.
- ^ “MALT Lymphoma of the Urinary Bladder Shows a Dramatic Female Predominance, Uneven Geographic Distribution, and Possible Infectious Etiology”. Research and Reports in Urology 13: 49–62. (2021). doi:10.2147/RRU.S283366. PMC 7873029. PMID 33575225.
- ^ “Infective endocarditis in paediatric population”. European Journal of Pediatrics 180 (10): 3089–3100. (April 2021). doi:10.1007/s00431-021-04062-7. PMID 33852085.
- ^ “Predatory prokaryotes: an emerging research opportunity”. Journal of Molecular Microbiology and Biotechnology 4 (5): 467–77. (September 2002). PMID 12432957.
- ^ “Experimental social evolution with Myxococcus xanthus”. Antonie van Leeuwenhoek 81 (1–4): 155–64. (August 2002). doi:10.1023/A:1020546130033. PMID 12448714.
- ^ “Bacterial Predation on Cyanobacteria”. Microbial Physiology 31 (2): 99–108. (May 2021). doi:10.1159/000516427. ISSN 2673-1665. PMID 34010833.
- ^ “Bacterial predators”. Current Biology 19 (2): R55–56. (January 2009). doi:10.1016/j.cub.2008.10.043. PMID 19174136.
- ^ “Exocellular electron transfer in anaerobic microbial communities”. Environmental Microbiology 8 (3): 371–82. (March 2006). doi:10.1111/j.1462-2920.2006.00989.x. PMID 16478444.
- ^ “Cross-protection from hydrogen peroxide by helper microbes: the impacts on the cyanobacterium Prochlorococcus and other beneficiaries in marine communities”. Environmental Microbiology Reports 10 (4): 399–411. (August 2018). doi:10.1111/1758-2229.12625. PMID 29411546.
- ^ “Microbial co-operation in the rhizosphere”. Journal of Experimental Botany 56 (417): 1761–78. (July 2005). doi:10.1093/jxb/eri197. PMID 15911555.
- ^ “The gut flora as a forgotten organ”. EMBO Reports 7 (7): 688–93. (July 2006). doi:10.1038/sj.embor.7400731. PMC 1500832. PMID 16819463.
- ^ “A microbial world within us”. Molecular Microbiology 59 (6): 1639–50. (March 2006). doi:10.1111/j.1365-2958.2006.05056.x. PMID 16553872.
- ^ “Lactic acid bacteria and human health”. Annals of Medicine 22 (1): 37–41. (February 1990). doi:10.3109/07853899009147239. PMID 2109988.
- ^ “Probiotics that modify disease risk”. The Journal of Nutrition 135 (5): 1294–98. (May 2005). doi:10.1093/jn/135.5.1294. PMID 15867327.
- ^ “Vitamin B12 sources and microbial interaction”. Experimental Biology and Medicine (Maywood, N.J.) 243 (2): 148–58. (January 2018). doi:10.1177/1535370217746612. PMC 5788147. PMID 29216732.
- ^ Pommerville 2014, pp. 16–21.
- ^ Clark 2010, p. 215.
- ^ Wheelis 2008, p. 44.
- ^ Clark 2010, pp. 30, 195, 233, 236.
- ^ “Helicobacter pylori infection causes both protective and deleterious effects in human health and disease”. Genes and Immunity 22 (4): 218–226. (July 2021). doi:10.1038/s41435-021-00146-4. PMC 8390445. PMID 34244666.
- ^ “40 years of veterinary papers in JAC – what have we learnt?”. The Journal of Antimicrobial Chemotherapy 71 (10): 2681–90. (October 2016). doi:10.1093/jac/dkw363. PMID 27660260.
- ^ “Optimal antimicrobial therapy for sepsis”. American Journal of Health-System Pharmacy 59 Suppl 1: S13–19. (February 2002). doi:10.1093/ajhp/59.suppl_1.S13. PMID 11885408.
- ^ “Chlamydia pneumoniae and atherosclerosis”. Cellular Microbiology 6 (2): 117–27. (February 2004). doi:10.1046/j.1462-5822.2003.00352.x. PMID 14706098.
- ^ “Diseases associated with immunosuppression”. Environmental Health Perspectives 43: 9–19. (February 1982). doi:10.2307/3429162. JSTOR 3429162. PMC 1568899. PMID 7037390.
- ^ “Microbiology of early CF lung disease”. Paediatric Respiratory Reviews 5 Suppl A: S367–69. (2004). doi:10.1016/S1526-0542(04)90065-6. PMID 14980298.
- ^ Pommerville 2014, p. 118.
- ^ a b Pommerville 2014, pp. 646–47.
- ^ Krasner 2014, pp. 165, 369.
- ^ “Ribosomal crystallography: initiation, peptide bond formation, and amino acid polymerization are hampered by antibiotics”. Annual Review of Microbiology 58: 233–51. (2004). doi:10.1146/annurev.micro.58.030603.123822. PMID 15487937.
- ^ “Agricultural use of antibiotics and the evolution and transfer of antibiotic-resistant bacteria”. CMAJ 159 (9): 1129–36. (November 1998). PMC 1229782. PMID 9835883.
- ^ “Disinfection Processes”. Water Environment Research 89 (10): 1206–44. (October 2017). doi:10.2175/106143017X15023776270278. PMID 28954657.
- ^ “Major technological advances and trends in cheese”. Journal of Dairy Science 89 (4): 1174–78. (April 2006). doi:10.3168/jds.S0022-0302(06)72186-5. PMID 16537950.
- ^ Krasner 2014, pp. 25–26.
- ^ “Bioremediation of oil by marine microbial mats”. International Microbiology 5 (4): 189–93. (December 2002). doi:10.1007/s10123-002-0089-5. PMID 12497184.
- ^ “Biofiltration methods for the removal of phenolic residues”. Applied Biochemistry and Biotechnology 129–132 (1–3): 130–52. (2006). doi:10.1385/ABAB:129:1:130. PMID 16915636.
- ^ “Production of fine chemicals using biocatalysis”. Current Opinion in Biotechnology 10 (6): 595–603. (December 1999). doi:10.1016/S0958-1669(99)00040-3. PMID 10600695.
- ^ “Why Bacillus thuringiensis insecticidal toxins are so effective: unique features of their mode of action”. FEMS Microbiology Letters 195 (1): 1–8. (February 2001). doi:10.1111/j.1574-6968.2001.tb10489.x. PMID 11166987.
- ^ “Susceptibility of adult Coccinella septempunctata (Coleoptera: Coccinellidae) to insecticides with different modes of action”. Pest Management Science 62 (7): 651–54. (July 2006). doi:10.1002/ps.1221. PMID 16649191.
- ^ “Bacterial insecticidal toxins”. Critical Reviews in Microbiology 30 (1): 33–54. (2004). doi:10.1080/10408410490270712. PMID 15116762.
- ^ “A functional update of the Escherichia coli K-12 genome”. Genome Biology 2 (9): RESEARCH0035. (2001). doi:10.1186/gb-2001-2-9-research0035. PMC 56896. PMID 11574054.
- ^ “Global organization of metabolic fluxes in the bacterium Escherichia coli”. Nature 427 (6977): 839–43. (February 2004). arXiv:q-bio/0403001. Bibcode: 2004Natur.427..839A. doi:10.1038/nature02289. PMID 14985762.
- ^ “An expanded genome-scale model of Escherichia coli K-12 (iJR904 GSM/GPR)”. Genome Biology 4 (9): R54. (2003). doi:10.1186/gb-2003-4-9-r54. PMC 193654. PMID 12952533.
- ^ “Therapeutic insulins and their large-scale manufacture”. Applied Microbiology and Biotechnology 67 (2): 151–59. (April 2005). doi:10.1007/s00253-004-1809-x. PMID 15580495.
- ^ “Manufacturing of recombinant therapeutic proteins in microbial systems”. Biotechnology Journal 1 (2): 164–86. (February 2006). doi:10.1002/biot.200500051. PMID 16892246.
- ^ “Diversity of phage types among archived cultures of the Demerec collection of Salmonella enterica serovar Typhimurium strains”. Applied and Environmental Microbiology 70 (2): 664–69. (February 2004). Bibcode: 2004ApEnM..70..664R. doi:10.1128/aem.70.2.664-669.2004. PMC 348941. PMID 14766539.
- ^ Wheelis 2008.
- ^ Asimov's Biographical Encyclopedia of Science and Technology (2nd ed.). Garden City, NY: Doubleday and Company. (1982). p. 143]
- ^ Pommerville 2014, p. 7.
- ^ “The Status of the Generic Term Bacterium Ehrenberg 1828”. Journal of Bacteriology 31 (5): 517–18. (May 1936). doi:10.1128/jb.31.5.517-518.1936. PMC 543738. PMID 16559906.
- ^ (ドイツ
語 ) Dritter Beitrag zur Erkenntniss grosser Organisation in der Richtung des kleinsten Raumes. [Third contribution to the knowledge of great organization in the direction of the smallest space]. Berlin: Physikalische Abhandlungen der Koeniglichen Akademie der Wissenschaften. (1835). pp. 143–336 - ^ “Pasteur's Papers on the Germ Theory”. LSU Law Center's Medical and Public Health Law Site, Historic Public Health Articles. 2006
年 12月18日 時点 のオリジナルよりアーカイブ。2006年 11月23日 閲覧 。 - ^ ‘Wash your hands’ was once controversial medical advice, National Geographic.
- ^ “The Nobel Prize in Physiology or Medicine 1905”. Nobelprize.org. 2006
年 12月 10日 時点 のオリジナルよりアーカイブ。2006年 11月22日 閲覧 。 - ^ “HIV causes AIDS: Koch's postulates fulfilled”. Current Opinion in Immunology 8 (5): 613–18. (October 1996). doi:10.1016/S0952-7915(96)80075-6. PMID 8902385.
- ^ Chung. “Ferdinand Julius Cohn (1828–1898): Pioneer of Bacteriology”. Department of Microbiology and Molecular Cell Sciences, The University of Memphis. 2011
年 7月 27日 時点 のオリジナルよりアーカイブ。2019年 3月 23日 閲覧 。 - ^ Drews, Gerhart (1999). “Ferdinand Cohn, a founder of modern microbiology”. ASM News 65 (8): 547–52.
- ^ “Of blood, inflammation and gunshot wounds: the history of the control of sepsis”. The Australian and New Zealand Journal of Surgery 70 (12): 855–61. (December 2000). doi:10.1046/j.1440-1622.2000.01983.x. PMID 11167573.
- ^ “Paul Ehrlich's magic bullets”. The New England Journal of Medicine 350 (11): 1079–80. (March 2004). doi:10.1056/NEJMp048021. PMID 15014180.
- ^ “Biography of Paul Ehrlich”. Nobelprize.org. 2006
年 11月28日 時点 のオリジナルよりアーカイブ。2006年 11月26日 閲覧 。 - ^ “Phylogenetic structure of the prokaryotic domain: the primary kingdoms”. Proceedings of the National Academy of Sciences of the United States of America 74 (11): 5088–90. (November 1977). Bibcode: 1977PNAS...74.5088W. doi:10.1073/pnas.74.11.5088. PMC 432104. PMID 270744.
- ^ Zaremba-Niedzwiedzka, Katarzyna; Caceres, Eva F.; Saw, Jimmy H.; Bäckström, Disa; Juzokaite, Lina; Vancaester, Emmelien; Seitz, Kiley W.; Anantharaman, Karthik et al. (2017-01). “Asgard archaea illuminate the origin of eukaryotic cellular complexity” (
英語 ). Nature 541 (7637): 353–358. doi:10.1038/nature21031. ISSN 0028-0836.